Dissecando a proteínas multi sinalização complexos pela purificação da afinidade de complementação Bimolecular (BiCAP)
Summary
Este manuscrito descreve o protocolo para a purificação de afinidade de complementação Bimolecular (BiCAP). Este novo método facilita o isolamento específico e caracterização proteômica a jusante de algum duas proteínas interagindo, enquanto excluir un-complexado proteínas individuais, bem como os complexos formados com concorrentes parceiros de ligação.
Abstract
A montagem de complexos de proteínas é um mecanismo central subjacente a regulação da célula muitas vias de sinalização. Um grande foco de investigação biomédica é decifrar como estes complexos de proteína dinâmica agem para integrar sinais de várias fontes a fim de direcionar uma resposta biológica específica, e como isto se torna desregulamentado em muitas configurações de doença. Apesar da importância deste mecanismo bioquímico chave, há uma falta de técnicas experimentais que podem facilitar a deconvolução destes complexos de sinalização multi molecular específica e sensível.
Aqui esta lacuna é abordada através da combinação de um ensaio de complementação da proteína com um nanobody de conformação específica, que nós temos denominado Bimolecular complementação de purificação da afinidade (BiCAP). Esta nova técnica facilita o isolamento específico e caracterização proteômica a jusante de qualquer par de interação de proteínas, com exclusão das proteínas individuais das Nações Unidas-complexado e complexos formados com concorrentes parceiros de ligação.
A técnica de BiCAP é adaptável a uma ampla gama de ensaios experimentais a jusante, e o alto grau de especificidade, proporcionada por esta técnica permite mais matizadas investigações sobre a mecânica da montagem complexa de proteína do que é actualmente possível usando técnicas de purificação de afinidade padrão.
Introduction
Montagem complexa de proteínas é um processo chave na manutenção da especificidade spatiotemporal de muitas vias sinalização1,2. Enquanto a natureza crítica desse papel regulador é amplamente reconhecida, há uma falta de técnicas experimentais disponíveis para escrutinar esses complexos. A maioria dos estudos interactomics focar nas interações com proteínas individuais, ou o enriquecimento sequencial de componentes complexos individuais. Aqui apresentamos uma técnica para o isolamento de um dímero de proteínas específicas, enquanto excluir as partes individuais de proteínas componente os complexos formados com vinculação parceiros3a competir. Chamamos esta técnica Bimolecular complementação de purificação da afinidade (BiCAP), que é uma combinação de um previamente existentes ensaio de complementação fragmento da proteína, complementação de fluorescência Bimolecular (BiFC), com o novo uso de um nanobody recombinante de conformação específica para as boas práticas agrícolas e seus derivados (ver tabela de materiais).
Um ensaio de complementação do proteína-fragmento típica baseia-se na expressão de proteínas de “isca” e “presas” fundidos para separar fragmentos de repórteres como luciferase4, β-galactosidase5ou proteína verde fluorescente (GFP)6 ( Figura 1A). Através da interação das proteínas presas e isca, os domínios de repórter de divisão são encorajados a redobrar-se em uma estrutura funcional, permitindo a interação da isca e atacam proteínas para ser visualizado ou quantificada. BiCAP foi adaptado de uma versão dessa técnica que fez uso de fragmentos da variante GFP Venus. Ensaios de complementação de proteínas fluorescentes são um método popular para poder visualizar as interações da proteína-proteína em uma célula viva, mas até agora têm sido limitados a esta um função7. BiCAP representa um avanço significativo a este respeito, como esta técnica não só permite a visualização, mas também o isolamento e interrogatório da interação da proteína-proteína resultante.
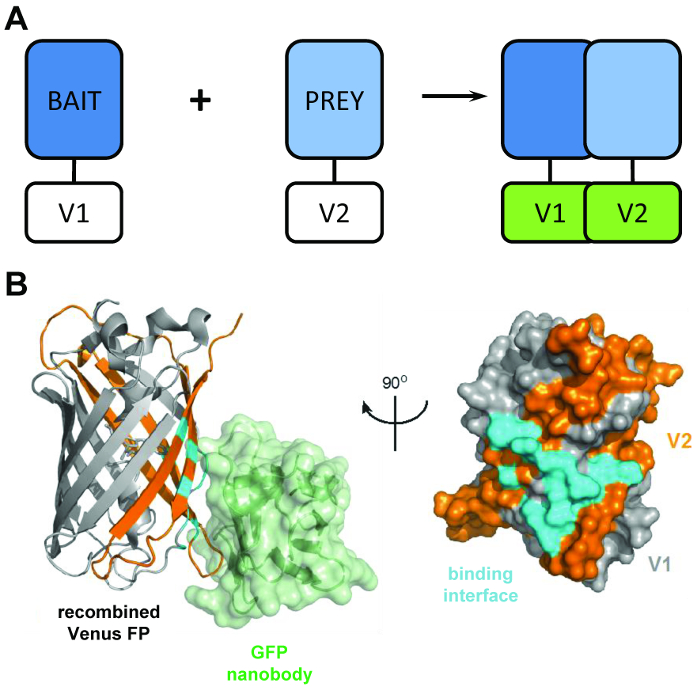
Figura 1: O estrutural principal por trás da técnica BiCAP. (A) um esquema descrevendo o diretor por trás mostrando de complementação de fluorescência bimolecular as proteínas ‘isca’ e ‘presas’ marcado com o N-terminal V1 ou V2 C-terminal fragmentos da proteína completo Venus. (B) análise estrutural da interface de interação (ciano) entre o GFP nanobody (verde) e recombinada Vênus, mostrando a posição do (cinza) V1 e V2 (laranja) fragmentos (PDB 3OGO de adesão). Esta figura é republicado fromCroucher et al3 reproduzido com permissão da AAAS. Clique aqui para ver uma versão maior desta figura.
O BiCAP técnica faz uso de dois fragmentos de não-fluorescente de Vênus (chamado V1 e V2) que associam com baixo grau de afinidade, a menos que uma interação ocorre entre seus parceiros de fusão. Neste caso, os divisão de dois domínios redobrar na estrutura funcional do β-tambor do fluoróforo (figura 1B)6. A inovação chave de BiCAP vem desde a introdução do nanobody recombinante de GFP, que reconhece um epítopo tridimensional sobre o β-barril de GFP (e variantes como Venus), que só está presente no fluoróforo corretamente recombinado e dobrado ( Figura 1B)8. Crucialmente, a GFP nanobody não liga para qualquer um dos fragmentos individuais Venus. Isso facilita o isolamento de dímeros de proteína somente depois que as duas proteínas formaram um complexo de vontade própria, levando a resultados mais representativos do que as adquiridas de métodos que fazem usam de interações quimicamente induzida, forçada9.
BiCAP é uma técnica poderosa que incide especificamente sobre proteínas multi complexos, que potencialmente podem ser combinados com um número de aplicações a jusante para melhorar a granularidade da nossa compreensão do papel que destes complexos jogar na transdução de sinal . Também abrange a característica importante de permitir a visualização de proteína interações in situ. Até à data, foi demonstrado como um método eficaz de analisar a interactome de receptores tirosina quinase (RTK) dímeros3BiCAP, mas a capacidade de adaptação desse método significa que pode ser adotado em quase qualquer contexto de interação de proteínas.
Protocol
Representative Results
Discussion
BiCAP é um poderoso método para isolar dímeros de proteína específica excluindo os componentes individuais e seus concorrentes de parceiros de ligação3. BiCAP baseia-se na adaptação de um ensaio de complementação da fluorescência da proteína chamada BiFC6. Os métodos existentes, incluindo ensaios de ligadura BiFC e proximidade, têm sido amplamente utilizados para visualizar e quantificar as interações da proteína em células vivas7,…
Disclosures
The authors have nothing to disclose.
Acknowledgements
D.R.C é um companheiro de NSW do Instituto de câncer e D.N.S anteriormente foi um companheiro de NSW do Instituto de câncer. Os findings da pesquisa apresentados neste manuscrito foram financiados pelo câncer Instituto NSW (13/FRL/1-02 e 09/CDF/2-39), NHMRC (projeto Grant GNT1052963), Science Foundation Ireland (11/GRIC/B2157), NSW escritório de ciência e pesquisa médica, a família de comentários Comunhão e família Mostyn Foundation. J.F.H. e R.S. foram os destinatários de um prêmio de pós-graduação australiana.
Materials
| LR Clonase II Plus enzyme | Thermo Fisher Scientific | 12538120 | Recombinase enzyme required for Gateway cloning (Step 1) into pDEST BiFC destination vectors |
| Proteinase K, recombinant, PCR grade | Thermo Fisher Scientific | EO0491 | |
| 14 mL round-bottomed polypropylene tube | Corning | 352059 | |
| Ampicillin | Roche Diagnostics Australia | 10835242001 | Stock solution prepared at 100 μg/mL in distilled water. |
| Miniprep kit | Promega Corporation | A1330 | |
| Maxiprep kit | Life Technologies Australia | K2100-07 | |
| DMEM | Gibco | 11995-073 | |
| FBS | Life Technologies Australia | 10099-141 | |
| Penicillin/Streptomycin | Life Technologies Australia | 15070-063 | |
| jetPRIME transfection buffer | Polyplus | 114-15 | |
| jetPRIME transfection reagent | Polyplus | 114-15 | |
| PhosSTOP (Phosphatase inhibitor) | Sigma-Aldrich | 4906837001 | |
| cOmplete, Mini, EDTA-free Protease inhibitor cocktail | Roche | 11873580001 | |
| Cell Scraper | Sarstedt | 83.1832 | |
| GFP-Trap_A | Chromotek Gmbh | gta-100 | GFP nanobody coupled to agarose beads |
| N-terminal GFP monoclonal antibody | Covance | MMS-118P | Will detect the V1 tag within the BiFC vectors |
| C-terminal GFP monoclonal antibody | Roche | 11814460001 | Will detect the V2 tag within the BiFC vectors |
| Sample buffer | Invitrogen | NP0008 | Supplemented with 1 mL β-mercaptoethanol. |
| Sequencing grade modified trypsin | Promega Corporation | V5117h | |
| LoBind microcentrifuge tubes | Point of Care Diagnostics | 0030 108 116 | |
| Iodoacetamide | Sigma-Aldrich | I1149-5G | Prepared at 5 mg/mL in ultrapure water |
| Trifluoroacetic Acid – Sequanal Grade | Thermo Fisher | 10628494 | |
| 3M Empore solid phase extraction C18 disks (octadecyl) – 4.7 cm | Thermo Fisher | 14-386-2 | To prepare stage tips, cut 1 mm disks using an appropriately sized hole punch. Alternatively, pre-prepared stage tips can also be purchased, see below. |
| C18 Stage Tips, 10 µL bed | Thermo Fisher | 87782 | |
| Formic acid OPTIMA for LC/MS grade 50mL | Thermo Fisher | FSBA117-50 | |
| 1.9 μm C18 ReproSil particles | Dr. Maisch GmbH | r119.aq. | Stationary phase particles |
| Acetonitrile OPTIMA LC/MS grade | Thermo Fisher | FSBA955-4 | |
| Easy-nLC HPLC | Thermo Fisher | ||
| LTQ Orbitrap Velos Pro | Thermo Fisher | ||
| Triton X-100 | Sigma-Aldrich | T8787 | Non-ionic detergent (100%) |
| DH5α cells | Thermo Fisher | Heat-shock-competent cells |
References
- Kolch, W., Pitt, A. Functional proteomics to dissect tyrosine kinase signalling pathways in cancer. Nat Rev Cancer. 10 (9), 618-629 (2010).
- Pawson, T., Kofler, M. Kinome signaling through regulated protein-protein interactions in normal and cancer cells. Curr Opin Cell Biol. 21 (2), 147-153 (2009).
- Croucher, D. R., et al. Bimolecular complementation affinity purification (BiCAP) reveals dimer-specific protein interactions for ERBB2 dimers. Sci Signal. 9 (436), ra69 (2016).
- Cassonnet, P., et al. Benchmarking a luciferase complementation assay for detecting protein complexes. Nat Methods. 8 (12), 990-992 (2011).
- Rossi, F., Charlton, C. A., Blau, H. M. Monitoring protein-protein interactions in intact eukaryotic cells by beta-galactosidase complementation. Proc Natl Acad Sci U S A. 94 (16), 8405-8410 (1997).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: scope and mechanism. J Am Chem Soc. 127 (1), 146-157 (2005).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21 (5), 539-545 (2003).
- Kubala, M. H., Kovtun, O., Alexandrov, K., Collins, B. M. Structural and thermodynamic analysis of the GFP:GFP-nanobody complex. Protein Sci. 19 (12), 2389-2401 (2010).
- Fegan, A., White, B., Carlson, J. C., Wagner, C. R. Chemically controlled protein assembly: techniques and applications. Chem Rev. 110 (6), 3315-3336 (2010).
- JoVE Science Education Database. Basic methods in cellular and molecular biology. plasmid purification. J Vis Exp. , (2017).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Shearer, R. F., et al. The E3 ubiquitin ligase UBR5 regulates centriolar satellite stability and primary cilia formation via ubiquitylation of CSPP-L. Mol Biol Cell. , (2018).
- Shannon, P., et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks. Genome Res. 13 (11), 2498-2504 (2003).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, 15690 (2017).

