Dissekere multi protein Signaling komplekser af Bimolecular komplementering affinitet rensning (BiCAP)
Summary
Dette manuskript beskriver protokollen for Bimolecular komplementering affinitet rensning (BiCAP). Denne nye metode letter specifikke isolering og downstream proteom karakterisering af enhver to interagerende proteiner, mens bortset fra FN-kompleksbundet individuelle proteiner samt komplekser dannet med konkurrerende bindende partnere.
Abstract
Samling af proteinkomplekser er en central mekanisme bag regulering af mange celle signalering veje. En større fokus på biomedicinsk forskning decifrere hvordan disse dynamiske proteinkomplekser handle for at integrere signalerne fra flere kilder for at lede en bestemte biologiske respons, og hvordan det bliver liberaliseret i mange indstillinger for sygdom. Trods vigtigheden af denne centrale biokemiske mekanisme er der en mangel på eksperimentelle teknikker, der kan lette de specifikke og følsomme deconvolution af disse multi molekylære signaling komplekser.
Her er denne mangel behandlet gennem kombinationen af en protein komplementering assay med en kropsbygning-specifikke nanobody, som vi har kaldt Bimolecular komplementering affinitet rensning (BiCAP). Denne roman teknik letter specifikke isolering og downstream proteom karakterisering af ethvert par af interagerende proteiner, med udelukkelse af FN-kompleksbundet individuelle proteiner og komplekser dannet med konkurrerende bindende partnere.
BiCAP teknikken er at tilpasse til en bred vifte af nedstrøms eksperimentelt assays, og den høje grad af specificitet ved denne teknik giver mulighed for mere nuancerede undersøgelser af mekanikken i protein kompleks forsamling end det er i øjeblikket muligt ved hjælp af standard affinitet rensning teknikker.
Introduction
Protein komplekse forsamling er en central proces i at bevare for spatiotemporelle specificiteten af mange signalsystemer veje1,2. Mens den kritiske karakter af denne lovgivningsmæssige rolle er bredt anerkendt, er der en mangel på eksperimentelle teknikker til rådighed til at granske disse komplekser. De fleste interactomics studier fokusere på interaktioner med individuelle proteiner eller sekventiel berigelse af komplekse enkeltkomponenter. Her præsenterer vi en teknik til isoleringen af et specifikt protein dimer mens undtagen de enkelte fraspaltning af komponent proteiner samt komplekser dannet med konkurrerende bindende partnere3. Vi har kaldt denne teknik Bimolecular komplementering affinitet rensning (BiCAP), da det er en kombination af et tidligere eksisterende protein fragment komplementering assay, Bimolecular fluorescens komplementering (BiFC), med den roman brug af en kropsbygning-specifikke rekombinante nanobody mod normal god landbrugspraksis og derivater heraf (Se tabel of Materials).
En typisk protein-fragment komplementering analysen bygger på udtryk for “lokkemad” og “bytte” proteiner smeltet for at opdele fragmenter af journalister som luciferase4, β-galactosidase5eller grøn fluorescerende proteiner (NGL)6 ( Figur 1A). Gennem samspillet mellem agn og bytte proteiner, split reporter domæner til at refold til en funktionel struktur, så interaktionen mellem agn og bytte proteiner skal visualiseres eller kvantificeres. BiCAP blev tilpasset fra en version af denne teknik, der gjorde brug af fragmenter af normal god landbrugspraksis variant Venus. Fluorescerende proteiner komplementering assays er en populær metode til at visualisere protein-protein interaktioner i en levende celle, men indtil nu har været begrænset til denne ene funktion7. BiCAP repræsenterer et betydeligt fremskridt i denne henseende, da denne teknik ikke kun giver mulighed for visualisering, men også isolationen og forhør i den resulterende protein-protein interaktion.
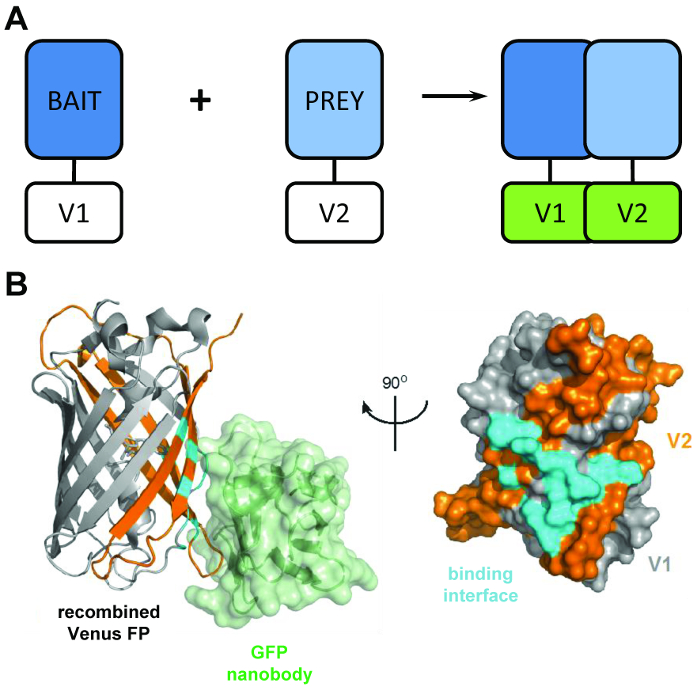
Figur 1: den strukturelle principal bag BiCAP teknik. (A) en skematisk skitserer hovedforpligtet bag bimolecular fluorescens komplementering viser ‘bait’ og ‘bytte’ proteiner markeret med N-terminalen V1 eller C-terminale V2 fragmenter af fuld længde Venus protein. (B) strukturel analyse af samspil interface (cyan) mellem normal god landbrugspraksis nanobody (grøn) og rekombineret Venus, viser placeringen af V1 (grå) og V2 (orange) fragmenter (FBF tiltrædelse 3OGO). Dette tal er genudgives fromCroucher et al.3 Reprinted med tilladelse fra AAAS. Venligst klik her for at se en større version af dette tal.
BiCAP teknik gør brug af to ikke-fluorescerende fragmenter af Venus (opkaldt V1 og V2), som forbinder med en lav grad af affinitet, medmindre en interaktion opstår mellem deres fusion partnere. I dette tilfælde refold to split domænerne i den funktionelle β-tønde struktur af fluorophore (figur 1B)6. Den vigtigste fornyelse af BiCAP kommer fra indførelsen af af rekombinante normal god landbrugspraksis nanobody, som anerkender en tre-dimensionel epitop på β-tønde af normal god landbrugspraksis (og varianter som Venus), som kun er til stede på korrekt rekombineret og foldet fluorophore ( Figur 1B)8. Normal god landbrugspraksis nanobody binder afgørende, ikke til nogen af de individuelle Venus fragmenter. Dette letter isolering af protein dimerer kun efter de to proteiner har dannet et kompleks af deres egen fri vilje, fører til mere repræsentative resultater end dem, erhvervet fra metoder, der gør brug af kemisk induceret, tvungen interaktioner9.
BiCAP er en effektiv teknik, der specifikt fokuserer på multi protein komplekser, som potentielt kan kombineres med en række downstream applikationer til at forbedre granulering af vores forståelse af den rolle, disse komplekser spiller i signaltransduktion . Det omfatter også den vigtige funktion at tillade visualisering af protein interaktioner in situ. Til dato, BiCAP er blevet bevist som en effektiv metode til at analysere interactome receptor tyrosin kinase (RTK) dimerer3, men tilpasningsevne af denne metode betyder, at det kan vedtages i næsten enhver protein interaktion sammenhæng.
Protocol
Representative Results
Discussion
BiCAP er en kraftfuld metode til at isolere bestemte protein dimerer mens undtagen de enkelte komponenter og deres konkurrerende bindende partnere3. BiCAP er baseret på tilpasning af et fluorescens protein komplementering assay kaldet BiFC6. Eksisterende metoder, herunder BiFC og nærhed ligatur assays, har været brugt i udstrakt grad til at visualisere og kvantificere protein interaktioner i levende celler7, men giver ikke et effektivt middel til …
Disclosures
The authors have nothing to disclose.
Acknowledgements
D.R.C er en Cancer Institute NSW fyr og D.N.S var tidligere en Cancer Institute NSW fyr. De forskningsresultater, der er fremlagt i dette manuskript blev finansieret af Cancer Institute NSW (13/FRL/1-02 og 09/CDF/2-39), NHMRC (Project Grant GNT1052963), Science Foundation Irland (11/SIRG/B2157), NSW kontor for videnskab og medicinalforskning, gæst familie Fellowship og Mostyn Family Foundation. J.F.H. og R.S. var modtagerne af en australsk Postgraduate Award.
Materials
| LR Clonase II Plus enzyme | Thermo Fisher Scientific | 12538120 | Recombinase enzyme required for Gateway cloning (Step 1) into pDEST BiFC destination vectors |
| Proteinase K, recombinant, PCR grade | Thermo Fisher Scientific | EO0491 | |
| 14 mL round-bottomed polypropylene tube | Corning | 352059 | |
| Ampicillin | Roche Diagnostics Australia | 10835242001 | Stock solution prepared at 100 μg/mL in distilled water. |
| Miniprep kit | Promega Corporation | A1330 | |
| Maxiprep kit | Life Technologies Australia | K2100-07 | |
| DMEM | Gibco | 11995-073 | |
| FBS | Life Technologies Australia | 10099-141 | |
| Penicillin/Streptomycin | Life Technologies Australia | 15070-063 | |
| jetPRIME transfection buffer | Polyplus | 114-15 | |
| jetPRIME transfection reagent | Polyplus | 114-15 | |
| PhosSTOP (Phosphatase inhibitor) | Sigma-Aldrich | 4906837001 | |
| cOmplete, Mini, EDTA-free Protease inhibitor cocktail | Roche | 11873580001 | |
| Cell Scraper | Sarstedt | 83.1832 | |
| GFP-Trap_A | Chromotek Gmbh | gta-100 | GFP nanobody coupled to agarose beads |
| N-terminal GFP monoclonal antibody | Covance | MMS-118P | Will detect the V1 tag within the BiFC vectors |
| C-terminal GFP monoclonal antibody | Roche | 11814460001 | Will detect the V2 tag within the BiFC vectors |
| Sample buffer | Invitrogen | NP0008 | Supplemented with 1 mL β-mercaptoethanol. |
| Sequencing grade modified trypsin | Promega Corporation | V5117h | |
| LoBind microcentrifuge tubes | Point of Care Diagnostics | 0030 108 116 | |
| Iodoacetamide | Sigma-Aldrich | I1149-5G | Prepared at 5 mg/mL in ultrapure water |
| Trifluoroacetic Acid – Sequanal Grade | Thermo Fisher | 10628494 | |
| 3M Empore solid phase extraction C18 disks (octadecyl) – 4.7 cm | Thermo Fisher | 14-386-2 | To prepare stage tips, cut 1 mm disks using an appropriately sized hole punch. Alternatively, pre-prepared stage tips can also be purchased, see below. |
| C18 Stage Tips, 10 µL bed | Thermo Fisher | 87782 | |
| Formic acid OPTIMA for LC/MS grade 50mL | Thermo Fisher | FSBA117-50 | |
| 1.9 μm C18 ReproSil particles | Dr. Maisch GmbH | r119.aq. | Stationary phase particles |
| Acetonitrile OPTIMA LC/MS grade | Thermo Fisher | FSBA955-4 | |
| Easy-nLC HPLC | Thermo Fisher | ||
| LTQ Orbitrap Velos Pro | Thermo Fisher | ||
| Triton X-100 | Sigma-Aldrich | T8787 | Non-ionic detergent (100%) |
| DH5α cells | Thermo Fisher | Heat-shock-competent cells |
References
- Kolch, W., Pitt, A. Functional proteomics to dissect tyrosine kinase signalling pathways in cancer. Nat Rev Cancer. 10 (9), 618-629 (2010).
- Pawson, T., Kofler, M. Kinome signaling through regulated protein-protein interactions in normal and cancer cells. Curr Opin Cell Biol. 21 (2), 147-153 (2009).
- Croucher, D. R., et al. Bimolecular complementation affinity purification (BiCAP) reveals dimer-specific protein interactions for ERBB2 dimers. Sci Signal. 9 (436), ra69 (2016).
- Cassonnet, P., et al. Benchmarking a luciferase complementation assay for detecting protein complexes. Nat Methods. 8 (12), 990-992 (2011).
- Rossi, F., Charlton, C. A., Blau, H. M. Monitoring protein-protein interactions in intact eukaryotic cells by beta-galactosidase complementation. Proc Natl Acad Sci U S A. 94 (16), 8405-8410 (1997).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: scope and mechanism. J Am Chem Soc. 127 (1), 146-157 (2005).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21 (5), 539-545 (2003).
- Kubala, M. H., Kovtun, O., Alexandrov, K., Collins, B. M. Structural and thermodynamic analysis of the GFP:GFP-nanobody complex. Protein Sci. 19 (12), 2389-2401 (2010).
- Fegan, A., White, B., Carlson, J. C., Wagner, C. R. Chemically controlled protein assembly: techniques and applications. Chem Rev. 110 (6), 3315-3336 (2010).
- JoVE Science Education Database. Basic methods in cellular and molecular biology. plasmid purification. J Vis Exp. , (2017).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Shearer, R. F., et al. The E3 ubiquitin ligase UBR5 regulates centriolar satellite stability and primary cilia formation via ubiquitylation of CSPP-L. Mol Biol Cell. , (2018).
- Shannon, P., et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks. Genome Res. 13 (11), 2498-2504 (2003).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, 15690 (2017).

