Dissekere multi protein signalnettverk komplekser av resultatet av dette Complementation affinitet rensing (BiCAP)
Summary
Dette manuskriptet beskriver protokollen for resultatet av dette Complementation affinitet rensing (BiCAP). Denne romanen metoden forenkler bestemt isolasjon og nedstrøms proteomic karakteristikk av noen to samspill proteiner, mens unntatt FN-kompleksbundet personlige proteiner som komplekser dannet med konkurrerende bindende partnere.
Abstract
Montering av protein komplekser er en sentral mekanisme underliggende regulering av mange celle signalnettverk trasé. Av biomedisinsk forskning er å tyde hvordan disse dynamiske protein komplekser handle for å integrere signaler fra flere kilder for å lede en bestemt biologiske respons, og hvordan dette blir deregulert i mange sykdom-innstillinger. Til tross for betydningen av denne viktige biokjemiske mekanismen er det mangel på eksprementelle teknikker som kan gjøre den spesifikke og følsom deconvolution av disse multi molekylær signalnettverk komplekser.
Her er dette brist adressert gjennom kombinasjonen av et protein complementation analysen med en conformation-spesifikke nanobody, som vi har kalt resultatet av dette Complementation affinitet rensing (BiCAP). Denne teknikken forenkler bestemt isolasjon og nedstrøms proteomic karakteristikk av noen par samspill proteiner, av FNs-kompleksbundet personlige proteiner og komplekser dannet med konkurrerende bindende partnere.
BiCAP teknikken er tilpasses en rekke nedstrøms eksperimentelle analyser, og høye grad av spesifisitet by av denne teknikken tillater mer nyansert undersøkelser i mekanikken i protein kompleks sammenstilling enn er mulig med standard affinitet rensing teknikker.
Introduction
Protein komplekse samlingen er en viktig prosess opprettholde spatiotemporal spesifisitet av mange signal veier1,2. Mens kritiske natur denne regelverk rolle er anerkjent, er det mangel på eksperimentell teknikker tilgjengelig granske disse kompleksene. De fleste interactomics studier fokusere på samspill med personlige proteiner eller sekvensiell anriking av komplekse enkeltkomponenter. Her presenterer vi en teknikk for isolering av et bestemt protein dimer mens unntatt de personlige moieties av komponenten proteiner samt komplekser dannet med konkurrerende bindende partnere3. Vi har kalt denne teknikken resultatet av dette Complementation affinitet rensing (BiCAP), som er en kombinasjon av en tidligere eksisterende protein fragment complementation analysen, resultatet av dette fluorescens Complementation (BiFC), med romanen bruk av en konformasjon-spesifikke rekombinant nanobody mot GFP og dets derivater (se tabell av materialer).
En typisk protein-fragment complementation analysen er avhengig av uttrykket “agn” og “byttedyr” proteiner smeltet sammen for å dele fragmenter av journalister som luciferase4, β-galactosidase5eller grønne fluorescerende protein (GFP)6 ( Figur 1A). Gjennom samhandling av agn og bytte proteiner, er delt reporter domenene oppmuntret å refold til en funksjonell struktur, slik at samspillet av agnet og byttedyr proteiner visualisert eller kvantifisert. BiCAP var tilpasset fra en versjon av denne teknikken som gjorde bruk av fragmenter av GFP varianten Venus. Fluorescerende protein complementation analyser er en populær metode for å visualisere protein-protein interaksjoner i en levende celle, men inntil nå har vært begrenset til denne funksjon7. BiCAP representerer en betydelig fordel i denne forbindelse, som denne teknikken ikke bare tillater for visualisering, men også isolasjon og avhør av resulterende protein-protein samhandlingen.
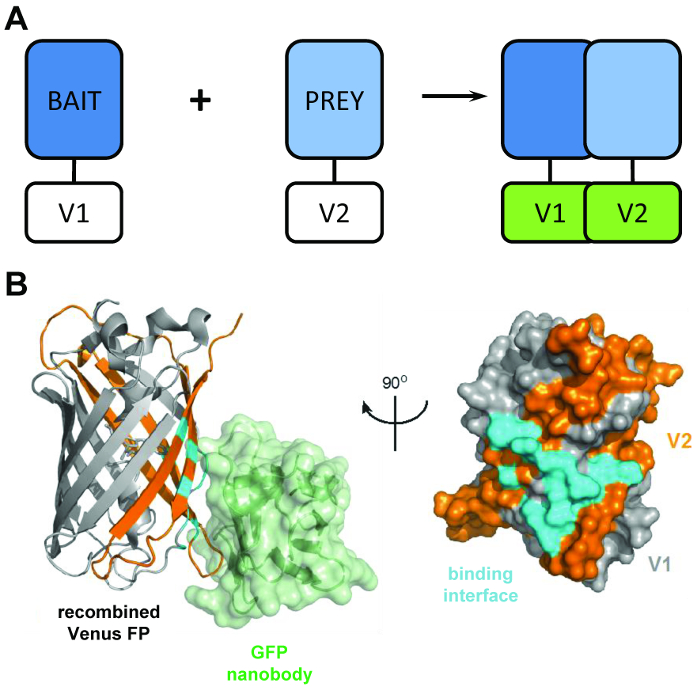
Figur 1: strukturelle rektor bak BiCAP teknikken. (A) en skjematisk beskriver viktigste bak resultatet av dette fluorescens complementation viser ‘agn’ og ‘prey’ proteiner merket med N-terminal V1 eller C-terminalen V2 fragmenter av full lengde Venus protein. (B) strukturell analyse av samhandling grensesnittet (cyan) mellom GFP nanobody (grønn) og saman Venus, viser plasseringen av V1 (grå) og V2 (oransje) fragmenter (PDB tiltredelse 3OGO). Dette tallet er publiseres fromCroucher et al.3 Reprinted med tillatelse fra AAAS. Klikk her for å se en større versjon av dette tallet.
BiCAP teknikken gjør bruk av to ikke-fluorescerende fragmenter av Venus (kalt V1 og V2) som forbinder med en lav grad av affinitet med mindre en samhandling skjer mellom samarbeidspartnerne fusion. I dette tilfellet refold to delt domenene i funksjonelle β fat strukturen av fluorophore (figur 1B)6. Den viktigste nyskapningen av BiCAP kommer fra innføringen av rekombinant GFP nanobody, som gjenkjenner en tredimensjonal epitope på β-fat GFP (og varianter som Venus) som finnes kun på riktig saman og kastet fluorophore ( Figur 1B)8. Avgjørende, det GFP nanobody ikke kan bindes til en av personlige Venus fragmenter. Dette forenkler isolering av protein dimers etter to proteinene har dannet et kompleks av egen vilje, fører til mer representativt resultater enn de fra metoder som gjør bruk av kjemisk indusert, tvungen interaksjoner9.
BiCAP er en kraftfull teknikk som spesielt fokuserer på flere protein kompleksene, som potensielt kan kombineres med en rekke nedstrøms programmer å forbedre detaljnivå av vår forståelse av hvilken rolle disse kompleksene spille i signaltransduksjon . Det omfatter også viktige funksjonen tillater visualisering av protein interaksjoner i situ. Hittil har BiCAP vist som en effektiv metode for å analysere interactome receptor tyrosine kinase (RTK) dimers3, men tilpasningsevne denne metoden betyr at den kan bli vedtatt i nesten alle protein samhandling sammenheng.
Protocol
Representative Results
Discussion
BiCAP er en kraftfull metode for å isolere bestemte protein dimers mens utenom de enkelte komponentene og deres konkurrerende bindende partnere3. BiCAP er basert på tilpasning av en fluorescens protein complementation analysen kalt BiFC6. Eksisterende metoder, inkludert BiFC og nærhet ligation analyser, har vært mye brukt til å visualisere og kvantifisere protein interaksjoner i lever celler7, men gi ikke en effektiv måte å isolere og karakter…
Disclosures
The authors have nothing to disclose.
Acknowledgements
D.R.C er medlem Cancer Institute NSW D.N.S var tidligere medlem Cancer Institute NSW. Undersøkelsen presenteres i dette manuskriptet ble finansiert av Cancer Institute NSW (13/FRL/1-02 og 09/CDF/2-39), NHMRC (Project Grant GNT1052963), Science Foundation Irland (11/SIRG/B2157), NSW Office of Science og medisinsk forskning, gjest familie Fellesskap og Mostyn Family Foundation. J.F.H. og R.S. var mottakere av en australsk Postgraduate Award.
Materials
| LR Clonase II Plus enzyme | Thermo Fisher Scientific | 12538120 | Recombinase enzyme required for Gateway cloning (Step 1) into pDEST BiFC destination vectors |
| Proteinase K, recombinant, PCR grade | Thermo Fisher Scientific | EO0491 | |
| 14 mL round-bottomed polypropylene tube | Corning | 352059 | |
| Ampicillin | Roche Diagnostics Australia | 10835242001 | Stock solution prepared at 100 μg/mL in distilled water. |
| Miniprep kit | Promega Corporation | A1330 | |
| Maxiprep kit | Life Technologies Australia | K2100-07 | |
| DMEM | Gibco | 11995-073 | |
| FBS | Life Technologies Australia | 10099-141 | |
| Penicillin/Streptomycin | Life Technologies Australia | 15070-063 | |
| jetPRIME transfection buffer | Polyplus | 114-15 | |
| jetPRIME transfection reagent | Polyplus | 114-15 | |
| PhosSTOP (Phosphatase inhibitor) | Sigma-Aldrich | 4906837001 | |
| cOmplete, Mini, EDTA-free Protease inhibitor cocktail | Roche | 11873580001 | |
| Cell Scraper | Sarstedt | 83.1832 | |
| GFP-Trap_A | Chromotek Gmbh | gta-100 | GFP nanobody coupled to agarose beads |
| N-terminal GFP monoclonal antibody | Covance | MMS-118P | Will detect the V1 tag within the BiFC vectors |
| C-terminal GFP monoclonal antibody | Roche | 11814460001 | Will detect the V2 tag within the BiFC vectors |
| Sample buffer | Invitrogen | NP0008 | Supplemented with 1 mL β-mercaptoethanol. |
| Sequencing grade modified trypsin | Promega Corporation | V5117h | |
| LoBind microcentrifuge tubes | Point of Care Diagnostics | 0030 108 116 | |
| Iodoacetamide | Sigma-Aldrich | I1149-5G | Prepared at 5 mg/mL in ultrapure water |
| Trifluoroacetic Acid – Sequanal Grade | Thermo Fisher | 10628494 | |
| 3M Empore solid phase extraction C18 disks (octadecyl) – 4.7 cm | Thermo Fisher | 14-386-2 | To prepare stage tips, cut 1 mm disks using an appropriately sized hole punch. Alternatively, pre-prepared stage tips can also be purchased, see below. |
| C18 Stage Tips, 10 µL bed | Thermo Fisher | 87782 | |
| Formic acid OPTIMA for LC/MS grade 50mL | Thermo Fisher | FSBA117-50 | |
| 1.9 μm C18 ReproSil particles | Dr. Maisch GmbH | r119.aq. | Stationary phase particles |
| Acetonitrile OPTIMA LC/MS grade | Thermo Fisher | FSBA955-4 | |
| Easy-nLC HPLC | Thermo Fisher | ||
| LTQ Orbitrap Velos Pro | Thermo Fisher | ||
| Triton X-100 | Sigma-Aldrich | T8787 | Non-ionic detergent (100%) |
| DH5α cells | Thermo Fisher | Heat-shock-competent cells |
References
- Kolch, W., Pitt, A. Functional proteomics to dissect tyrosine kinase signalling pathways in cancer. Nat Rev Cancer. 10 (9), 618-629 (2010).
- Pawson, T., Kofler, M. Kinome signaling through regulated protein-protein interactions in normal and cancer cells. Curr Opin Cell Biol. 21 (2), 147-153 (2009).
- Croucher, D. R., et al. Bimolecular complementation affinity purification (BiCAP) reveals dimer-specific protein interactions for ERBB2 dimers. Sci Signal. 9 (436), ra69 (2016).
- Cassonnet, P., et al. Benchmarking a luciferase complementation assay for detecting protein complexes. Nat Methods. 8 (12), 990-992 (2011).
- Rossi, F., Charlton, C. A., Blau, H. M. Monitoring protein-protein interactions in intact eukaryotic cells by beta-galactosidase complementation. Proc Natl Acad Sci U S A. 94 (16), 8405-8410 (1997).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: scope and mechanism. J Am Chem Soc. 127 (1), 146-157 (2005).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21 (5), 539-545 (2003).
- Kubala, M. H., Kovtun, O., Alexandrov, K., Collins, B. M. Structural and thermodynamic analysis of the GFP:GFP-nanobody complex. Protein Sci. 19 (12), 2389-2401 (2010).
- Fegan, A., White, B., Carlson, J. C., Wagner, C. R. Chemically controlled protein assembly: techniques and applications. Chem Rev. 110 (6), 3315-3336 (2010).
- JoVE Science Education Database. Basic methods in cellular and molecular biology. plasmid purification. J Vis Exp. , (2017).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Shearer, R. F., et al. The E3 ubiquitin ligase UBR5 regulates centriolar satellite stability and primary cilia formation via ubiquitylation of CSPP-L. Mol Biol Cell. , (2018).
- Shannon, P., et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks. Genome Res. 13 (11), 2498-2504 (2003).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, 15690 (2017).

