Identificazione e isolamento di Oligopotent e dei progenitori mieloidi Lineage-commesso dal midollo osseo del topo
Summary
Dimostriamo come identificare e isolare 6 sottoinsiemi dei progenitori mieloidi dal midollo osseo murino utilizzando una combinazione di magnetico e fluorescenza ordinamento (MACS e FACS). Questo protocollo può essere usato per analisi di RNA/proteine, in vivo gli esperimenti di trasferimento adottivo e saggi di cultura in vitro (colture di metilcellulosa o liquido).
Abstract
Progenitori mieloidi che producono neutrofili, monociti e cellule dendritiche (DCs) possono essere identificati e isolati dal midollo osseo di topi per analisi ematologiche ed immunologiche. Ad esempio, studi delle proprietà molecolari e cellulari delle popolazioni di cellule progenitrici mieloidi possono rivelare meccanismi sottostanti la trasformazione leucemica o dimostrare come il sistema immunitario risponde all’esposizione di agente patogeno. In precedenza strategie di citometria a flusso descritto per l’identificazione di cellule progenitrici mieloidi hanno permesso significativi progressi in molti campi, ma le frazioni che identificano sono molto eterogenee. Le strategie più comunemente usate di Gate definiscono frazioni del midollo osseo che sono arricchite per le popolazioni desiderate, ma contengono anche un numero elevato di “contaminare” progenitori. Nostri studi recenti hanno risolto gran parte di questa eterogeneità e il protocollo che presentiamo qui consente l’isolamento 6 sottopopolazioni di oligopotent e dei progenitori mieloidi lignaggio-commessi da 2 precedentemente descritto frazioni del midollo osseo. Il protocollo descrive 3 fasi: 1) l’isolamento del midollo osseo cellule, 2) arricchimento di progenitori ematopoietici di ordinamento magnetico-attivata delle cellule (svuotamento di stirpe di MACS) e 3) identificazione dei sottoinsiemi di cellule progenitrici mieloidi mediante citometria a flusso (tra cui fluorescenza-attivato cella ordinamento, FACS, se lo si desidera). Questo approccio consente la quantificazione di progenitore e isolamento per una varietà di applicazioni in vitro e in vivo e ha già dato romanzo insight sulle vie e sui meccanismi di neutrofili, monociti e differenziazione delle DC.
Introduction
Monociti, neutrofili e cellule dendritiche (DCs) sono cellule mieloidi che derivano da progenitori ematopoietici, principalmente nel midollo osseo, tramite un processo chiamato mielopoiesi. Comune dei progenitori mieloidi (CMPs) hanno il potenziale per produrre cellule mieloidi, come pure i megacariociti ed eritrociti, ma cellule non linfoidi. Progenitori del granulocyte-monocito (GMP), che sono derivati da CMPs, producono di granulociti e monociti, ma hanno perso dei megacariociti e potenziali dell’eritrocito. Monociti e classica e plasmacytoid DCs (cDCs/PDC) sono anche pensato di derivano da progenitori comuni, noti come progenitori del monocito-DC (MDPs), che sono prodotte da CMPs. graduale limitazione del potenziale di lignaggio infine provoca lignaggio-commesso progenitori: progenitori granulociti, monociti progenitori e dei progenitori delle cellule dendritiche (Figura 1).
Weissman e colleghi ha riferito che CMPs si trovano in Lin– c-Kit+ Sca-1– CD34 (LKS–)+ FcγRlo frazione del midollo osseo di topo, mentre le GMP sono contenute nel CD34 LKS– + FcγR Ciao frazione1. Tuttavia, queste frazioni “CMP” e “GMP” sono molto eterogenee. Per esempio, la frazione “GMP” contiene anche dei progenitori del granulocyte lignaggio-commessi e monocito progenitori1,2. MDPs separatamente sono stati segnalati per essere CX3CR1+ Flt3+ CD115+ progenitori che inoltre esprimono CD34 e FcγR3,4. MDPs danno luogo a cDC/pDC-produzione comune DC progenitori (CDP), che sono stati segnalati per esprimere più bassi livelli di c-Kit (CD117) e non sono inclusi nella frazione LKS– 5.
Si è precedentemente ipotizzato che monociti derivano attraverso una singola via (CMP-GMP-MDP-monocito). Coerente con questo modello, monocito-commesso dei progenitori prodotto da PGM (denominato progenitori del monocito, MPs)2 e MDPs (denominato comuni progenitori del monocito, cMoPs)6 sembrano essere le stesse celle sulla base dell’espressione dei marker di superficie condivisa . Tuttavia, abbiamo recentemente dimostrato che monociti sono prodotte in modo indipendente da GMPs e MDPs e sono stati in grado di distinguere tra MPs e cMoPs di cella singola sequenziamento del RNA,7.
Abbiamo recentemente modificato il Weissman “CMP” e “GMP” gating strategia per identificare 6 subfractions del midollo osseo di topo C57BL/6J contenente diversi oligopotent e sottoinsiemi di lignaggio-commesso progenitrici mieloidi. Abbiamo segnalato prima che la macchiatura per Ly6C e CD115 consente l’isolamento di GMPs oligopotent, così come di progenitori del granulocyte (GPs) e di progenitori del monocito (MPs e cMoPs, che siamo attualmente in grado di separare) dalla frazione “GMP”2 (LKS – CD34+ FcγRCiao cancello; Figura 1). Abbiamo successivamente dimostrato che MDPs si trovano principalmente nella frazione “CMP” (CD34 LKS– + FcγRlo cancello), che contiene anche Flt3+ CD115lo e Flt3– sottoinsiemi7 (Figura 1 ). Il CMP-Flt3+ CD115lo frazione rendimenti sia GMPs e MDPs su trasferimento adottivo. Il sottoinsieme di CMP-Flt3– contiene dei progenitori che sembrano essere intermedi tralo cellule CD115 CMP-Flt3+ e GMP. A differenza di MDPs, CMP-Flt3+ CD115lo sia il CMP-Flt3– frazioni anche possiedono megacariocita e potenziali dell’eritrocito.
È importante notare, tuttavia, che non è attualmente chiaro se le frazioni “CMP” contengono progenitori che sono veramente oligopotent (ad es., cellule individuali all’interno del CMP-Flt3+ CD115lo frazione che possiedono dei neutrofili, monociti, DC, megacariocita e potenziali dell’eritrocito), o in alternativa, comprendono una miscela di progenitori con più limitato potenziale di lignaggio. Colonie del saggi (culture metilcellulosa) ha rivelato le cellule con potenziale megacariocita (cellule di GEMM) in “CMP”, dell’eritrocito, monociti e granulociti (neutrofili) CMP-Flt3+ CD115lo e frazioni di CMP-Flt3– 1 ,7, ma non consentono la valutazione del potenziale di DC. Al contrario, Colonia formando le analisi ha dimostrato l’esistenza di oligopotent PGM (progenitori con neutrofili e monociti potenziali) in “GMP” frazione1,2, e questo è supportato da recenti unicellulare di analisi trascrittomica8. Non è attualmente noto, tuttavia, se questi PGM oligopotent produrre anche altri granulociti (eosinofili, basofili e mastociti).
Sulla base di questi studi, ora dimostriamo come 7 marcatori (c-Kit, Sca-1, CD34, FcγR, Flt3, Ly6C e CD115) di superficie può essere utilizzato per identificare e isolare questi 6 sottogruppi della oligopotent e dei progenitori mieloidi lignaggio-commesso. Il protocollo descritto qui può essere applicato per i test di cultura in vitro (colture di metilcellulosa o liquido), esperimenti di trasferimento adottivo in vivo in topi e l’analisi molecolare (sequenziamento di RNA alla rinfusa e a cella singola, Western blotting, ecc).
Il protocollo consiste di 3 fasi: 1) preparazione di una sospensione di singola cellula del midollo osseo cellule, 2) arricchimento di progenitori ematopoietici (ordinamento magnetico-attivata delle cellule) e 3) l’identificazione e l’isolamento se lo si desidera, dei sottoinsiemi di cellule progenitrici di flusso cytometry (utilizzando un analizzatore o un fascicolatore, come appropriato). Il primo passo è l’isolamento delle cellule del midollo osseo dai femori e le tibie di topi eutanasizzati ed è simile ad altri protocolli precedentemente descritti9. Successivamente, il campione si arricchisce per cellule staminali e progenitrici usando un cocktail di anticorpi contro gli indicatori di superficie delle cellule di eritrociti, i neutrofili, monociti, linfociti, ecc., per vuotare le cellule differenziate. Questo non è obbligatorio, ma fortemente consigliato per ottimizzare il rilevamento dei sottoinsiemi progenitore e per ridurre la quantità di anticorpi necessari per l’identificazione di cellule progenitrici e il tempo necessario per citometria a flusso. Il protocollo di deplezione di lignaggio qui sotto descrive l’ordinamento delle cellule Magnetic-Activated (MACS) utilizzando un Kit di svuotamento di Mouse Lineage delle cellule (che contiene anticorpi biotinilati CD5, CD45R (B220), CD11b, Gr-1 (Ly6G/C), 7-4 e Ter-119, plus anti-biotina microsfere) e un separatore magnetico automatizzato. Il passo finale è l’individuazione (e l’ordinamento, se lo si desidera) dei sottoinsiemi progenitore tramite flusso cytometry. Il pannello di anticorpi descritti di seguito (Vedi anche tabella 1) è stato progettato per essere utilizzato in un citometro a flusso (analizzatore o selezionatore) con 4 laser (405 nm, 488 nm, 561 nm, 640 nm).
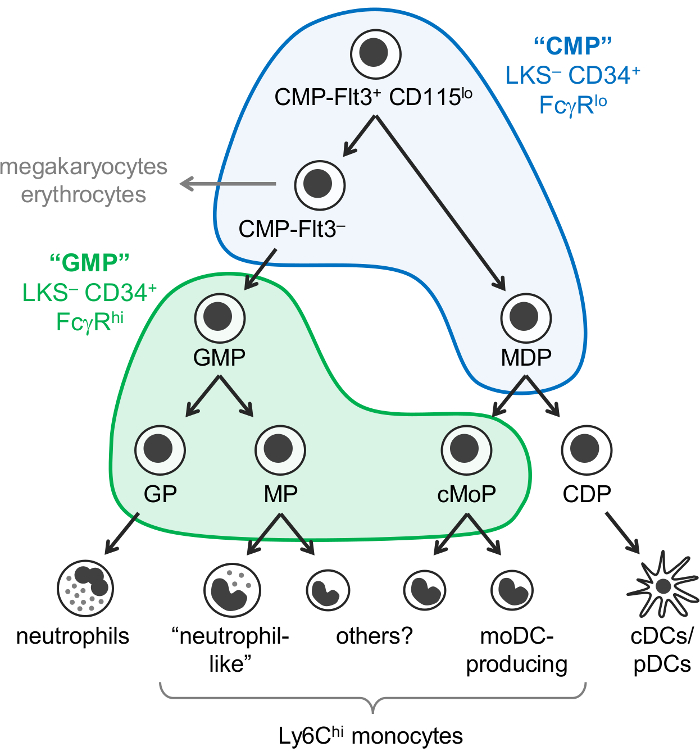
Figura 1: vie di differenziazione e progenitori dei neutrofili, monociti e DC. Il modello recentemente riveduto della mielopoiesi7 è illustrato, con i cancelli di Weissman per “CMP” (blu) e “PGM” (verde)1 sovrapposti. Questa figura è stata modificata da Yáñez et al 20177. Clicca qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
Weissman gating strategia per mouse progenitrici mieloidi identificazione1 è stato il gold standard per gli immunologi ed ematologi per quasi 20 anni, ma ora è evidente che i cancelli “CMP” e “GMP” sono molto eterogenei e più preciso sono necessarie strategie di Gate. Il protocollo che abbiamo descritto qui consenta l’identificazione dell’oligopotent e dei sottoinsiemi di lignaggio-commesso in topi C57BL/6J per più precisa quantificazione dei progenitori mieloidi specifici e mappatura delle vi…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Questo protocollo è stato sviluppato utilizzando fondi dal Consiglio dei governatori rigenerativa medicina Institute al Cedars-Sinai Medical Center (HSG), una carriera in comunione di immunologia dal Associazione americana di immunologi (a AY e HSG) e un Scholar Award da la società americana di Ematologia (di AY). Ringraziamo il nucleo di Cytometry di flusso al Cedars-Sinai Medical Center per assistenza con l’ordinamento FACS.
Materials
| Mouse: Wild-type C57BL/6J (CD45.2) | The Jackson Laboratories | Cat#JAX:000664 | |
| Lineage Cell Depletion Kit, mouse | Miltenyi Biotec | Cat#130-090-858 | |
| Rat anti-mouse CD34 (clone RAM34) FITC | BD Biosciences | Cat#553733 | |
| Rat anti-mouse CD16/CD32 (FcγR; clone 93) APC-Cy7 | BioLegend | Cat#101327 | |
| Rat anti-mouse Ly6A/E (Sca-1; clone 108113) PE-Cy7 | BioLegend | Cat#108114 | |
| Rat anti-mouse CD117 (c-Kit; clone 2B8) Pacific Blue | BioLegend | Cat#105820 | |
| Rat anti-mouse Ly6C (clone HK1.4) PerCP-Cy5.5 | BioLegend | Cat#128012 | |
| Rat anti-mouse CD115 (clone AFS98) PE | BioLegend | Cat#135506 | |
| Rat anti-mouse CD135 (Flt3; clone A2F10.1) APC | BD Biosciences | Cat#560718 | |
| CountBright Absolute Counting Beads | Thermo Fisher Scientific | Cat#C36950 | |
| AutoMACS Separator | Miltenyi Biotec | N/A | Use the "deplete" program |
| BD LSRFortessa | BD Biosciences | N/A | 5 lasers, 15 colors |
| BD FACS Aria III cell sorter | BD Biosciences | N/A | 5 lasers, 13 colors |
| FlowJo | FlowJo, LLC | https://www.flowjo.com | For further analysis of the .fcs files |
References
- Akashi, K., Traver, D., Miyamoto, T., Weissman, I. L. A clonogenic common myeloid progenitor that gives rise to all myeloid lineages. Nature. 404 (6774), 193-197 (2000).
- Yáñez, A., Ng, M. Y., Hassanzadeh-Kiabi, N., Goodridge, H. S. IRF8 acts in lineage-committed rather than oligopotent progenitors to control neutrophil vs monocyte production. Blood. 125 (9), 1452-1459 (2015).
- Auffray, C., et al. CX3CR1+ CD115+ CD135+ common macrophage/DC precursors and the role of CX3CR1 in their response to inflammation. Journal of Experimental Medicine. 206 (3), 595-606 (2009).
- Fogg, D. K., et al. A clonogenic bone marrow progenitor specific for macrophages and dendritic cells. Science. 311 (5757), 83-87 (2006).
- Onai, N., et al. Identification of clonogenic common Flt3+M-CSFR+ plasmacytoid and conventional dendritic cell progenitors in mouse bone marrow. Nature Immunology. 8 (11), 1207-1216 (2007).
- Hettinger, J., et al. Origin of monocytes and macrophages in a committed progenitor. Nature Immunology. 14 (8), 821-830 (2013).
- Yáñez, A., et al. Granulocyte-monocyte progenitors and monocyte-dendritic cell progenitors independently produce functionally distinct monocytes. Immunity. 47 (5), 890-902 (2017).
- Olsson, A., et al. Single-cell analysis of mixed-lineage states leading to a binary cell fate choice. Nature. 537 (7622), 698-702 (2016).
- Amend, S. R., Valkenburg, K. C., Pienta, K. J. Murine hind limb long bone dissection and bone marrow isolation. Journal of Visualized Experiments. (110), (2016).
- Yáñez, A., Goodridge, H. S. Interferon regulatory factor 8 and the regulation of neutrophil, monocyte, and dendritic cell production. Current Opinion in Hematology. 23 (1), 11-17 (2016).
- Manz, M. G., Miyamoto, T., Akashi, K., Weissman, I. L. Prospective isolation of human clonogenic common myeloid progenitors. Proceedings of the National Academy of Sciences of the United States of America. 99 (18), 11872-11877 (2002).

