Genom-Bearbeitung in Säugetieren Zelllinien mit CRISPR-Cas
Summary
CRISPR-Cas ist eine leistungsstarke Technologie, die komplexe Genome von Pflanzen und Tieren zu entwickeln. Hier zeigen wir ein Protokoll, das menschliche Genom mit verschiedenen Cas jedoch effizient zu bearbeiten. Wir markieren wichtige Überlegungen und Design-Parameter zur Optimierung der Bearbeitung Effizienz.
Abstract
Die gruppierten regelmäßig dazwischen kurze palindromische Wiederholungen (CRISPR) System funktioniert natürlich in bakteriellen adaptive Immunität, sondern hat erfolgreich für Genom-Engineering in vielen verschiedenen Lebewesen zweckentfremdet worden. In den meisten Fällen der Wildtyp CRISPR verbundenen 9 (Cas9) oder Cas12a Endonuklease wird verwendet, um bestimmte Stellen im Genom, Spalten, nach denen die DNA-Doppelstrang-Pause über die nicht-homologe Ende verbinden (NHEJ) Weg oder die Reparatur unter der Regie von Homologie (repariert wird HDR) Weg, je nachdem, ob eine Spender Vorlage abwesend oder bzw. zu präsentieren. Bisher wurden die CRISPR-Systeme von verschiedenen Bakterienarten durchführen, Genom-Bearbeitung in Säugetierzellen werden gezeigt. Jedoch trotz der scheinbaren Einfachheit der Technik mehrere Design-Parameter berücksichtigt werden müssen, die oft Benutzer verwirrt darüber, wie am besten ihr Genom Bearbeitung Experimente durchführen lassen. Hier beschreiben wir einen kompletten Workflow von Versuchsanordnung zur Identifikation von Zellklonen, die gewünschte DNA-Modifikationen, mit dem Ziel der Erleichterung der erfolgreichen Ausführung des Genoms Bearbeitung Experimente in Säugetieren Zelllinien zu tragen. Wir markieren wichtige Überlegungen für die Nutzer zu beachten, einschließlich der Auswahl der CRISPR-System, die Abstandhalter Länge und das Design eine einzelsträngige Oligodeoxynucleotide (SsODN)-Spender-Vorlage. Wir uns vorstellen, dass dieses Workflows nützlich für gen Knockout Studien, Modellierung Bemühungen, Krankheit oder die Generation der Reporter Zellinien.
Introduction
Die Fähigkeit, das Genom jeder lebende Organismus Ingenieur hat viele biomedizinische und biotechnologische Anwendungen, wie die Korrektur von krankheitserregenden Mutationen, Bau der korrekte Zellmodellen für Krankheit Studien oder Erzeugung von landwirtschaftlichen Pflanzen mit gewünschten Eigenschaften. Seit der Wende des Jahrhunderts, verschiedene Technologien wurden entwickelt für Genom-Engineering in Säugerzellen, einschließlich Meganucleases1,2,3, Zink-Finger Nukleasen4,5, oder Transkription-Aktivator-ähnliche Effektor Nukleasen (TALENs)6,7,8,9. Allerdings sind dieser früheren Technologien schwierig, Programm oder mühsam zu montieren, dabei behindern ihre Verbreitung in Forschung und Industrie.
In den letzten Jahren die gruppierten regelmäßig dazwischen kurze palindromische Wiederholungen (CRISPR) – CRISPR-assoziierten (Cas) System ist als eine mächtige neue Genom engineering Technologie10,11aufgetaucht. Ursprünglich eine adaptive Immunsystem in Bakterien, es wurde erfolgreich eingesetzt für Genom-Modifikation in Pflanzen und Tieren einschließlich des Menschen. Einer der Hauptgründe warum CRISPR-Cas in so kurzer Zeit so viel Popularität gewonnen hat ist, dass das Element, das die wichtigsten Cas Endonuklease, z. B. Cas9 oder Cas12a (auch bekannt als Cpf1) bringt, an die richtige Stelle im Genom ist einfach ein kurzes Stück der Chimären einzelne Führer RN A (SgRNA) ist das Design einfach und billig zu synthetisieren. Nach an den Zielstandort rekrutiert, die Cas-Enzym fungiert als molekulare Schere und bindet sich die gebundene DNA mit seiner RuvC, Unsichern oder Nuc Domänen12,13,14. Daraus resultierende gestrandeten Doppelunterbrechung (DSB) wird anschließend durch die Zellen entweder über die nicht-homologe Ende verbinden (NHEJ) oder unter der Regie von Homologie Reparatur (HDR) Weg repariert. In Ermangelung einer Reparatur Vorlage der DSB Reparaturen durch die fehleranfällige NHEJ Weg, der pseudo-zufälligen einfügen oder Löschen von Nukleotiden (Indels) an der geschliffenen Stelle hervorrufen kann, verursachen und Frameshift-Mutationen im Protein-kodierenden Gene. Jedoch ist in der Gegenwart eine Spender-Vorlage, die die gewünschten DNS-Änderungen enthält, die DSB durch die High-Fidelity-HDR-Weg repariert. Allgemeine Arten der Spender Vorlagen gehören einsträngige Oligonukleotide (SsODNs) und Plasmide. Das ehemalige wird normalerweise verwendet, wenn die beabsichtigte DNA-Veränderungen (z. B. Änderung von einem einzigen Basenpaar), klein sind, während letztere in der Regel verwendet wird wenn man eine relativ lange Sequenz einfügen will (z. B. die kodierende Sequenz ein grün fluoreszierendes Protein oder GFP) in den Target Locus.
Die Endonuklease-Aktivität des Proteins Cas erfordert die Anwesenheit von ein Protospacer angrenzenden Motiv (PAM) in die Ziel-Seite15. PAM Cas9 ist am 3′ Ende der Protospacer, während die PAM Cas12a (auch Cpf1 genannt) am 5′-Ende statt16. Der Cas-Guide RNA Komplex ist nicht in der Lage, ein DSB einführen, wenn die PAM abwesend17. Daher stellt die PAM eine Einschränkung für die genomische Standorte einer bestimmten Cas-Nuklease in der Lage ist zu Spalten. Zum Glück weisen Cas Nukleasen aus verschiedenen Bakterienarten in der Regel unterschiedliche PAM-Anforderungen. Daher können wir durch die Integration verschiedener CRISPR-Cas-Systeme in unserer engineering-Toolbox, die Palette der Websites erweitern, die in einem Genom ausgerichtet werden kann. Darüber hinaus kann ein natürliches Enzym Cas entwickelt oder weiterentwickelt, um alternative PAM folgen, weitere Ausweitung des Geltungsbereichs der genomischen Ziele für Manipulation18,19,20zugänglich zu erkennen.
Obwohl mehrere CRISPR-Cas-Systeme für technische Zwecke Genom verfügbar sind, haben die meisten Anwender der Technologie vor allem auf die Cas9-Nuklease von Streptococcus Pyogenes (SpCas9) aus mehreren Gründen verlassen. Erstens bedarf es einem relativ einfach NGG PAM, im Gegensatz zu vielen anderen Cas-Proteinen, die nur in Anwesenheit von komplexeren PAMs Spalten können. Zweitens ist es die erste Cas-Endonuklease erfolgreich in menschlichen Zellen21,22,23,24eingesetzt werden. Drittens ist SpCas9 bei weitem das am besten charakterisierten Enzym bis heute. Wenn ein Forscher einen anderen Cas-Nuklease nutzen möchte, wäre er oder sie oft unklar, wie am besten zu entwerfen, das Experiment und wie gut andere Enzyme in verschiedenen biologischen zusammenhängen, die im Vergleich zu SpCas9 führen werden.
Um Klarheit für die relative Performance der verschiedenen CRISPR-Cas-Systeme zu schaffen, haben wir vor kurzem einen systematischen Vergleich der fünf Cas Endonucleases – SpCas9, das Cas9-Enzym von Staphylococcus Aureus (SaCas9), das Cas9 Enzym aus durchgeführt. Neisseria Meningitidis (NmCas9), das Cas12a Enzym aus Acidaminococcus SP. BV3L6 (AsCas12a) und das Cas12a Enzym aus Lachnospiraceae Bakterium ND2006 (LbCas12a)25. Für einen fairen Vergleich haben wir die verschiedenen Cas-Nukleasen mit dem gleichen Satz von Ziel-Sites und anderen experimentellen Bedingungen ausgewertet. Die Studie auch abgegrenzt-Design-Parameter für jedes CRISPR-Cas-System, das als nützliches Nachschlagewerk für Anwender der Technologie dienen würde. Hier, besser es Wissenschaftlern ermöglichen, nutzen Sie das CRISPR-Cas-System, bieten wir eine Schritt für Schritt Protokoll für optimale Genom Engineering mit verschiedenen Cas9 und Cas12a Enzyme (siehe Abbildung 1). Das Protokoll enthält nicht nur experimentelle Details aber auch wichtige Überlegungen, die Wahrscheinlichkeit eines erfolgreichen Genom engineering Ergebnisses in Säugetierzellen zu maximieren.
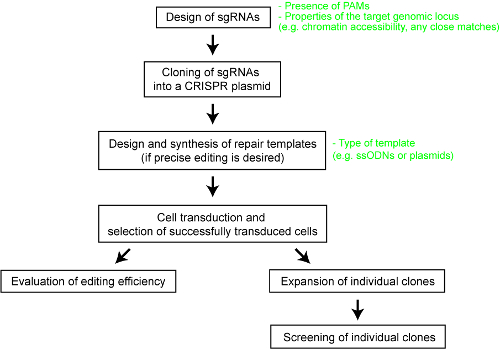
Abbildung 1 : Ein Überblick über den Workflow Genom generieren bearbeitet humanen Zelllinien. Bitte klicken Sie hier für eine größere Version dieser Figur.
Protocol
Representative Results
Discussion
Das CRISPR-Cas-System ist eine leistungsstarke, revolutionäre Technologie, die Genome und Transkriptom von Pflanzen und Tieren zu entwickeln. CRISPR-Cas-Systeme enthalten, die möglicherweise für Genom und technische Zwecke44Transkriptom angepasst wurden zahlreiche Bakterienarten gefunden. Obwohl die Cas9 Endonuklease von Streptococcus Pyogenes (SpCas9) war das erste Enzym erfolgreich in menschlichen Zellen21,22,<sup …
Disclosures
The authors have nothing to disclose.
Acknowledgements
M.HS wird unterstützt von einer Agentur für Wissenschaft, Technologie und Forschung gemeinsame Ratsbüro Grant (1431AFG103), ein National Medical Research Council zu gewähren (OFIRG/0017/2016), National Research Foundation gewährt (NRF2013-THE001-046 und NRF2013-THE001-093), eine Ministerium der Ausbildung Stufe 1 Grant (RG50/17 (S)), ein Start-up gewähren von Nanyang Technological University, und die Mittel für die International genetisch Engineering Machine (iGEM) Wettbewerb der Nanyang Technological University.
Materials
| T4 Polynucleotide Kinase (PNK) | NEB | M0201 | |
| Shrimp Alkaline Phosphatase (rSAP) | NEB | M0371 | |
| Tris-Acetate-EDTA (TAE) Buffer, 50X | 1st Base | BUF-3000-50X4L | Dilute to 1X before use. The 1X solution contains 40 mM Tris, 20 mM acetic acid, and 1 mM EDTA. |
| Tris-EDTA (TE) Buffer, 10X | 1st Base | BUF-3020-10X4L | Dilute to 1X before use. The 1X solution contains 10 mM Tris (pH 8.0) and 1 mM EDTA. |
| BbsI | NEB | R0539 | |
| BsmBI | NEB | R0580 | |
| T4 DNA Ligase | NEB | M0202 | 400,000 units/ml |
| Quick Ligation Kit | NEB | M2200 | An alternative to T4 DNA Ligase. |
| Rapid DNA Ligation Kit | Thermo Scientific | K1423 | An alternative to T4 DNA Ligase. |
| Zero Blunt TOPO PCR Cloning Kit | Thermo Scientific | 451245 | The salt solution comes with the TOPO vector in the kit. |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Kit for Gibson assembly. |
| One Shot Stbl3 Chemically Competent E.Coli | Thermo Scientific | C737303 | |
| LB Broth (Lennox), powder | Sigma Aldrich | L3022 | Reconstitute in ddH20, and autoclave before use |
| LB Broth with Agar (Lennox), powder | Sigma Aldrich | L2897 | Reconstitute in ddH20, and autoclave before use |
| SOC media | – | – | 2.5 mM KCl, 10 mM MgCl2, 20 mM glucose in 1 L of LB Broth |
| Ampicillin (Sodium), USP Grade | Gold Biotechnology | A-301 | |
| REDiant 2X PCR Mastermix | 1st Base | BIO-5185 | |
| Agarose | 1st Base | BIO-1000 | |
| T7 Endonuclease I | NEB | M0302 | |
| Plasmid DNA Extraction Miniprep Kit | Favorgen | FAPDE 300 | |
| Dulbecco's Modified Eagle Medium (DMEM), High Glucose | Hyclone | SH30081.01 | 4.5 g/L Glucose, no L-glutamine, HEPES and Sodium Pyruvate |
| L-Glutamine, 200mM | Gibco | 25030 | |
| Penicillin-Streptomycin, 10, 000U/mL | Gibco | 15140 | |
| 0.25% Trypsin-EDTA, 1X | Gibco | 25200 | |
| Fetal Bovine Serum | Hyclone | SV30160 | FBS is heat inactivated before use at 56 oC for 30 min |
| Phosphate Buffered Saline, 1X | Gibco | 20012 | |
| jetPRIME transfection reagent | Polyplus Transfection | 114-75 | |
| QuickExtract DNA Extraction Solution, 1.0 | Epicentre | LUCG-QE09050 | |
| ISOLATE II Genomic DNA Kit | Bioline | BIO-52067 | An alternative to QuickExtract |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491 | |
| Deoxynucleotide (dNTP) Solution Mix | NEB | N0447 | |
| 6X DNA Loading Dye | Thermo Scientific | R0611 | 10 mM Tris-HCl (pH 7.6) 0.03% bromophenol blue, 0.03% xylene cyanol FF, 60% glycerol, 60 mM EDTA |
| Protease Inhibitor Cocktail, Set3 | Merck | 539134 | |
| Nitrocellulose membrane, 0.2µm | Bio-Rad | 1620112 | |
| Tris-glycine-SDS buffer, 10X | Bio-Rad | 1610772 | Dilute to 1X before use. The 1x solution contains 25 mM Tris, 192 mM glycine, and 0.1% SDS. |
| Tris-glycine buffer, 10X | 1st base | BUF-2020 | Dilute to 1X before use. The 1x solution contains 25 mM Tris and 192 mM glycine. |
| Ponceau S solution | Sigma Aldrich | P7170 | |
| TBS, 20X | 1st base | BUF-3030 | Dilute to 1X before use. The 1x solution contains 25 mM Tris-HCl (pH 7.5) and 150 mM NaCl. |
| Tween 20 | Sigma Aldrich | P9416 | |
| Skim Milk for immunoassay | Nacalai Tesque | 31149-75 | |
| WesternBright Sirius-femtogram HRP | Advansta | K12043 | |
| Antibody for β-actin (C4) | Santa Cruz Biotechnology | sc-47778 | Lot number: C0916 |
| MiSeq system | Illumina | SY-410-1003 | |
| NanoDrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit fluorometer | Thermo Scientific | Q33226 | |
| EVOS FL Cell Imaging System | Thermo Scientific | AMF4300 | |
| CRISPR plasmid: pSpCas9(BB)-2A-GFP (PX458) | Addgene | 48138 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: pX601-AAV-CMV::NLS-SaCas9-NLS-3xHA-bGHpA | Addgene | 61591 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: xCas9 3.7 | Addgene | 108379 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: pX330-U6-Chimeric_BB-CBh-hSpCas9 | Addgene | 42230 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: hCas9 | Addgene | 41815 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: eSpCas9(1.1) | Addgene | 71814 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: VP12 (SpCas9-HF1) | Addgene | 72247 | Dual vector system: The gRNA is expressed from a different plasmid. |
References
- Epinat, J. C., et al. A novel engineered meganuclease induces homologous recombination in yeast and mammalian cells. Nucleic Acids Research. 31 (11), 2952-2962 (2003).
- Arnould, S., et al. Engineered I-CreI derivatives cleaving sequences from the human XPC gene can induce highly efficient gene correction in mammalian cells. Journal of Molecular Biology. 371 (1), 49-65 (2007).
- Chapdelaine, P., Pichavant, C., Rousseau, J., Paques, F., Tremblay, J. P. Meganucleases can restore the reading frame of a mutated dystrophin. Gene Therapy. 17 (7), 846-858 (2010).
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nature Biotechnology. 29 (2), 143-148 (2011).
- Zhang, F., et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription. Nature Biotechnology. 29 (2), 149-153 (2011).
- Boch, J., et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science. 326 (5959), 1509-1512 (2009).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Nishimasu, H., et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell. 156 (5), 935-949 (2014).
- Yamano, T., et al. Crystal Structure of Cpf1 in Complex with Guide RNA and Target DNA. Cell. 165 (4), 949-962 (2016).
- Swarts, D. C., Mosterd, C., van Passel, M. W., Brouns, S. J. CRISPR interference directs strand specific spacer acquisition. PLoS One. 7 (4), e35888 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Kleinstiver, B. P., et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition. Nature Biotechnology. 33 (12), 1293-1298 (2015).
- Kleinstiver, B. P., et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature. 523 (7561), 481-485 (2015).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Jinek, M., et al. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Cho, S. W., Kim, S., Kim, J. M., Kim, J. S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nature Biotechnology. 31 (3), 230-232 (2013).
- Wang, Y., et al. Systematic evaluation of CRISPR-Cas systems reveals design principles for genome editing in human cells. Genome Biology. 19 (1), 62 (2018).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences U S A. 110 (39), 15644-15649 (2013).
- Kim, E., et al. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni. Nature Communications. 8, 14500 (2017).
- Edraki, A., et al. A Compact, High-Accuracy Cas9 with a Dinucleotide PAM for In Vivo Genome Editing. Molecular Cell. , (2018).
- Chatterjee, P., Jakimo, N., Jacobson, J. M. Minimal PAM specificity of a highly similar SpCas9 ortholog. Science Advances. 4 (10), (2018).
- Muller, M., et al. Streptococcus thermophilus CRISPR-Cas9 Systems Enable Specific Editing of the Human Genome. Mol Therapy. 24 (3), 636-644 (2016).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Boratyn, G. M., et al. BLAST: a more efficient report with usability improvements. Nucleic Acids Research. 41 (Web Server issue), W29-W33 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Montague, T. G., Cruz, J. M., Gagnon, J. A., Church, G. M., Valen, E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research. 42 (Web Server issue), W401-W407 (2014).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 122-123 (2014).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30 (10), 1473-1475 (2014).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Richardson, C. D., Ray, G. J., Bray, N. L., Corn, J. E. Non-homologous DNA increases gene disruption efficiency by altering DNA repair outcomes. Nature Communications. 7, 12463 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biology. 18 (1), 35 (2017).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shmakov, S., et al. Diversity and evolution of class 2 CRISPR-Cas systems. Nature Reviews Microbiology. 15 (3), 169-182 (2017).
- Moreno-Mateos, M. A., et al. CRISPR-Cpf1 mediates efficient homology-directed repair and temperature-controlled genome editing. Nature Communications. 8 (1), 2024 (2017).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Yang, L., et al. Optimization of scarless human stem cell genome editing. Nucleic Acids Research. 41 (19), 9049-9061 (2013).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).

