Genomet redigering i pattedyr linjer med CRISPR-Cas
Summary
CRISPR-Cas er en kraftig teknologi til ingeniør komplekse genomer av planter og dyr. Her, detalj vi en protokoll effektivt redigere det menneskelige genomet bruker forskjellige Cas-endonucleases. Vi markere viktige hensyn og design parametere for å optimalisere redigering effektivitet.
Abstract
Gruppert regelmessig interspaced kort palindromic gjentar (CRISPR) systemet fungerer naturlig bakteriell adaptiv immunitet, men har blitt vellykket repurposed genomet Engineering i mange forskjellige levende organismer. Oftest wildtype CRISPR tilknyttet 9 (Cas9) eller Cas12a endonuclease brukes til å holde seg bestemte nettsteder i genomet, hvoretter DNA double-strandet pause repareres via ikke-homologe slutten å bli (NHEJ) veien eller homologi-rettet reparasjon ( HDR) sti avhengig av om en giver mal er fraværende eller presentere henholdsvis. Hittil har CRISPR systemer fra ulike bakterie-art vist å kunne utføre genomet redigering i pattedyrceller. Men til tross for den tilsynelatende enkelheten av teknologi må flere design parametere vurderes, som ofte lar brukerne rådvill om hvordan best for å utføre sine genom redigering eksperimenter. Her beskriver vi en komplett arbeidsflyt fra experimental design til identifikasjon av cellen kloner som bærer DNA endringer, med mål om å tilrettelegge vellykket gjennomføring av genomet redigering eksperimenter i pattedyr linjer. Vi markere nøkkelfaktorer for brukere å merke, inkludert valg av CRISPR system, hvor mellomrom og utformingen av en enkelt-strandet oligodeoxynucleotide (ssODN) giver mal. Vi forventer at denne arbeidsflyten vil være nyttig for genet knockout studier, sykdom modellering innsats, eller generering av reporteren cellelinjer.
Introduction
Muligheten til å ingeniør genomet av enhver levende organisme har mange biomedisinsk og bioteknologisk programmer, for eksempel korrigering av sykdomsfremkallende mutasjoner, bygging av nøyaktige mobilnettet modeller for sykdom studier eller generering av landbruket vekster med ønskelig egenskaper. Siden begynnelsen av århundret, ulike teknologier er utviklet for genomet engineering i pattedyrceller, inkludert meganucleases1,2,3, sink finger nucleases4,5, eller transkripsjon aktivator som effektor nucleases (TALENs)6,7,8,9. Men er disse tidligere teknologiene vanskelig å programmet eller kjedelig å montere, og dermed hindrer deres utbredt adopsjon i forskning og industri.
De siste årene, de klynget interspaced regelmessig kort palindromic gjentar (CRISPR) – CRISPR-assosiert (Cas) systemet har dukket opp som en kraftig ny genomet engineering teknologi10,11. Opprinnelig en adaptive immunsystemet i bakterier, det har blitt deployert for genomet endring i planter og dyr, inkludert mennesker. Hovedårsak hvorfor CRISPR-Cas har fått så mye popularitet i så kort tid er at elementet som bringer den viktigste Cas-endonuclease, som Cas9 eller Cas12a (også kjent som Cpf1), til riktig sted i genomet er bare et kort stykke chimeric enkelt støttelinje RN En (sgRNA), som er enkelt å utforme og billig å syntetisere. Etter blir rekruttert til målområdet, Cas enzymet fungerer som et par molekylær saks og innstiftet bundet DNA med sin RuvC, HNH eller Nuc domener12,13,14. Resulterende dobbel strandet pause (DSB) repareres senere av cellene via enten ikke-homologe slutten med (NHEJ) eller homologi-rettet reparasjon (HDR) veien. I fravær av en reparasjon mal repareres DSB av feilutsatte NHEJ veien, som kan gi opphav til pseudo-tilfeldige innsetting eller sletting av nukleotider (indeler) på webområdet kutt, potensielt forårsake frameshift mutasjoner i protein-koding gener. Men i nærvær av en donor mal som inneholder endringene i DNA, er DSB reparert av Hi-Fi HDR veien. Vanlige typer donor maler inkluderer enkelt-strandet oligonucleotides (ssODNs) og plasmider. Tidligere brukes vanligvis hvis tiltenkte DNA endringene er små (for eksempel endring av en enkelt base-par), mens sistnevnte brukes vanligvis hvis man ønsker å sette inn en relativt lang sekvens (for eksempel koding sekvensen av en grønn fluorescerende protein eller GFP) i målet locus.
Endonuclease aktiviteten til Cas protein krever tilstedeværelse av en protospacer tilstøtende motiv (PAM) mål område15. PAM Cas9 er på 3 slutten av protospacer, PAM Cas12a (også kalt Cpf1) er på 5′ slutten i stedet16. Cas-guiden RNA komplekse er ikke innføre en DSB hvis PAM er fraværende17. Derfor plasserer PAM en begrensning på genomisk stedene der en bestemt Cas-nuclease er i stand til å holde seg. Heldigvis viser Cas nucleases fra ulike bakterie-art vanligvis ulike PAM krav. Ved å integrere ulike CRISPR-Cas systemer i våre tekniske verktøykassen, kan vi derfor utvide omfanget av nettsteder som kan målrettes i et genom. Videre kan et naturlig Cas enzym være konstruert eller utviklet for å gjenkjenne alternativ PAM sekvenser, ytterligere utvide omfanget av genomisk mål tilgjengelig for manipulasjon18,19,20.
Selv om flere CRISPR-Cas-systemer finnes genomet engineering formål, har de fleste brukere av teknologi stolt på Cas9 nuclease fra Streptococcus pyogenes (SpCas9) av flere grunner. Først krever det en relativt enkelt NGG PAM, i motsetning til mange andre sertifiseringsinstanser proteiner som kan bare holde seg i nærvær av mer komplekse PAMs. Det andre er den første Cas endonuclease å bli distribuert i menneskeceller21,22,23,24. Tredje er SpCas9 langt den beste preget enzymet hittil. Hvis en forsker ønsker å bruke en annen Cas nuclease, vil han eller hun ofte være uklart om hvordan best å utforme eksperimentet og hvor godt andre enzymer vil opptre i ulike biologiske sammenhenger i forhold til SpCas9.
For å utdype den relative ytelsen til forskjellige CRISPR-Cas-systemer, har vi nylig utførte en systematisk Sammenlikning av fem Cas endonucleases-SpCas9, Cas9 enzymet fra Staphylococcus aureus (SaCas9), Cas9 enzymet fra Neisseria meningitidis (NmCas9), Cas12a enzymet fra Acidaminococcus sp. BV3L6 (AsCas12a) og Cas12a enzymet fra Lachnospiraceae bakterie ND2006 (LbCas12a)25. For en rettferdig sammenligning vurdert vi de ulike Cas nucleases bruker samme sett med målområder og andre eksperimentelle forhold. Studien også avgrenset design parameterne for hver CRISPR-Cas-system, som ville tjene som en nyttig referanse for brukere av teknologi. Her, bedre aktiverer forskere å bruke CRISPR-Cas system, gir vi en trinnvis protokoll for optimal genomet engineering med forskjellige Cas9 og Cas12a enzymer (se figur 1). Protokollen inkluderer ikke bare eksperimentelle detaljer men også viktige Utformingshensyn å maksimere sannsynligheten for et vellykket genomet engineering resultat i pattedyrceller.
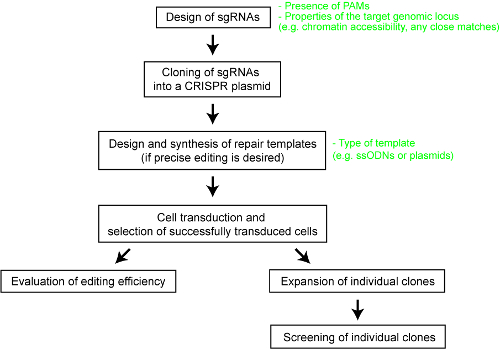
Figur 1 : En oversikt over arbeidsflyten skal generere genomet endret humane cellelinjer. Klikk her for å se en større versjon av dette tallet.
Protocol
Representative Results
Discussion
CRISPR-Cas systemet er en kraftig og revolusjonerende teknologi til ingeniør genomes og transcriptomes av planter og dyr. Mange bakteriell arter er funnet for å inneholde CRISPR-Cas-systemer, som kan være tilpasset genom og transcriptome engineering formål44. Selv om Cas9 endonuclease fra Streptococcus pyogenes (SpCas9) var den første enzymet distribueres vellykket i menneskeceller21,22,23<s…
Disclosures
The authors have nothing to disclose.
Acknowledgements
M.H.T. støttes av et byrå for vitenskap teknologi og Research felles rådet Office grant (1431AFG103), gir en National Medical Research Council (OFIRG/0017/2016), National Research Foundation gir (NRF2013-THE001-046 og NRF2013-THE001-093), en Departementet for utdanning Tier 1 grant (RG50-17 (S)), en oppstart gi fra Nanyang Technological University, og midler til internasjonale genetisk Engineering maskin (iGEM) konkurransen fra Nanyang Technological University.
Materials
| T4 Polynucleotide Kinase (PNK) | NEB | M0201 | |
| Shrimp Alkaline Phosphatase (rSAP) | NEB | M0371 | |
| Tris-Acetate-EDTA (TAE) Buffer, 50X | 1st Base | BUF-3000-50X4L | Dilute to 1X before use. The 1X solution contains 40 mM Tris, 20 mM acetic acid, and 1 mM EDTA. |
| Tris-EDTA (TE) Buffer, 10X | 1st Base | BUF-3020-10X4L | Dilute to 1X before use. The 1X solution contains 10 mM Tris (pH 8.0) and 1 mM EDTA. |
| BbsI | NEB | R0539 | |
| BsmBI | NEB | R0580 | |
| T4 DNA Ligase | NEB | M0202 | 400,000 units/ml |
| Quick Ligation Kit | NEB | M2200 | An alternative to T4 DNA Ligase. |
| Rapid DNA Ligation Kit | Thermo Scientific | K1423 | An alternative to T4 DNA Ligase. |
| Zero Blunt TOPO PCR Cloning Kit | Thermo Scientific | 451245 | The salt solution comes with the TOPO vector in the kit. |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Kit for Gibson assembly. |
| One Shot Stbl3 Chemically Competent E.Coli | Thermo Scientific | C737303 | |
| LB Broth (Lennox), powder | Sigma Aldrich | L3022 | Reconstitute in ddH20, and autoclave before use |
| LB Broth with Agar (Lennox), powder | Sigma Aldrich | L2897 | Reconstitute in ddH20, and autoclave before use |
| SOC media | – | – | 2.5 mM KCl, 10 mM MgCl2, 20 mM glucose in 1 L of LB Broth |
| Ampicillin (Sodium), USP Grade | Gold Biotechnology | A-301 | |
| REDiant 2X PCR Mastermix | 1st Base | BIO-5185 | |
| Agarose | 1st Base | BIO-1000 | |
| T7 Endonuclease I | NEB | M0302 | |
| Plasmid DNA Extraction Miniprep Kit | Favorgen | FAPDE 300 | |
| Dulbecco's Modified Eagle Medium (DMEM), High Glucose | Hyclone | SH30081.01 | 4.5 g/L Glucose, no L-glutamine, HEPES and Sodium Pyruvate |
| L-Glutamine, 200mM | Gibco | 25030 | |
| Penicillin-Streptomycin, 10, 000U/mL | Gibco | 15140 | |
| 0.25% Trypsin-EDTA, 1X | Gibco | 25200 | |
| Fetal Bovine Serum | Hyclone | SV30160 | FBS is heat inactivated before use at 56 oC for 30 min |
| Phosphate Buffered Saline, 1X | Gibco | 20012 | |
| jetPRIME transfection reagent | Polyplus Transfection | 114-75 | |
| QuickExtract DNA Extraction Solution, 1.0 | Epicentre | LUCG-QE09050 | |
| ISOLATE II Genomic DNA Kit | Bioline | BIO-52067 | An alternative to QuickExtract |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491 | |
| Deoxynucleotide (dNTP) Solution Mix | NEB | N0447 | |
| 6X DNA Loading Dye | Thermo Scientific | R0611 | 10 mM Tris-HCl (pH 7.6) 0.03% bromophenol blue, 0.03% xylene cyanol FF, 60% glycerol, 60 mM EDTA |
| Protease Inhibitor Cocktail, Set3 | Merck | 539134 | |
| Nitrocellulose membrane, 0.2µm | Bio-Rad | 1620112 | |
| Tris-glycine-SDS buffer, 10X | Bio-Rad | 1610772 | Dilute to 1X before use. The 1x solution contains 25 mM Tris, 192 mM glycine, and 0.1% SDS. |
| Tris-glycine buffer, 10X | 1st base | BUF-2020 | Dilute to 1X before use. The 1x solution contains 25 mM Tris and 192 mM glycine. |
| Ponceau S solution | Sigma Aldrich | P7170 | |
| TBS, 20X | 1st base | BUF-3030 | Dilute to 1X before use. The 1x solution contains 25 mM Tris-HCl (pH 7.5) and 150 mM NaCl. |
| Tween 20 | Sigma Aldrich | P9416 | |
| Skim Milk for immunoassay | Nacalai Tesque | 31149-75 | |
| WesternBright Sirius-femtogram HRP | Advansta | K12043 | |
| Antibody for β-actin (C4) | Santa Cruz Biotechnology | sc-47778 | Lot number: C0916 |
| MiSeq system | Illumina | SY-410-1003 | |
| NanoDrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit fluorometer | Thermo Scientific | Q33226 | |
| EVOS FL Cell Imaging System | Thermo Scientific | AMF4300 | |
| CRISPR plasmid: pSpCas9(BB)-2A-GFP (PX458) | Addgene | 48138 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: pX601-AAV-CMV::NLS-SaCas9-NLS-3xHA-bGHpA | Addgene | 61591 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: xCas9 3.7 | Addgene | 108379 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: pX330-U6-Chimeric_BB-CBh-hSpCas9 | Addgene | 42230 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: hCas9 | Addgene | 41815 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: eSpCas9(1.1) | Addgene | 71814 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: VP12 (SpCas9-HF1) | Addgene | 72247 | Dual vector system: The gRNA is expressed from a different plasmid. |
References
- Epinat, J. C., et al. A novel engineered meganuclease induces homologous recombination in yeast and mammalian cells. Nucleic Acids Research. 31 (11), 2952-2962 (2003).
- Arnould, S., et al. Engineered I-CreI derivatives cleaving sequences from the human XPC gene can induce highly efficient gene correction in mammalian cells. Journal of Molecular Biology. 371 (1), 49-65 (2007).
- Chapdelaine, P., Pichavant, C., Rousseau, J., Paques, F., Tremblay, J. P. Meganucleases can restore the reading frame of a mutated dystrophin. Gene Therapy. 17 (7), 846-858 (2010).
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nature Biotechnology. 29 (2), 143-148 (2011).
- Zhang, F., et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription. Nature Biotechnology. 29 (2), 149-153 (2011).
- Boch, J., et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science. 326 (5959), 1509-1512 (2009).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Nishimasu, H., et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell. 156 (5), 935-949 (2014).
- Yamano, T., et al. Crystal Structure of Cpf1 in Complex with Guide RNA and Target DNA. Cell. 165 (4), 949-962 (2016).
- Swarts, D. C., Mosterd, C., van Passel, M. W., Brouns, S. J. CRISPR interference directs strand specific spacer acquisition. PLoS One. 7 (4), e35888 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Kleinstiver, B. P., et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition. Nature Biotechnology. 33 (12), 1293-1298 (2015).
- Kleinstiver, B. P., et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature. 523 (7561), 481-485 (2015).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Jinek, M., et al. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Cho, S. W., Kim, S., Kim, J. M., Kim, J. S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nature Biotechnology. 31 (3), 230-232 (2013).
- Wang, Y., et al. Systematic evaluation of CRISPR-Cas systems reveals design principles for genome editing in human cells. Genome Biology. 19 (1), 62 (2018).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences U S A. 110 (39), 15644-15649 (2013).
- Kim, E., et al. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni. Nature Communications. 8, 14500 (2017).
- Edraki, A., et al. A Compact, High-Accuracy Cas9 with a Dinucleotide PAM for In Vivo Genome Editing. Molecular Cell. , (2018).
- Chatterjee, P., Jakimo, N., Jacobson, J. M. Minimal PAM specificity of a highly similar SpCas9 ortholog. Science Advances. 4 (10), (2018).
- Muller, M., et al. Streptococcus thermophilus CRISPR-Cas9 Systems Enable Specific Editing of the Human Genome. Mol Therapy. 24 (3), 636-644 (2016).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Boratyn, G. M., et al. BLAST: a more efficient report with usability improvements. Nucleic Acids Research. 41 (Web Server issue), W29-W33 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Montague, T. G., Cruz, J. M., Gagnon, J. A., Church, G. M., Valen, E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research. 42 (Web Server issue), W401-W407 (2014).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 122-123 (2014).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30 (10), 1473-1475 (2014).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Richardson, C. D., Ray, G. J., Bray, N. L., Corn, J. E. Non-homologous DNA increases gene disruption efficiency by altering DNA repair outcomes. Nature Communications. 7, 12463 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biology. 18 (1), 35 (2017).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shmakov, S., et al. Diversity and evolution of class 2 CRISPR-Cas systems. Nature Reviews Microbiology. 15 (3), 169-182 (2017).
- Moreno-Mateos, M. A., et al. CRISPR-Cpf1 mediates efficient homology-directed repair and temperature-controlled genome editing. Nature Communications. 8 (1), 2024 (2017).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Yang, L., et al. Optimization of scarless human stem cell genome editing. Nucleic Acids Research. 41 (19), 9049-9061 (2013).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).

