Биоинформатика трубопровод точно и эффективно анализировать microRNA транскриптомы в растениях
Summary
Конвейер биоинформатики, а именно miRDeep-P2 (miRDP2 для краткости), с обновленными критериями miRNA растений и пересмотренным алгоритмом, может точно и эффективно анализировать транскриптомы микроРНК в растениях, особенно для видов со сложными и крупными геномами.
Abstract
МикроРНК (миРНК) составляют от 20 до 24 нуклеотидов (нт) эндогенных малых РНК (РНК), широко существующих у растений и животных, которые играют мощную роль в регулировании экспрессии генов на посттранскрипционном уровне. Секвенирование библиотек sRNA методами секвенирования следующего поколения (NGS) широко использовалось для выявления и анализа транскриптомов miRNA в последнее десятилетие, что привело к быстрому увеличению числа обнаружений miRNA. Тем не менее, две основные проблемы возникают в аннотации miRNA растений из-за увеличения глубины секвенированных библиотек sRNA, а также размер и сложность геномов растений. Во-первых, многие другие типы СРНК, в частности, короткие интерферинговые РНК (siRNAs) из библиотек сРНК, ошибочно аннотируются как миРНК многими вычислительными инструментами. Во-вторых, он становится чрезвычайно трудоемким процессом для анализа транскриптомов miRNA у видов растений с большими и сложными геномами. Чтобы преодолеть эти проблемы, мы недавно модернизировали miRDeep-P (популярный инструмент для анализа транскриптома miRNA) до miRDeep-P2 (miRDP2 для краткости) с использованием новой стратегии фильтрации, пересмотра алгоритма скоринга и включения недавно обновленного завода miRNA критерии аннотации. Мы протестировали miRDP2 против секвенированных популяций сРНК на пяти репрезентативных растениях с возрастающей геномной сложностью, включая арабидопсис, рис, помидоры, кукурузу и пшеницу. Результаты показывают, что miRDP2 обрабатывают эти задачи с очень высокой эффективностью. Кроме того, miRDP2 превзошел другие инструменты прогнозирования в отношении чувствительности и точности. Взятые вместе, наши результаты демонстрируют miRDP2 как быстрый и точный инструмент для анализа транскриптомы miRNA растений, поэтому полезный инструмент, помогающий сообществу лучше аннотировать миРНК в растениях.
Introduction
Одним из самых захватывающих открытий за последние два десятилетия в биологии является распространяющаяся роль видов sRNA в регулировании различных функций генома1. В частности, miRNAs представляют собой важный класс от 20 до 24-nt sRNAs в эукариоты, и в основном функции на посттранскрипционном уровне в качестве видных регуляторов генов на протяжении всей стадии развития жизненного цикла, а также в стимулировочных и стрессовых реакций2,3. В растениях, miRNAs возникают из первичных стенограмм ы называется pri-miRNAs, которые, как правило, транскрибируется РНК полимеразы II как отдельные блоки транскрипции4,5. Обработанные эволюционно сохраненным клеточным оборудованием (Drosha RNase III у животных, DICER-как в растениях), при-миРНК иссечены в непосредственные прекурсоры miRNA, предварительно миРНК, которые содержат последовательности, образующие внутримолекулярные структуры стволовой петли6,7. Pre-miRNAs затем обрабатываются в двухцепочечные промежуточные, а именно miRNA дуплексы, состоящие из функциональной нити, зрелые miRNA, и реже функциональный партнер, miRNA2,8. После загрузки в РНК-индуцированной глушитель комплекса (RISC), зрелые miRNAs может распознавать их мРНК цели на основе последовательности взаимодополняемости, в результате чего негативная регулятивная функция2,8. miRNAs может либо дестабилизировать свои целевые стенограммы или предотвратить целевой перевод, но в первом случае доминируют в растениях8,9.
С случайного открытия первого miRNA в nematode Caenorhabditis elegans10,11, много исследований было совершено для идентификации miRNA и его функциональный анализ, особенно после наличия метода NGS. Широкое применение метода NGS значительно способствовало использованию вычислительных инструментов, которые были разработаны для захвата уникальной особенностью miRNAs, таких как структура стволовых петли прекурсоров и их преференциальное накопление последовательности читает на зрелых miRNA и miRNA. В результате исследователи добились значительных успехов в выявлении miRNAs у различных видов. Основываясь на ранее описанной вероятностной модели12, мы разработали miRDeep-P13, который был первым вычислительным инструментом для обнаружения растительных миРНК из данных NGS. miRDeep-P был специально направлен на завоевание проблем декодирования завода miRNAs с более переменной длиной прекурсоров и большими парализующими семьями13,14,15. После его выпуска, эта программа была загружена тысячи раз и используется для аннотации miRNA транскриптомы в более чем 40 видов растений16. Propelled NGS-основанных инструментов, таких как miRDeep-P, наблюдается резкое увеличение числа зарегистрированных miRNAs в общественном репозитории miRNA miRBase17, где более 38000 miRNA пунктов в настоящее время размещается (выпуск 22.1) по сравнению только с 500 miRNA пунктов (выпуск 2.0) в 200818.
Тем не менее, две новые проблемы возникли из аннотации miRNA завода. Во-первых, высокие коэффициенты ложных срабатываний оказали значительное влияние на качество аннотации миннакирастений 16,19 по следующим причинам: 1) поток эндогенных короткой интерферинговых РНК (siRNAs) из библиотек NGS sRNA были ошибочно аннотированы как миРНК из-за отсутствия строгих критериев аннотации миРНК; 2) для видов без информации miRNA, ложные срабатывания предсказанные на основе данных NGS трудно устранить. Используя miRBase в качестве примера, Taylor et al.20 обнаружили, что одна треть записей растительной miRNA в общественном хранилище21 (выпуск 21) не имеет убедительных подтверждающих доказательств, и даже три четверти семейсемей сярей miRNA были сомнительными. Во-вторых, он становится чрезвычайно трудоемким процессом для прогнозирования растительных миРНК с большими и сложными геномами16. Чтобы преодолеть эти проблемы, мы обновили miRDeep-P, добавив новую стратегию фильтрации, перепланировку алгоритма скоринга и интеграцию новых критериев для аннотации miRNA завода, и выпустили новую версию miRDP2. Кроме того, мы протестировали miRDP2 с использованием наборов данных NGS sRNA с постепенным увеличением размеров генома: арабидопсис, рис, помидоры, кукуруза и пшеница. По сравнению с другими пятью широко используемыми инструментами и его старой версией, miRDP2 разогнал эти данные sRNA и проанализировал транскриптомы miRNA быстрее с улучшенной точностью и чувствительностью.
Содержимое пакета miRDP2
Пакет miRDP2 состоит из шести задокументированных скриптов Perl, которые должны быть запущены последовательно подготовленным скриптом Bash. Из шести скриптов три(convert_bowtie_to_blast.pl, filter_alignments.pl,и excise_candidate.pl)унаследованы от miRDeep-P. Другие скрипты изменены из исходной версии. Функции шести скриптов описаны следующим образом:
preprocess_reads.pl фильтры ввода читает, в том числе читает, которые являются слишком длинными или слишком короткими (Злт;19 nt или йgt;25 nt), и читает коррелирует с Rfam ncRNA последовательностей, а также читает с RPM (читает на миллион) менее 5. Затем скрипт извлекает считываемые, коррелированные с известными зрелыми последовательностями miRNA. Файлы ввода являются оригинальными считываемыми в формате FASTA/FASTи и выходом bowtie2 считываний на miRNA и ncRNA последовательностей.
Формула расчета RPM:
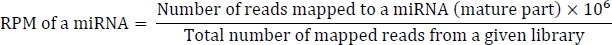
convert_bowtie_to_blast.pl изменяет формат bowtie в BLAST-разбора формата. BLAST-parsed формат представляет собой пользовательский табулярный разделенный формат, полученный от стандартного формата NCBI BLASToutput.
filter_alignments.pl фильтрует выравнивания глубокого секвенирования считывает на геном. Он фильтрует частичные выравнивания, а также многовыровные считывавшие ся (определенное пользователем частотное отсечение). Основным входом является файл в формате BLAST-parsed.
excise_candidate.pl вырезает потенциальные последовательности прекурсоров из референтной последовательности, используя выровненные чтения в качестве руководящих принципов. Основным входом является файл в формате BLAST-parsed и файл FASTA. Выход — это все потенциальные последовательности прекурсоров в формате FASTA.
mod-miRDP.pl требуется два файла ввода, файл подписи и файл структуры, который изменяется из основного алгоритма miRDeep-P путем изменения системы скоринга с конкретными параметрами завода. Файлы ввода представляют сяртово-кронштейнный файл структуры прекурсоров и считывают файл подписи распределения.
mod-rm_redundant_meet_plant.pl нуждается в трех файлах ввода: chromosome_length, прекурсорах и original_prediction, генерируемых mod-miRDP.pl. Он генерирует два выходных файла, неизлишний прогнозируемый файл и прогнозируемый файл, отфильтрованный по недавно обновленным критериям miRNA завода. Подробная информация о формате вывода файла описана в разделе 1.4.
Protocol
Representative Results
Discussion
С появлением NGS, большое количество loci miRNA были определены из постоянно растущего количества данных секвенирования sRNA в различных видах29,30. В централизованной базе данных сообщества miRBase21, депонированные элементы miRNA увеличились почти в 100 ра…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Эта работа была поддержана Пекинской академией сельскохозяйственных и лесных наук (KJCX201917, KJCX20180425 и KJCX20180204) в XY и Национальный фонд естественных наук Китая (31621001) в LL.
Materials
| Computer/computing node | N/A | N/A | Perl is required; at least 8 GB RAM and 100 GB storage are recommended |
References
- Ghildiyal, M., Zamore, P. D. Small silencing RNAs: an expanding universe. Nature Reviews Genetics. 10 (2), 94-108 (2009).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136 (2), 215-233 (2009).
- Moran, Y., Agron, M., Praher, D., Technau, U. The evolutionary origin of plant and animal microRNAs. Nature Ecology Evolution. 1 (3), 27 (2017).
- Xie, Z., et al. Expression of Arabidopsis MIRNA genes. Plant Physiology. 138 (4), 2145-2154 (2005).
- Zhao, X., Zhang, H., Li, L. Identification and analysis of the proximal promoters of microRNA genes in Arabidopsis. Genomics. 101 (3), 187-194 (2013).
- Bologna, N. G., Mateos, J. L., Bresso, E. G., Palatnik, J. F. A loop-to-base processing mechanism underlies the biogenesis of plant microRNAs miR319 and miR159. EMBO JOURNAL. 28 (23), 3646-3656 (2009).
- Rogers, K., Chen, X. Biogenesis, turnover, and mode of action of plant microRNAs. Plant Cell. 25 (7), 2383-2399 (2013).
- Voinnet, O. Origin, biogenesis, and activity of plant microRNAs. Cell. 136 (4), 669-687 (2009).
- Iwakawa, H. O., Tomari, Y. The Functions of MicroRNAs: mRNA Decay and Translational Repression. Trends in Cell Biology. 25 (11), 651-665 (2015).
- Lee, R. C., Feinbaum, R. L., Ambros, V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 75 (5), 843-854 (1993).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75 (5), 855-862 (1993).
- Friedlander, M. R., et al. Discovering microRNAs from deep sequencing data using miRDeep. Nature Biotechnology. 26 (4), 407-415 (2008).
- Yang, X., Li, L. miRDeep-P: a computational tool for analyzing the microRNA transcriptome in plants. Bioinformatics. 27 (18), 2614-2615 (2011).
- Meyers, B. C., et al. Criteria for annotation of plant MicroRNAs. Plant Cell. 20 (12), 3186-3190 (2008).
- Yang, X., Zhang, H., Li, L. Global analysis of gene-level microRNA expression in Arabidopsis using deep sequencing data. Genomics. 98 (1), 40-46 (2011).
- Kuang, Z., Wang, Y., Li, L., Yang, X. miRDeep-P2: accurate and fast analysis of the microRNA transcriptome in plants. Bioinformatics. , (2018).
- Kozomara, A., Birgaoanu, M., Griffiths-Jones, S. miRBase: from microRNA sequences to function. Nucleic Acids Research. 47 (1), 155-162 (2019).
- Griffiths-Jones, S., Saini, H. K., van Dongen, S., Enright, A. J. miRBase: tools for microRNA genomics. Nucleic Acids Research. 36, 154-158 (2008).
- Axtell, M. J., Meyers, B. C. Revisiting Criteria for Plant MicroRNA Annotation in the Era of Big Data. Plant Cell. 30 (2), 272-284 (2018).
- Taylor, R. S., Tarver, J. E., Hiscock, S. J., Donoghue, P. C. Evolutionary history of plant microRNAs. Trends in Plant Science. 19 (3), 175-182 (2014).
- Kozomara, A., Griffiths-Jones, S. miRBase: annotating high confidence microRNAs using deep sequencing data. Nucleic Acids Research. 42, 68-73 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Lorenz, R., et al. ViennaRNA Package 2.0. Algorithms for Molecular Biology. 6, 26 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (3), 25 (2009).
- An, J., Lai, J., Sajjanhar, A., Lehman, M. L., Nelson, C. C. miRPlant: an integrated tool for identification of plant miRNA from RNA sequencing data. BMC Bioinformatics. 15, 275 (2014).
- Lei, J., Sun, Y. miR-PREFeR: an accurate, fast and easy-to-use plant miRNA prediction tool using small RNA-Seq data. Bioinformatics. 30 (19), 2837-2839 (2014).
- Evers, M., Huttner, M., Dueck, A., Meister, G., Engelmann, J. C. miRA: adaptable novel miRNA identification in plants using small RNA sequencing data. BMC Bioinformatics. 16, 370 (2015).
- Mathelier, A., Carbone, A. MIReNA: finding microRNAs with high accuracy and no learning at genome scale and from deep sequencing data. Bioinformatics. 26 (18), 2226-2234 (2010).
- Zhu, Q. H., et al. A diverse set of microRNAs and microRNA-like small RNAs in developing rice grains. Genome Research. 18 (9), 1456-1465 (2008).
- Fahlgren, N., et al. MicroRNA gene evolution in Arabidopsis lyrata and Arabidopsis thaliana. Plant Cell. 22 (4), 1074-1089 (2010).
- Fromm, B., et al. A Uniform System for the Annotation of Vertebrate microRNA Genes and the Evolution of the Human microRNAome. Annual Review of Genetics. 49, 213-242 (2015).
- Blevins, T., et al. Identification of Pol IV and RDR2-dependent precursors of 24 nt siRNAs guiding de novo DNA methylation in Arabidopsis. Elife. 4, 09591 (2015).
- Zhai, J., et al. A One Precursor One siRNA Model for Pol IV-Dependent siRNA Biogenesis. Cell. 163 (2), 445-455 (2015).
- Werner, S., Wollmann, H., Schneeberger, K., Weigel, D. Structure determinants for accurate processing of miR172a in Arabidopsis thaliana. Current Biology. 20 (1), 42-48 (2010).
- Mateos, J. L., Bologna, N. G., Chorostecki, U., Palatnik, J. F. Identification of microRNA processing determinants by random mutagenesis of Arabidopsis MIR172a precursor. Current Biology. 20 (1), 49-54 (2010).
- Vitsios, D. M., et al. Mirnovo: genome-free prediction of microRNAs from small RNA sequencing data and single-cells using decision forests. Nucleic Acids Research. 45 (21), 177 (2017).

