Identifikation af alternativ splejsning og polyadenylering i RNA-seq-data
Summary
Alternativ splejsning (AS) og alternativ polyadenylering (APA) udvider mangfoldigheden af transkriptionsisoformer og deres produkter. Her beskriver vi bioinformatiske protokoller til analyse af bulk RNA-seq og 3 ‘slutsekventeringsassays for at detektere og visualisere AS og APA, der varierer på tværs af eksperimentelle forhold.
Abstract
Ud over den typiske analyse af RNA-Seq til måling af differentiel genekspression (DGE) på tværs af eksperimentelle / biologiske tilstande kan RNA-seq-data også bruges til at udforske andre komplekse reguleringsmekanismer på exon-niveau. Alternativ splejsning og polyadenylering spiller en afgørende rolle i et gens funktionelle mangfoldighed ved at generere forskellige isoformer til regulering af genekspression på post-transkriptionelt niveau, og begrænsning af analyser til hele genniveauet kan gå glip af dette vigtige regulatoriske lag. Her demonstrerer vi detaljerede trin for trin analyser til identifikation og visualisering af differentiel exon og polyadenylering site brug på tværs af betingelser ved hjælp af Bioconductor og andre pakker og funktioner, herunder DEXSeq, diffSplice fra Limma-pakken og rMATS.
Introduction
RNA-seq har været meget udbredt gennem årene typisk til estimering af differentiel genekspression og genopdagelse1. Derudover kan det også bruges til at estimere varierende exon-niveau brug på grund af gen, der udtrykker forskellige isoformer, hvilket bidrager til en bedre forståelse af genregulering på post-transkriptionelt niveau. Størstedelen af eukaryote gener genererer forskellige isoformer ved alternativ splejsning (AS) for at øge mangfoldigheden af mRNA-ekspression. AS-begivenheder kan opdeles i forskellige mønstre: spring over komplette exons (SE), hvor en (“kassette”) exon fjernes helt ud af udskriften sammen med dens flankerende introns; alternativ (donor) 5′ splejsningssted (A5SS) og alternativ 3′ (acceptor) splejsningssted (A3SS), når to eller flere splejsningssteder er til stede i hver ende af en exon retention af introns (RI), når en intron bevares inden for det modne mRNA-transkript og gensidig udelukkelse af exonbrug (MXE), hvor kun en af de to tilgængelige exoner kan bevares ad gangen 2,3. Alternativ polyadenylering (APA) spiller også en vigtig rolle i reguleringen af genekspression ved hjælp af alternative poly (A) steder til at generere flere mRNA-isoformer fra et enkelt transkript4. De fleste polyadenyleringssteder (pA’er) er placeret i den 3′ uoversatte region (3′ UTR’er), der genererer mRNA-isoformer med forskellige 3′ UTR-længder. Da 3′ UTR er det centrale knudepunkt for genkendelse af regulatoriske elementer, kan forskellige 3′ UTR-længder påvirke mRNA-lokalisering, stabilitet og translation5. Der er en klasse af 3 ‘ende sekventeringsassays optimeret til at detektere APA, der adskiller sig i detaljerne i protokollen6. Rørledningen beskrevet her er designet til PolyA-seq, men kan tilpasses til andre protokoller som beskrevet.
I denne undersøgelse præsenterer vi en pipeline af differentielle exonanalysemetoder7,8 (figur 1), som kan opdeles i to brede kategorier: exonbaseret (DEXSeq9, diffSplice 10) og begivenhedsbaseret (replikeret multivariat analyse af transkriptionsplejsning (rMATS)11). De exon-baserede metoder sammenligner foldændringen på tværs af betingelser for individuelle exoner mod et mål for den samlede genfoldændring for at kalde differentielt udtrykt exon-brug og ud fra det beregne et mål på genniveau for AS-aktivitet. Hændelsesbaserede metoder bruger exon-intron-spanning junction-læsninger til at registrere og klassificere specifikke splejsningshændelser såsom exon-spring eller tilbageholdelse af introns og skelne mellem disse AS-typer i output3. Disse metoder giver således supplerende synspunkter til en komplet analyse af AS12,13. Vi valgte DEXSeq (baseret på DESeq214 DGE-pakken) og diffSplice (baseret på Limma10 DGE-pakken) til undersøgelsen, da de er blandt de mest anvendte pakker til differentiel splejsningsanalyse. rMATS blev valgt som en populær metode til begivenhedsbaseret analyse. En anden populær begivenhedsbaseret metode er MISO (Blanding af isoformer)1. For APA tilpasser vi den exon-baserede tilgang.
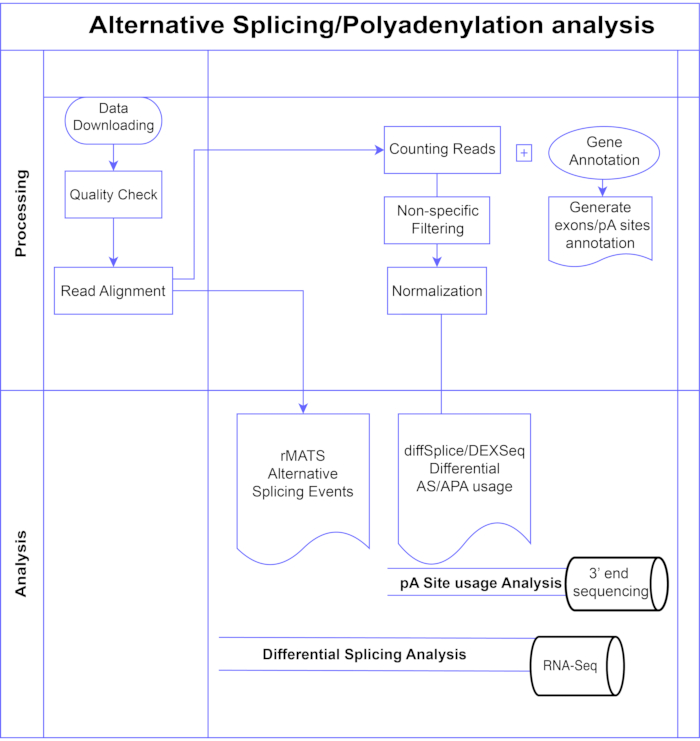
Figur 1. Analyse pipeline. Rutediagram over de trin, der bruges i analysen. Trin inkluderer: indhentning af data, udførelse af kvalitetskontrol og læsejustering efterfulgt af optælling af læsninger ved hjælp af kommentarer til kendte exons, introns og pA-websteder, filtrering for at fjerne lave tællinger og normalisering. PolyA-seq-data blev analyseret for alternative pA-steder ved hjælp af diffSplice/DEXSeq-metoder, bulk RNA-Seq blev analyseret for alternativ splejsning på exonniveau med diffSplice/DEXseq-metoder og AS-hændelser analyseret med rMATS. Klik her for at se en større version af denne figur.
RNA-seq-dataene, der blev brugt i denne undersøgelse, blev erhvervet fra Gene Expression Omnibus (GEO) (GSE138691)15. Vi brugte mus RNA-seq data fra denne undersøgelse med to tilstandsgrupper: wild-type (WT) og muskelblind-lignende type 1 knockout (Mbnl1 KO) med tre replikater hver. For at demonstrere differentiel analyse af brugen af polyadenyleringsstedet opnåede vi museembryofibroblaster (MEF’er) PolyA-seq-data (GEO-tiltrædelse GSE60487)16. Dataene har fire tilstandsgrupper: Wild-type (WT), Muscleblind-lignende type1/type 2 dobbelt knockout (Mbnl1/2 DKO), Mbnl 1/2 DKO med Mbnl3 knockdown (KD) og Mbnl1/2 DKO med Mbnl3 kontrol (Ctrl). Hver tilstandsgruppe består af to replikater.
| GEO tiltrædelse | SRA Kør nummer | Eksempel på navn | Betingelse | Kopiere | Væv | Sekventering | Læs længde | |
| RNA-Seq | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1 knockout | Rep 1 | Thymus | Parret ende | 100 bp |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1 knockout | Repræsentant 2 | Thymus | Parret ende | 100 bp | |
| GSM4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1 knockout | Rep 3 | Thymus | Parret ende | 100 bp | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | Vild type | Rep 1 | Thymus | Parret ende | 100 bp | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | Vild type | Repræsentant 2 | Thymus | Parret ende | 100 bp | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | Vild type | Rep 3 | Thymus | Parret ende | 100 bp | |
| 3P-Seq | GSM1480973 | SRR1553129 | WT_1 | Vildtype (WT) | Rep 1 | Museembryonale fibroblaster (MEF’er) | Single-end | 40 bp |
| GSM1480974 | SRR1553130 | WT_2 | Vildtype (WT) | Repræsentant 2 | Museembryonale fibroblaster (MEF’er) | Single-end | 40 bp | |
| GSM1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 dobbelt knockout (DKO) | Rep 1 | Museembryonale fibroblaster (MEF’er) | Single-end | 40 bp | |
| GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 dobbelt knockout (DKO) | Repræsentant 2 | Museembryonale fibroblaster (MEF’er) | Single-end | 40 bp | |
| GSM1480977 | SRR1553133 | DKOsiRNA_1 | Mbnl 1/2 dobbelt knockout med Mbnl 3 siRNA (KD) | Rep 1 | Museembryonale fibroblaster (MEF’er) | Single-end | 40 bp | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 1/2 dobbelt knockout med Mbnl 3 siRNA (KD) | Repræsentant 2 | Museembryonale fibroblaster (MEF’er) | Single-end | 36 bp | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | Mbnl 1/2 dobbelt knockout med ikke-målrettet siRNA (Ctrl) | Rep 1 | Museembryonale fibroblaster (MEF’er) | Single-end | 40 bp | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 dobbelt knockout med ikke-målrettet siRNA (Ctrl) | Repræsentant 2 | Museembryonale fibroblaster (MEF’er) | Single-end | 40 bp |
Tabel 1. Resumé af RNA-Seq og PolyA-seq datasæt, der anvendes til analysen.
Protocol
Representative Results
Discussion
I denne undersøgelse evaluerede vi exon-baserede og begivenhedsbaserede tilgange til at detektere AS og APA i bulk RNA-Seq og 3 ‘slutsekventeringsdata. De exonbaserede AS-tilgange producerer både en liste over differentielt udtrykte exoner og en rangordning på genniveau ordnet efter den statistiske signifikans af den samlede differentielle splejsningsaktivitet på genniveau (tabel 1-2, 4-5). For diffSplice-pakken bestemmes differentiel brug ved at tilpasse vægtede lineære modeller på exon-niveau fo…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Denne undersøgelse blev støttet af et australsk forskningsråd (ARC) Future Fellowship (FT16010043) og ANU Futures Scheme.
References
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3′ end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).

