आरएनए-सेक डेटा में वैकल्पिक स्प्लिसिंग और पॉलीएडेनाइलेशन की पहचान
Summary
वैकल्पिक स्प्लिसिंग (एएस) और वैकल्पिक पॉलीएडेनाइलेशन (एपीए) ट्रांसक्रिप्ट आइसोफॉर्म और उनके उत्पादों की विविधता का विस्तार करते हैं। यहां, हम प्रयोगात्मक स्थितियों में अलग-अलग एएस और एपीए का पता लगाने और कल्पना करने के लिए थोक आरएनए-सेक और 3 ‘एंड सीक्वेंसिंग परख का विश्लेषण करने के लिए जैव सूचना विज्ञान प्रोटोकॉल का वर्णन करते हैं।
Abstract
जैविक स्थितियों में विभेदक जीन अभिव्यक्ति (डीजीई) को मापने के लिए आरएनए-सेक के विशिष्ट विश्लेषण के साथ-साथ, आरएनए-सेक डेटा का उपयोग एक्सॉन स्तर पर अन्य जटिल नियामक तंत्रों का पता लगाने के लिए भी किया जा सकता है। वैकल्पिक स्प्लिसिंग और पॉलीएडेनाइलेशन पोस्ट-ट्रांसक्रिप्शनल स्तर पर जीन अभिव्यक्ति को विनियमित करने के लिए विभिन्न आइसोफॉर्म उत्पन्न करके जीन की कार्यात्मक विविधता में महत्वपूर्ण भूमिका निभाते हैं, और पूरे जीन स्तर तक विश्लेषण को सीमित करने से इस महत्वपूर्ण नियामक परत को याद किया जा सकता है। यहां, हम बायोकंडक्टर और अन्य पैकेजों और कार्यों का उपयोग करके स्थितियों में अंतर एक्सॉन और पॉलीएडेनाइलेशन साइट उपयोग की पहचान और विज़ुअलाइज़ेशन के लिए विस्तृत चरण-दर-चरण विश्लेषण प्रदर्शित करते हैं, जिसमें डेक्ससेक, लिम्मा पैकेज से डिफस्प्लिस और आरएमएटीएस शामिल हैं।
Introduction
आरएनए-सेक का व्यापक रूप से वर्षों से व्यापक रूप से उपयोग किया गया है आमतौर पर अंतर जीन अभिव्यक्ति और जीन खोज1 का आकलन करने के लिए। इसके अलावा, इसका उपयोग विभिन्न आइसोफॉर्म व्यक्त करने वाले जीन के कारण अलग-अलग एक्सॉन स्तर के उपयोग का अनुमान लगाने के लिए भी किया जा सकता है, इसलिए पोस्ट-ट्रांसक्रिप्शनल स्तर पर जीन विनियमन की बेहतर समझ में योगदान देता है। यूकेरियोटिक जीन के बहुमत एमआरएनए अभिव्यक्ति की विविधता को बढ़ाने के लिए वैकल्पिक स्प्लिसिंग (एएस) द्वारा विभिन्न आइसोफॉर्म उत्पन्न करते हैं। एएस घटनाओं को विभिन्न पैटर्नों में विभाजित किया जा सकता है: पूर्ण एक्सॉन (एसई) को छोड़ना जहां एक (“कैसेट”) एक्सॉन को इसके फ्लैंकिंग इंट्रोन्स के साथ प्रतिलेख से पूरी तरह से हटा दिया जाता है; वैकल्पिक (दाता) 5 ‘स्प्लिस साइट चयन (ए 5 एसएस) और वैकल्पिक 3 ‘ (स्वीकर्ता) स्प्लिस साइट चयन (ए 3 एसएस) जब एक्सॉन के दोनों छोर पर दो या दो से अधिक स्प्लिस साइटें मौजूद होती हैं; इंट्रोन्स (आरआई) का प्रतिधारण जब एक इंट्रोन को परिपक्व एमआरएनए प्रतिलेख और एक्सॉन उपयोग (एमएक्सई) के पारस्परिक बहिष्करण के भीतर बनाए रखा जाता है, जहां दो उपलब्ध एक्सॉन में से केवल एक कोएक समय में बनाए रखा जा सकता है। वैकल्पिक पॉलीएडेनाइलेशन (एपीए) एक एकल प्रतिलेख4 से कई एमआरएनए आइसोफॉर्म उत्पन्न करने के लिए वैकल्पिक पॉली (ए) साइटों का उपयोग करके जीन अभिव्यक्ति को विनियमित करने में भी महत्वपूर्ण भूमिका निभाता है। अधिकांश पॉलीएडेनाइलेशन साइटें (पीए) 3 ‘अअनुवादित क्षेत्र (3’ यूटीआर) में स्थित हैं, जो विविध 3 ‘ यूटीआर लंबाई के साथ एमआरएनए आइसोफॉर्म उत्पन्न करती हैं। चूंकि 3 ‘यूटीआर नियामक तत्वों को पहचानने के लिए केंद्रीय केंद्र है, इसलिए विभिन्न 3 ‘यूटीआर लंबाई एमआरएनए स्थानीयकरण, स्थिरता और अनुवाद5 को प्रभावित कर सकती है। एपीए का पता लगाने के लिए अनुकूलित 3 ‘अंत अनुक्रमण परखों का एक वर्ग है जो प्रोटोकॉल6 के विवरण में भिन्न है। यहां वर्णित पाइपलाइन पॉलीए-सेक के लिए डिज़ाइन की गई है, लेकिन वर्णित अन्य प्रोटोकॉल के लिए अनुकूलित किया जा सकता है।
इस अध्ययन में, हम विभेदक एक्सॉन विश्लेषण विधियों 7,8 (चित्रा 1) की एक पाइपलाइन प्रस्तुत करते हैं, जिसे दो व्यापक श्रेणियों में विभाजित किया जा सकता है: एक्सॉन-आधारित (DEXSeq9, डिफस्पिस्प्लिस10) और इवेंट-आधारित (ट्रांसक्रिप्ट स्प्लिसिंग के मल्टीवेरिएट विश्लेषण (rMATS)11 को दोहराएं)। एक्सॉन-आधारित विधियां अलग-अलग एक्सॉन की स्थितियों में गुना परिवर्तन की तुलना करती हैं, समग्र जीन फोल्ड परिवर्तन के माप के खिलाफ अलग-अलग व्यक्त एक्सॉन उपयोग को कॉल करती हैं, और इससे एएस गतिविधि के जीन-स्तर माप की गणना करती हैं। इवेंट-आधारित विधियां एक्सॉन-इंट्रॉन-पैनिंग जंक्शन रीड का उपयोग विशिष्ट स्प्लिसिंग घटनाओं का पता लगाने और वर्गीकृत करने के लिए करती हैं जैसे कि एक्सॉन स्किपिंग या इंट्रोन्स के प्रतिधारण, और आउटपुट3 में इन एएस प्रकारों को अलग करना। इस प्रकार, ये विधियां एएस12,13 के पूर्ण विश्लेषण के लिए पूरक विचार प्रदान करती हैं। हमने अध्ययन के लिए DEXSeq (DESeq214 DGE पैकेज के आधार पर) और डिफस्प्लिस (लिम्मा10 DGE पैकेज के आधार पर) का चयन किया क्योंकि वे विभेदक स्प्लिसिंग विश्लेषण के लिए सबसे व्यापक रूप से उपयोग किए जाने वाले पैकेजों में से हैं। आरएमएटीएस को घटना-आधारित विश्लेषण के लिए एक लोकप्रिय विधि के रूप में चुना गया था। एक और लोकप्रिय घटना-आधारित विधि एमआईएसओ (आइसोफॉर्म का मिश्रण) 1 है। एपीए के लिए हम एक्सॉन-आधारित दृष्टिकोण को अनुकूलित करते हैं।
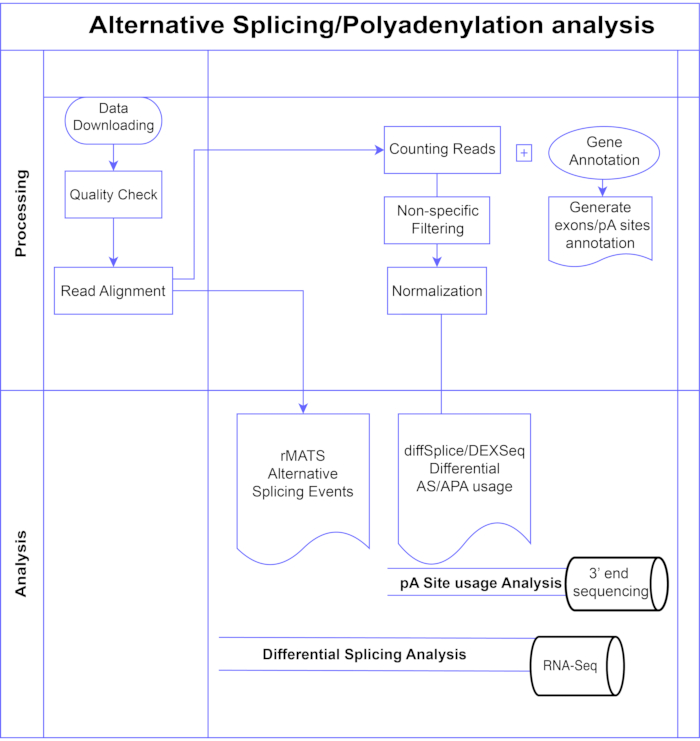
चित्र 1. विश्लेषण पाइपलाइन। विश्लेषण में उपयोग किए गए चरणों का फ़्लोचार्ट। चरणों में शामिल हैं: डेटा प्राप्त करना, गुणवत्ता जांच करना और संरेखण पढ़ना, जिसके बाद ज्ञात एक्सॉन, इंट्रोन्स और पीए साइटों के लिए एनोटेशन का उपयोग करके रीड की गिनती, कम गिनती को हटाने और सामान्यीकरण के लिए फ़िल्टर करना। डिफस्प्लिस/डीईएक्ससेक विधियों का उपयोग करके वैकल्पिक पीए साइटों के लिए पॉलीए-सेक डेटा का विश्लेषण किया गया था, थोक आरएनए-सेक का विश्लेषण एक्सॉन स्तर पर वैकल्पिक स्प्लिसिंग के लिए डिफस्प्लिस / डीईएक्ससेक विधियों के साथ किया गया था, और एएस घटनाओं का विश्लेषण आरएमएटीएस के साथ किया गया था। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।
इस सर्वेक्षण में उपयोग किए गए आरएनए-सेक डेटा को जीन एक्सप्रेशन ओमनीबस (जीईओ) (जीएसई 138691)15 से प्राप्त किया गया था। हमने इस अध्ययन से माउस आरएनए-सेक डेटा का उपयोग दो स्थिति समूहों के साथ किया: वाइल्ड-टाइप (डब्ल्यूटी) और मसलब्लाइंड जैसे टाइप 1 नॉकआउट (एमबीएनएल 1 केओ) प्रत्येक में तीन प्रतिकृतियां थीं। विभेदक पॉलीएडेनाइलेशन साइट उपयोग विश्लेषण का प्रदर्शन करने के लिए, हमने माउस भ्रूण फाइब्रोब्लास्ट (एमईएफ) पॉलीए-सेक डेटा (जीईओ परिग्रहण जीएसई 60487)16 प्राप्त किया। डेटा में चार स्थिति समूह हैं: वाइल्ड-टाइप (डब्ल्यूटी), मसलब्लाइंड जैसे टाइप 1/टाइप 2 डबल नॉकआउट (एमबीएनएल 1/2 डीकेओ), एमबीएनएल 1/2 डीकेओ के साथ एमबीएनएल 3 वध (केडी) और एमबीएनएल 3 नियंत्रण (सीटीआरएल) के साथ एमबीएनएल 1/2 डीकेओ। प्रत्येक स्थिति समूह में दो प्रतिकृतियां होती हैं।
| जीईओ परिग्रहण | SRA रन नंबर | नमूना नाम | दशा | प्रतिकृति | ऊतक | अनुक्रमण | पढ़ने की लंबाई | |
| आरएनए-सेक | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1 नॉकआउट | प्रतिनिधि 1 | थाइमस | पेयर-एंड | 100 bp |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1 नॉकआउट | प्रतिनिधि 2 | थाइमस | पेयर-एंड | 100 bp | |
| GSM4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1 नॉकआउट | प्रतिनिधि 3 | थाइमस | पेयर-एंड | 100 bp | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | जंगली प्रकार | प्रतिनिधि 1 | थाइमस | पेयर-एंड | 100 bp | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | जंगली प्रकार | प्रतिनिधि 2 | थाइमस | पेयर-एंड | 100 bp | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | जंगली प्रकार | प्रतिनिधि 3 | थाइमस | पेयर-एंड | 100 bp | |
| 3P-Seq | GSM1480973 | SRR1553129 | WT_1 | जंगली प्रकार (WT) | प्रतिनिधि 1 | माउस भ्रूण फाइब्रोब्लास्ट (एमईएफ) | सिंगल-एंड | 40 बीपी |
| GSM1480974 | SRR1553130 | WT_2 | जंगली प्रकार (WT) | प्रतिनिधि 2 | माउस भ्रूण फाइब्रोब्लास्ट (एमईएफ) | सिंगल-एंड | 40 बीपी | |
| GSM1480975 | SRR1553131 | DKO_1 | एमबीएनएल 1/2 डबल नॉकआउट (डीकेओ) | प्रतिनिधि 1 | माउस भ्रूण फाइब्रोब्लास्ट (एमईएफ) | सिंगल-एंड | 40 बीपी | |
| GSM1480976 | SRR1553132 | DKO_2 | एमबीएनएल 1/2 डबल नॉकआउट (डीकेओ) | प्रतिनिधि 2 | माउस भ्रूण फाइब्रोब्लास्ट (एमईएफ) | सिंगल-एंड | 40 बीपी | |
| GSM1480977 | SRR1553133 | DKOsiRNA_1 | Mbnl 3 sirna (KD) के साथ Mbnl 1/2 डबल नॉकआउट | प्रतिनिधि 1 | माउस भ्रूण फाइब्रोब्लास्ट (एमईएफ) | सिंगल-एंड | 40 बीपी | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 3 sirna (KD) के साथ Mbnl 1/2 डबल नॉकआउट | प्रतिनिधि 2 | माउस भ्रूण फाइब्रोब्लास्ट (एमईएफ) | सिंगल-एंड | 36 बीपी | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | गैर-लक्ष्यीकरण सीआरएनए (Ctrl) के साथ Mbnl 1/2 डबल नॉकआउट | प्रतिनिधि 1 | माउस भ्रूण फाइब्रोब्लास्ट (एमईएफ) | सिंगल-एंड | 40 बीपी | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | गैर-लक्ष्यीकरण सीआरएनए (Ctrl) के साथ Mbnl 1/2 डबल नॉकआउट | प्रतिनिधि 2 | माउस भ्रूण फाइब्रोब्लास्ट (एमईएफ) | सिंगल-एंड | 40 बीपी |
तालिका 1. विश्लेषण के लिए उपयोग किए जाने वाले आरएनए-सेक और पॉलीए-सेक डेटासेट का सारांश।
Protocol
Representative Results
Discussion
इस अध्ययन में, हमने थोक आरएनए-सेक और 3 ‘अंत अनुक्रमण डेटा में एएस और एपीए का पता लगाने के लिए एक्सॉन-आधारित और घटना-आधारित दृष्टिकोणों का मूल्यांकन किया। एक्सॉन-आधारित एएस दृष्टिकोण अलग-अलग व्यक्त एक्सॉ…
Disclosures
The authors have nothing to disclose.
Acknowledgements
इस अध्ययन को ऑस्ट्रेलियाई अनुसंधान परिषद (एआरसी) फ्यूचर फैलोशिप (एफटी 16010043) और एएनयू फ्यूचर्स स्कीम द्वारा समर्थित किया गया था।
References
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3′ end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).

