Identifisering av alternativ spleising og polyadenylering i RNA-seq-data
Summary
Alternativ spleising (AS) og alternativ polyadenylering (APA) utvider mangfoldet av transkripsjonsisoformer og deres produkter. Her beskriver vi bioinformatiske protokoller for å analysere bulk RNA-seq og 3 ‘end sekvenseringsanalyser for å oppdage og visualisere AS og APA som varierer på tvers av eksperimentelle forhold.
Abstract
I tillegg til den typiske analysen av RNA-Seq for å måle differensialgenuttrykk (DGE) på tvers av eksperimentelle / biologiske forhold, kan RNA-seq-data også brukes til å utforske andre komplekse reguleringsmekanismer på eksonnivå. Alternativ spleising og polyadenylering spiller en avgjørende rolle i det funksjonelle mangfoldet av et gen ved å generere forskjellige isoformer for å regulere genuttrykk på posttranskripsjonsnivå, og begrensende analyser til hele gennivået kan gå glipp av dette viktige reguleringslaget. Her demonstrerer vi detaljerte trinnvise analyser for identifisering og visualisering av differensial ekson- og polyadenyleringsstedsbruk på tvers av forhold, ved bruk av Bioconductor og andre pakker og funksjoner, inkludert DEXSeq, diffSplice fra Limma-pakken og rMATS.
Introduction
RNA-seq har blitt mye brukt gjennom årene, typisk for å estimere differensial genuttrykk og genoppdagelse1. I tillegg kan det også brukes til å estimere varierende eksonnivåbruk på grunn av genuttrykk for forskjellige isoformer, og dermed bidra til en bedre forståelse av genregulering på posttranskripsjonsnivå. Flertallet av eukaryote gener genererer forskjellige isoformer ved alternativ spleising (AS) for å øke mangfoldet av mRNA-uttrykk. AS-hendelser kan deles inn i forskjellige mønstre: hopping av komplette eksoner (SE) der en (“kassett”) ekson fjernes helt ut av transkripsjonen sammen med dens flankerende introner; alternativ (donor) 5′ valg av spleisested (A5SS) og alternativ 3′ (akseptor) spleisestedsvalg (A3SS) når to eller flere skjøtesteder er til stede i hver ende av en ekson; oppbevaring av introner (RI) når et intron beholdes i det modne mRNA-transkriptet og gjensidig utelukkelse av eksonbruk (MXE) der bare ett av de to tilgjengelige eksonene kan beholdes om gangen 2,3. Alternativ polyadenylering (APA) spiller også en viktig rolle i regulering av genuttrykk ved bruk av alternative poly (A) steder for å generere flere mRNA-isoformer fra et enkelt transkripsjon4. De fleste polyadenyleringssteder (pAs) er lokalisert i 3 ‘uoversatte regionen (3’ UTRs), og genererer mRNA-isoformer med forskjellige 3 ‘UTR-lengder. Siden 3′ UTR er det sentrale knutepunktet for å gjenkjenne regulatoriske elementer, kan forskjellige 3’ UTR-lengder påvirke mRNA-lokalisering, stabilitet og oversettelse5. Det er en klasse med 3 ‘endesekvenseringsanalyser optimalisert for å oppdage APA som er forskjellige i detaljene i protokollen6. Rørledningen beskrevet her er designet for PolyA-seq, men kan tilpasses for andre protokoller som beskrevet.
I denne studien presenterer vi en pipeline av differensielle eksonanalysemetoder7,8 (figur 1), som kan deles inn i to brede kategorier: eksonbasert (DEXSeq9, diffSplice 10) og hendelsesbasert (replikat multivariat analyse av transkripsjonsspleising (rMATS)11). De eksonbaserte metodene sammenligner foldeendringen på tvers av forholdene til individuelle eksoner, mot et mål på total genfoldendring for å kalle differensielt uttrykt eksonbruk, og fra det beregnes et gennivåmål for AS-aktivitet. Hendelsesbaserte metoder bruker ekson-intron-spennende kryssavlesninger for å oppdage og klassifisere spesifikke spleisehendelser som eksonhopping eller oppbevaring av introner, og skille disse AS-typene i utgangen3. Dermed gir disse metodene komplementære visninger for en fullstendig analyse av AS12,13. Vi valgte DEXSeq (basert på DESeq214 DGE-pakken) og diffSplice (basert på Limma10 DGE-pakken) for studien, da de er blant de mest brukte pakkene for differensiell spleisingsanalyse. rMATS ble valgt som en populær metode for hendelsesbasert analyse. En annen populær hendelsesbasert metode er MISO (Blanding av isoformer)1. For TFO tilpasser vi den eksonbaserte tilnærmingen.
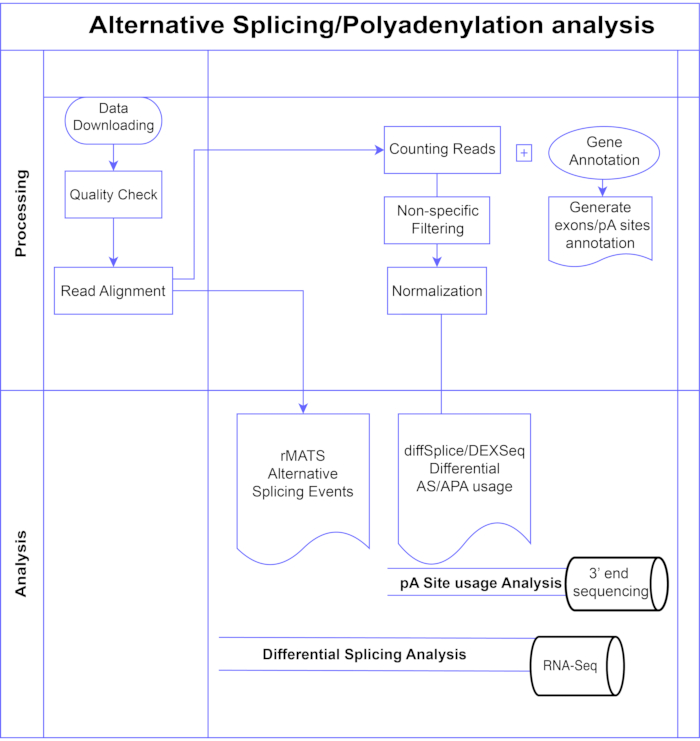
Figur 1. Analyse rørledning. Flytskjema for trinnene som brukes i analysen. Trinn inkluderer: innhenting av data, utførelse av kvalitetskontroller og lesejustering etterfulgt av telling av avlesninger ved hjelp av merknader for kjente eksoner, introner og pA-nettsteder, filtrering for å fjerne lave tellinger og normalisering. PolyA-seq-data ble analysert for alternative pA-lokaliteter med diffSplice/DEXSeq-metoder, bulk RNA-Seq ble analysert for alternativ spleising på eksonnivå med diffSplice/DEXseq-metoder, og AS-hendelser analysert med rMATS. Vennligst klikk her for å se en større versjon av denne figuren.
RNA-seq-dataene som ble brukt i denne undersøkelsen ble hentet fra Gene Expression Omnibus (GEO) (GSE138691)15. Vi brukte mus RNA-seq data fra denne studien med to tilstandsgrupper: villtype (WT) og Muskelblind-lignende type 1 knockout (Mbnl1 KO) med tre replikasjoner hver. For å demonstrere differensiell analyse av bruk av polyadenyleringsstedet, oppnådde vi museembryofibroblaster (MEFs) PolyA-seq-data (GEO Accession GSE60487)16. Dataene har fire tilstandsgrupper: Wild-type (WT), Muscleblind-lignende type1/type 2 dobbel knockout (Mbnl1/2 DKO), Mbnl 1/2 DKO med Mbnl3 knockdown (KD) og Mbnl1/2 DKO med Mbnl3 kontroll (Ctrl). Hver tilstandsgruppe består av to replikaer.
| GEO-tiltredelse | SRA Run nummer | Eksempel på navn | Betingelse | Replikere | Vev | Sekvensering | Lese lengde | |
| RNA-Seq | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1 knockout | Rep 1 | Thymus | Par-ende | 100 bp |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1 knockout | Rep 2 | Thymus | Par-ende | 100 bp | |
| GSM4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1 knockout | Rep 3 | Thymus | Par-ende | 100 bp | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | Vill type | Rep 1 | Thymus | Par-ende | 100 bp | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | Vill type | Rep 2 | Thymus | Par-ende | 100 bp | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | Vill type | Rep 3 | Thymus | Par-ende | 100 bp | |
| 3P-Seq | GSM1480973 | SRR1553129 | WT_1 | Vill type (WT) | Rep 1 | Mus embryonale fibroblaster (MEFs) | Single-end | 40 bp |
| GSM1480974 | SRR1553130 | WT_2 | Vill type (WT) | Rep 2 | Mus embryonale fibroblaster (MEFs) | Single-end | 40 bp | |
| GSM1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 dobbel knockout (DKO) | Rep 1 | Mus embryonale fibroblaster (MEFs) | Single-end | 40 bp | |
| GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 dobbel knockout (DKO) | Rep 2 | Mus embryonale fibroblaster (MEFs) | Single-end | 40 bp | |
| GSM1480977 | SRR1553133 | DKOsiRNA_1 | Mbnl 1/2 dobbel knockout med Mbnl 3 siRNA (KD) | Rep 1 | Mus embryonale fibroblaster (MEFs) | Single-end | 40 bp | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 1/2 dobbel knockout med Mbnl 3 siRNA (KD) | Rep 2 | Mus embryonale fibroblaster (MEFs) | Single-end | 36 bp | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | Mbnl 1/2 dobbel knockout med ikke-målretting siRNA (Ctrl) | Rep 1 | Mus embryonale fibroblaster (MEFs) | Single-end | 40 bp | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 dobbel knockout med ikke-målretting siRNA (Ctrl) | Rep 2 | Mus embryonale fibroblaster (MEFs) | Single-end | 40 bp |
Tabell 1. Sammendrag av RNA-Seq og PolyA-seq datasett som brukes til analysen.
Protocol
Representative Results
Discussion
I denne studien evaluerte vi eksonbaserte og hendelsesbaserte tilnærminger for å oppdage AS og APA i bulk RNA-Seq og 3 ‘end sekvenseringsdata. De eksonbaserte AS-tilnærmingene produserer både en liste over differensielt uttrykte eksoner og en gennivårangering sortert etter statistisk signifikans av samlet gennivådifferensiell spleisingsaktivitet (tabell 1-2, 4-5). For diffSplice-pakken bestemmes differensiell bruk ved å tilpasse vektede lineære modeller på eksonnivå for å estimere differensial…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Denne studien ble støttet av et Australian Research Council (ARC) Future Fellowship (FT16010043) og ANU Futures Scheme.
References
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3′ end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).

