Celle-afstamning guidet massespektrometri proteomics i udviklingen (frø) embryo
Summary
Her beskrives en massespektrometribaseret proteomisk karakterisering af cellelinjer med kendte vævsskæbner i hvirveldyrets Xenopus laevis embryo.
Abstract
Karakterisering af molekylære begivenheder, når celler giver anledning til væv og organer, rejser et potentiale for bedre at forstå normal udvikling og designe effektive midler mod sygdomme. Teknologier, der muliggør nøjagtig identifikation og kvantificering af forskellige typer og et stort antal proteiner, vil give stadig manglende information om molekylære mekanismer, der orkestrerer vævs- og organismeudvikling i rum og tid. Her præsenterer vi en massespektrometribaseret protokol, der muliggør måling af tusindvis af proteiner i identificerede cellelinjer i Xenopus laevis (frø) embryoner. Tilgangen bygger på reproducerbare celleskæbnekort og etablerede metoder til at identificere, fluorescerende mærke, spore og prøve celler og deres afkom (kloner) fra denne model af hvirveldyrs udvikling. Efter indsamling af cellulært indhold ved hjælp af mikrosampling eller isolerende celler ved dissektion eller fluorescensaktiveret cellesortering ekstraheres proteiner og behandles til bottom-up proteomisk analyse. Væskekromatografi og kapillærelektroforese anvendes til at tilvejebringe skalerbar adskillelse til proteindetektion og kvantificering med massespektrometri med høj opløsning (HRMS). Repræsentative eksempler er tilvejebragt til proteomisk karakterisering af neurale vævsceller. Celleafstamningsstyret HRMS-proteomics kan tilpasses forskellige væv og organismer. Det er tilstrækkeligt følsomt, specifikt og kvantitativt til at kigge ind i proteomets rumlige-tidsmæssige dynamik under hvirveldyrs udvikling.
Introduction
Vores forståelse af celledifferentiering og vævs og organers tilblivelse er resultatet af årtiers detaljerede målrettede screeninger af gener og deres produkter. At øge vores viden om alle biomolekylerne og deres mængder under vigtige cellulære begivenheder ville hjælpe med at optrævle molekylære mekanismer, der styrer det rumlige og tidsmæssige mønster af hvirveldyrs kropsplan. Teknologier, der muliggør molekylær amplifikation og sekventering, er nu i stand til rutinemæssigt at rapportere om et stort antal gener og transkripter, hvilket understøtter hypotesedrevne undersøgelser inden for grundlæggende biologisk og translationel forskning. For at forstå udviklingssystemer går et komplekst forhold mellem transkription og oversættelse ind for direkte analyse af flere proteiner og deres posttranslationelle modifikationer. Global proteomics ved hjælp af in vitro biologiske systemer, såsom inducerede pluripotente stamceller, begyndte at afgrænse mekanismer for vævsinduktion 1,2. I komplekse organismer, såsom hvirveldyrembryoet, afhænger udviklingen af morfogengradienter i sammenhæng med rum og tid3. Det følger heraf, at det at få viden om proteomiske ændringer, når celler differentierer sig til dannelse af specialiserede væv, såsom neurale væv, giver en nøgle til at låse op for molekylære programmer, der styrer normal og defekt udvikling og guide næste generations terapi.
Hvirveldyret sydafrikansk klofrø (Xenopus laevis) er en veletableret model inden for celle- og udviklings-, neuro- og regenerativ biologi. Sir John Gurdons Nobelpris i fysiologi eller medicin 4,5 i 2012 for opdagelsen af pluripotens af den somatiske kerne fremhævede betydningen af denne model for opdagelser i grundlæggende og translationelle studier. Xenopus-embryoner udvikler sig eksternt til moderen, hvilket letter direkte manipulation af celler, cellekloner og genekspression over forskellige udviklingsstadier. Asymmetrisk pigmentering og stereotype celledelinger gjorde det muligt at kortlægge reproducerbare skæbnekort fra 16-6 og 32-celle 7,8-stadieembryoet. For proteomics baseret på massespektrometri med høj opløsning (HRMS) omfatter yderligere fordele ved modellen relativt stor størrelse (~1 mm i diameter), hvilket giver rigeligt proteinindhold til analyse (~130 μg i embryoner i tidlig spaltningsfase, ~10 μg proteinindhold i enkeltceller i det 16-cellede embryo)9,10.
På nuværende tidspunkt er HRMS den førende foretrukne teknologi til påvisning af proteiner. Denne teknologi muliggør direkte, følsom og specifik detektion og kvantificering af flere, normalt hundreder til tusinder af forskellige proteiner11. Bottom-up proteomics af HRMS involverer en række indbyrdes forbundne trin. Efter ekstraktion fra celle/vævsprøven fordøjes proteiner med et proteolytisk enzym, såsom trypsin (bottom-up proteomics). De resulterende peptider adskilles baseret på deres forskellige fysisk-kemiske egenskaber, herunder hydrofobicitet (omvendt fase væskekromatografi, LC), nettoladning (ionbytterkromatografi), størrelse (størrelseseksklusionskromatografi) eller elektroforetisk mobilitet (kapillærelektroforese, CE). Peptider lades derefter (ioniseres), typisk ved hjælp af elektrosprayionisering (ESI), og peptidioner detekteres og sekventeres via gasfasefragmentering ved tandem HRMS. De resulterende peptiddata kortlægges til proteomet af organismen, der undersøges. Med proteinspecifik (proteotypisk) peptidionsignalintensitet, der korrelerer med koncentration, kan proteinkvantificering udføres etiketfri eller etiketbaseret (multiplexingkvantificering). HRMS proteomics giver en rig ressource af information om den molekylære tilstand af det undersøgte system, hvilket giver mulighed for generering af hypoteser og opfølgende funktionelle undersøgelser.
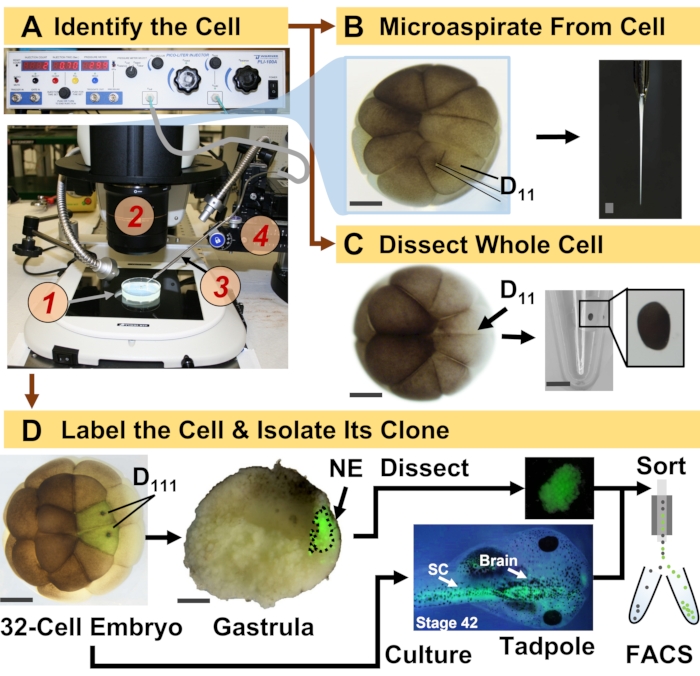
Figur 1: Spatiotemporalt skalerbar proteomics, der muliggør celleafstamningsstyret HRMS-proteomics i det udviklende (frø) embryo. (A) Visualisering af prøven (1) ved hjælp af et stereomikroskop (2) til injektion af en identificeret celle (indsats) ved hjælp af en fabrikeret mikropipette (3) under kontrol ved hjælp af et translationstrin (4). B) Subcellulær prøveudtagning af den identificerede venstre D11-celle i et 16-cellet embryo. C) Dissektion af en hel D11-celle fra et 16-cellet embryo. (D) Fluorescerende (grøn) sporing af venstre og højre D111-afkom fra et 32-cellet embryo for at styre dissektion af neurale ektoderm (NE) i gastrula (trin 10) og isolering af det nedadgående væv fra haletudsen ved hjælp af FACS. Skalastænger: 200 μm for embryoner, 1,25 mm for hætteglasset. Tallene er tilpasset med tilladelse fra referencerne 15,19,21,59. Klik her for at se en større version af denne figur.
Den protokol, der præsenteres her, muliggør HRMS-baseret kvantificering af et stort antal proteiner i identificerede celler/væv ved udvikling af X. laevis-embryoner. Tilgangen bygger på nøjagtig celleidentifikation, reproducerbare celleskæbnekort og etablerede metoder til at spore cellelinjer i denne biologiske model 6,7,8. Som vist i figur 1 studerer vi proteomer fra enkeltceller ved at anvende helcelledissektion eller kapillær mikrosampling til at aspirere cellulært indhold. Overvågning af en celles afstamning giver os mulighed for at studere proteomets rumlige temporale udvikling, når celler danner væv under gastrulation. Celleafkom markeres fluorescerende ved at injicere en fluorofor konjugeret til inert dextran eller mRNA til fluorescerende protein (fx grønt fluorescerende protein eller GFP). Det mærkede afkom isoleres på ønskede udviklingstidspunkter. Under gastrulation kan cellekloner, der er tæt grupperet, isoleres ved dissektion. Efter gastrulation kan cellekloner fordeles i embryoet på grund af migrationsbevægelser og kan isoleres fra dissocieret væv ved fluorescensaktiveret cellesortering (FACS). Proteiner i disse celler og væv måles via bottom-up proteomics ved hjælp af HPLC eller CE til separation og ESI tandem HRMS til identifikation. Celleafstamningsstyret HRMS-proteomics er skalerbar til forskellige cellestørrelser og slægter i embryoet og er specifik, følsom og kvantitativ. Gennem udvalgte eksempler vist her demonstrerer vi også, at denne protokol er skalerbar og bredt tilpasningsdygtig til forskellige typer celler og cellelinjer.
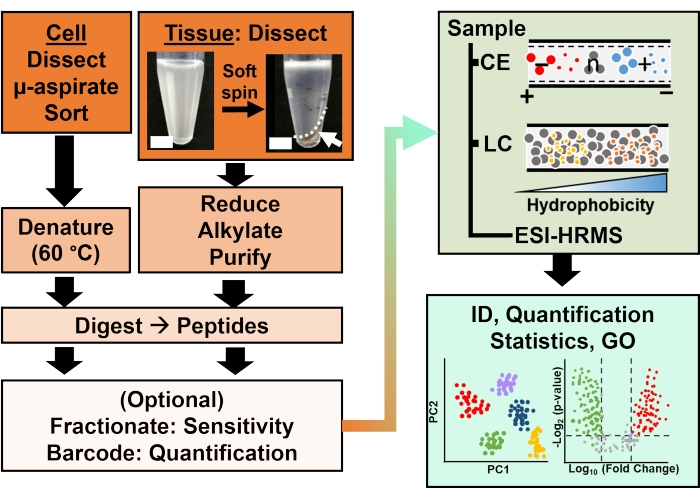
Figur 2: Den bioanalytiske arbejdsgang. Mikrodissektion og kapillær aspiration eller FACS lettede prøveudtagning af cellulært og klonalt proteinindhold. Udtømning af rigelige æggeblommeproteiner og adskillelse ved kapillær elektroforese (CE) eller nano-flow væskekromatografi (LC) forbedret identifikation (ID) følsomhed ved hjælp af elektrospray ionisering (ESI) massespektrometri med høj opløsning (HRMS). Kvantificering afslørede dysregulering og leverede ny information til hypotesedrevne undersøgelser i forbindelse med information tilgængelig fra genontologi (GO). Tallene er tilpasset med tilladelse fra reference15. Klik her for at se en større version af denne figur.
Protocol
Representative Results
Discussion
Denne protokol muliggør karakterisering af proteinekspression i identificerede cellelinjer i embryoner af Xenopus-arten . Metoden stammer fra HRMS og kombinerer udsøgt specificitet i molekylær identifikation, evne til multiproteindetektion uden molekylære sonder (normalt hundreder til tusinder af forskellige proteiner) og en evne til kvantificering. Tilpasningsevne til klassiske værktøjer og arbejdsgange inden for celle- og udviklingsmæssig (neuro)biologi udvider HRMS-proteomics til spændende anvendelser…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vi er taknemmelige for Jie Li (University of Maryland, College Park) for værdifulde diskussioner om embryonal dissociation og FACS. Vi takker Vi M. Quach og Camille Lombard-Banek for hjælp med prøveforberedelse og dataindsamling i tidligere undersøgelser, der eksemplificerer de proteomiske anvendelser, der er fremhævet i denne protokol. Dele af dette arbejde blev støttet af National Science Foundation under tildelingsnummer IOS-1832968 CAREER (til P.N.), National Institutes of Health under tildelingsnummer R35GM124755 (til P.N.), University of Maryland-National Cancer Institute Partnership Program (til P.N.) og COSMOS Club Foundation forskningspriser (til ABB og LRP).
Materials
| Acetonitrile (LC-MS-grade) | Fisher Scientific | A955 | |
| Agarose | ThermoFisher Scientific | R0492 | |
| Ammonium bicarbonate | Fisher Scientific | A643-500 | |
| Analytical Column | Thermo Scientific | 164941 | |
| Analytical microbalance | Mettler-Toledo | XSE105DU | |
| Automatic peptide fractionation platform | Agilent | 1260 Infinity II | |
| Borosilicate Capillaries | Sutter Instruments Co. | B100-50-10 | |
| Borosilicate Capillaries (for making Emmitters) | Sutter Instruments | B100-75-10 | |
| C18 spin columns (for desalting) | ThermoFisher Scientific | 89870 | |
| Camera ro monitor electrospray | Edmund Optics Inc. | EO-2018C | |
| Combretastatin A4 | Millipore Sigma | C7744 | |
| Commercial CESI system | AB SCIEX | CESI | |
| (Cyclohexylamino)-1-propanesulfonic acid (CAPS) | VWR | 97061-492 | |
| Cytochalasin D | Millipore Sigma | C8273 | |
| Dextran, Alexa Fluor 488; 10,000 MW, Anionic, Fixable | ThermoFisher Scientific | D22910 | |
| Diothiothreitol | Fisher Scientific | FERR0861 | |
| Dumont #5 Forceps | Fine Science Tools | 11252-30 | |
| EDTA | Fisher Scientific | AAJ62786AP | |
| Epifluorescence light source | Lumencore | AURA III | |
| Eppendorf LoBing microcentrifuge tubes: protein | Fisher Scientific | 13-698-793 | |
| Formic acid (LC-MS-grade) | Fisher Scientific | A117-50 | |
| Freezer (-20 °C) | Fisher Scientific | 97-926-1 | |
| Freezer (-80 °C) | Thermo Scientific | TSX40086A | |
| Fused silica capillary | Molex | 1088150596 | |
| Heat Block | Benchmark | BSH300 | |
| High pressure liquid Chromatography System | ThermoFisher Scientific | Dionex Ultimate 3000 RSLC nanosystem | |
| High voltage power supply | Spellman | CZE1000R | |
| High-resolution Mass Spectrometer | ThermoFisher Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | |
| HPLC caps | Thermo Scientific | C4013-40A | |
| HPLC Vials | Thermo Scientific | C4013-11 | |
| Illuminator e.g. Goosenecks | Nikon | C-FLED2 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide | Fisher Scientific | AC122275000 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456-4 | |
| Microcapillary puller | Suttor Instruments | P-2000 | |
| Microinjector | Warner Instrument, Handem, CT | PLI-100A | |
| Micropippette puller | Sutter Instruments Co. | P-1000 | |
| MS data analysis software, commercial | ProteomeDiscoverer | ||
| MS data analysis software, opensource | MaxQuant | ||
| non-idet 40 substitute | Millipore Sigma | 11754599001 | |
| Petri dish 60 mm and 80 mm | Fisher Scientific | S08184 | |
| Pierce 10 µL bed Zip-tips (for desalting) | ThermoFisher Scientific | 87782 | |
| Pierce bicinchoninic acid protein assay kit | ThermoFisher Scientific | 23225 | |
| Pierce quantitative colorimetric peptide assay | ThermoFisher Scientific | 23275 | |
| Pierce Trypsin Protease (MS Grade) | Fisher Scientific | PI90058 | |
| Protein LoBind vials | Eppendorf | 0030108434 , 0030108442 |
|
| Refrigerated Centrifuge | Eppendorf | 5430R | |
| Refrigerated Incubator | Thermo Scientific | PR505755R/3721 | |
| sodium isethionate | Millipore Sigma | 220078 | |
| sodium pyrophosphate | Sigma Aldrich | 221368-100G | |
| Stainless steel BGE vial | Custom-Built | ||
| Stainless steel sample vials | Custom-Built | ||
| Stereomicroscope (objective 10x) | Nikon | SMZ 1270, SZX18 | |
| Sucrose | VWR | 97063-790 | |
| Syringe pumps (2) | Harvard Apparatus | 704506 | |
| Syringes (gas-tight): 500–1000 µL | Hamilton | 1750TTL | |
| Transfer pipettes (Plastic, disposable) | Fisher Scientific | 13-711-7M | |
| Trap Column | Thermo Scientific | 164750 | |
| Tris-HCl (1 M solution) | Fisher Scientific | AAJ22638AP | |
| Vacuum concentrator capable of operation at 4–10 °C | Labconco | 7310022 | |
| Vortex-mixer | Benchmark | BS-VM-1000 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| XYZ translation stage | Thorlabs | PT3 | |
| XYZ translation stage | Custom-Built |
References
- Shoemaker, L. D., Kornblum, H. I. Neural Stem Cells (NSCs) and Proteomics. Molecular & Cellular Proteomics. 15 (2), 344-354 (2016).
- Cervenka, J., et al. Proteomic characterization of human neural stem cells and their secretome during in vitro differentiation. Frontiers in Cellular Neuroscience. 14, 612560 (2021).
- Christian, J. L. Morphogen gradients in development: From form to function. Wiley Interdisciplinary Reviews. Developmental Biology. 1 (1), 3-15 (2012).
- Gurdon, J. B., Elsdale, T. R., M, F. Sexually mature individuals of Xenopus laevis from the transplantation of single somatic nuclei. Nature. 182, 64-65 (1958).
- Harland, R. M., Grainger, R. M. Xenopus research: metamorphosed by genetics and genomics. Trends in Genetics. 27 (12), 507-515 (2011).
- Moody, S. A. Fates of the blastomeres of the 16-cell stage Xenopus embryo. Developmental Biology. 119 (2), 560-578 (1987).
- Moody, S. A. Fates of the blastomeres of the 32-cell stage Xenopus embryo. Developmental Biology. 122 (2), 300-319 (1987).
- Dale, L., Slack, J. M. W. Fate map for the 32-cell stage of Xenopus laevis. Development. 99 (4), 527-551 (1987).
- Sun, L. L., et al. Single cell proteomics using frog (Xenopus laevis) blastomeres isolated from early stage embryos, which form a geometric progression in protein content. Analytical Chemistry. 88 (13), 6653-6657 (2016).
- Lombard-Banek, C., Moody, S. A., Nemes, P. Single-cell mass spectrometry for discovery proteomics: quantifying translational cell heterogeneity in the 16-cell frog (Xenopus) embryo. Angewandte Chemie-International Edition. 55 (7), 2454-2458 (2016).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early development of Xenopus laevis: A laboratory manual. , (2000).
- Briggs, J. A., et al. The dynamics of gene expression in vertebrate embryogenesis at single-cell resolution. Science. 360 (6392), (2018).
- Gupta, M., Sonnett, M., Ryazanova, L., Presler, M., Wuhr, M., Vleminckx, K. Quantitative proteomics of xenopus embryos I, sample preparation. Xenopus. Methods in Molecular Biology. 1865, 175-194 (2018).
- Baxi, A. B., Lombard-Banek, C., Moody, S. A., Nemes, P. Proteomic characterization of the neural ectoderm fated cell clones in the Xenopus laevis embryo by high-resolution mass spectrometry. ACS Chemical Neuroscience. 9 (8), 2064-2073 (2018).
- Moody, S. A. Cell lineage analysis in Xenopus embryos. Methods in Molecular Biology. 135, 331-347 (2000).
- Sater, A. K., Moody, S. A. Using Xenopus to understand human diseases and developmental disorders. Genesis. 55 (1-2), 1-14 (2017).
- Lombard-Banek, C., Choi, S. B., Nemes, P., Allbritton, N. L., Kovarik, M. L. . Enzyme Activity in Single Cells. Methods in Enzymology. 628, 263-292 (2019).
- Lombard-Banek, C., Moody, S. A., Nemes, P. High-sensitivity mass spectrometry for probing gene translation in single embryonic cells in the early frog (Xenopus) embryo. Frontiers in Cell and Developmental Biology. 4, 11 (2016).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. Microprobe capillary electrophoresis mass spectrometry for single-cell metabolomics in live frog (Xenopus laevis) embryos. Journal of Visualized Experiments: JoVE. (130), e56956 (2017).
- Lombard-Banek, C., Moody, S. A., Manzin, M. C., Nemes, P. Microsampling capillary electrophoresis mass spectrometry enables single-cell proteomics in complex tissues: developing cell clones in live Xenopus laevis and zebrafish embryos. Analytical Chemistry. 91 (7), 4797-4805 (2019).
- Klein, S. L. The first cleavage furrow demarcates the dorsal-ventral axis in Xenopus embryos. Developmental Biology. 120 (1), 299-304 (1987).
- Karimi, K., et al. Xenbase: a genomic, epigenomic and transcriptomic model organism database. Nucleic Acids Research. 46 (1), 861-868 (2018).
- Kakebeen, A. D., Chitsazan, A. D., Wills, A. E. Tissue disaggregation and isolation of specific cell types from transgenic Xenopus appendages for transcriptional analysis by FACS. Developmental Dynamics. 250 (9), 1381-1392 (2021).
- Garcia, B. A. What does the future hold for top down mass spectrometry. Journal of the American Society for Mass Spectrometry. 21 (2), 193-202 (2010).
- Toby, T. K., Fornelli, L., Kelleher, N. L. Progress in top-down proteomics and the analysis of proteoforms. Annual Review of Analytical Chemistry. (Palo Alto Calif). 9 (1), 499-519 (2016).
- Zhang, Z. B., Dubiak, K. M., Huber, P. W., Dovichi, N. J. Miniaturized filter-aided sample preparation (MICRO-FASP) method for high throughput, ultrasensitive proteomics sample preparation reveals proteome asymmetry in Xenopus laevis Embryos. Analytical Chemistry. 92 (7), 5554-5560 (2020).
- Wisniewski, J. R., Becher, D. . Microbial Proteomics: Methods and Protocols.Methods in Molecular Biology. 1841, 3-10 (2018).
- Hughes, C. S., et al. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9, 882 (2018).
- Wessel, D., Flugge, U. I. A method for the quantitative recovery of protein in dilute-solution in the presence of detergents and lipids. Analytical Biochemistry. 138 (1), 141-143 (1984).
- Jiang, L., He, L., Fountoulakis, M. Comparison of protein precipitation methods for sample preparation prior to proteomic analysis. Journal of Chromatography A. 1023 (2), 317-320 (2004).
- Hildonen, S., Halvorsen, T. G., Reubsaet, L. Why less is more when generating tryptic peptides in bottom-up proteomics. Proteomics. 14 (17-18), 2031-2041 (2014).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19, 161 (2018).
- Drouin, N., et al. Capillary electrophoresis-mass spectrometry at trial by metabo-ring: effective electrophoretic mobility for reproducible and robust compound annotation. Analytical Chemistry. 92 (20), 14103-14112 (2020).
- Sun, L. L., Zhu, G. J., Zhang, Z. B., Mou, S., Dovichi, N. J. Third-generation electrokinetically pumped sheath-flow nanospray interface with improved stability and sensitivity for automated capillary zone electrophoresis-mass spectrometry analysis of complex proteome digests. Journal of Proteome Research. 14 (5), 2312-2321 (2015).
- DeLaney, K., Sauer, C. S., Vu, N. Q., Li, L. J. Recent advances and new perspectives in capillary electrophoresis-mass spectrometry for single cell "omics". Molecules. 24 (1), 21 (2019).
- Nemes, P., Rubakhin, S. S., Aerts, J. T., Sweedler, J. V. Qualitative and quantitative metabolomic investigation of single neurons by capillary electrophoresis electrospray ionization mass spectrometry. Nature Protocols. 8 (4), 783-799 (2013).
- Choi, S. B., Zamarbide, M., Manzini, M. C., Nemes, P. Tapered-tip capillary electrophoresis nano-electrospray ionization mass spectrometry for ultrasensitive proteomics: the mouse cortex. Journal of the American Society for Mass Spectrometry. 28 (4), 597-607 (2017).
- Pino, L. K., Rose, J., O’Broin, A., Shah, S., Schilling, B. Emerging mass spectrometry-based proteomics methodologies for novel biomedical applications. Biochemical Society Transactions. 48 (5), 1953-1966 (2020).
- Chen, C., Hou, J., Tanner, J. J., Cheng, J. L. Bioinformatics methods for mass spectrometry-based proteomics data analysis. International Journal of Molecular Sciences. 21 (8), 25 (2020).
- Peshkin, L., et al. On the relationship of protein and mRNA dynamics in vertebrate embryonic development. Developmental Cell. 35 (3), 383-394 (2015).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Gygi, S. P., et al. Quantitative analysis of complex protein mixtures using isotope-coded affinity tags. Nature Biotechnology. 17 (10), 994-999 (1999).
- Thompson, A., et al. Tandem mass tags: A novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75 (8), 1895-1904 (2003).
- Mi, H. Y., et al. PANTHER version 16: a revised family classification, tree-based classification tool, enhancer regions and extensive api. Nucleic Acids Research. 49, 394-403 (2021).
- Schmidt, E., et al. . On the Move Federated Workshops. , 710-719 (2006).
- Deutsch, E. W., et al. Trans-Proteomic pipeline, a standardized data processing pipeline for large-scale reproducible proteomics informatics. Proteomics Clinical Applications. 9 (7-8), 745-754 (2015).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Demsar, J., et al. Orange: Data mining toolbox in Python. Journal of Machine Learning Research. 14, 2349-2353 (2013).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. Journal of Proteome Research. 8 (5), 2144-2156 (2009).
- Jensen, L. J., et al. STRING 8 – a global view on proteins and their functional interactions in 630 organisms. Nucleic Acids Research. 37, 412-416 (2009).
- Schweppe, D. K., Huttlin, E. L., Harper, J. W., Gygi, S. P. BioPlex display: an interactive suite for large-scale AP-MS protein-protein interaction data. Journal of Proteome Research. 17 (1), 722-726 (2018).
- Hornbeck, P. V., et al. PhosphoSitePlus, 2014: mutations, PTMs and recalibrations. Nucleic Acids Research. 43, 512-520 (2015).
- Letunic, I., Khedkar, S., Bork, P. SMART: recent updates, new developments and status in 2020. Nucleic Acids Research. 49, 458-460 (2021).
- Lombard-Banek, C., et al. In vivo subcellular mass spectrometry enables proteo-metabolomic single-cell systems biology in a chordate embryo developing to a normally behaving tadpole (X. laevis). Angewandte Chemie-International Edition. 60 (23), 12852-12858 (2021).
- Lombard-Banek, C., Reddy, S., Moody, S. A., Nemes, P. Label-free quantification of proteins in single embryonic cells with neural fate in the cleavage-stage frog (Xenopus laevis) embryo using capillary electrophoresis electrospray ionization high-resolution mass spectrometry (CE-ESI-HRMS). Molecular & Cellular Proteomics. 15 (8), 2756-2768 (2016).
- Saha-Shah, A., et al. Single cell proteomics by data-independent acquisition to study embryonic asymmetry in Xenopus laevis. Analytical Chemistry. 91 (14), 8891-8899 (2019).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. In situ microprobe single-cell capillary electrophoresis mass spectrometry: metabolic reorganization in single differentiating cells in the live vertebrate (Xenopus laevis) embryo. Analytical Chemistry. 89 (13), 7069-7076 (2017).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).

