Proteómica de espectrometría de masas guiada por linaje celular en el embrión en desarrollo (rana)
Summary
Aquí describimos una caracterización proteómica basada en espectrometría de masas de linajes celulares con destinos tisulares conocidos en el embrión vertebrado de Xenopus laevis .
Abstract
La caracterización de eventos moleculares a medida que las células dan lugar a tejidos y órganos plantea un potencial para comprender mejor el desarrollo normal y diseñar remedios eficientes para las enfermedades. Las tecnologías que permiten la identificación y cuantificación precisas de diversos tipos y un gran número de proteínas proporcionarían información aún faltante sobre los mecanismos moleculares que orquestan el desarrollo de tejidos y organismos en el espacio y el tiempo. Aquí, presentamos un protocolo basado en espectrometría de masas que permite la medición de miles de proteínas en linajes celulares identificados en embriones de Xenopus laevis (rana). El enfoque se basa en mapas reproducibles de destino celular y métodos establecidos para identificar, etiquetar fluorescentemente, rastrear y muestrear células y su progenie (clones) a partir de este modelo de desarrollo de vertebrados. Después de recolectar el contenido celular utilizando micromuestreo o aislar células por disección o clasificación celular activada por fluorescencia, las proteínas se extraen y procesan para el análisis proteómico ascendente. La cromatografía líquida y la electroforesis capilar se utilizan para proporcionar una separación escalable para la detección y cuantificación de proteínas con espectrometría de masas de alta resolución (HRMS). Se proporcionan ejemplos representativos para la caracterización proteómica de células destinadas al tejido neural. La proteómica HRMS guiada por linaje celular es adaptable a diferentes tejidos y organismos. Es lo suficientemente sensible, específico y cuantitativo para observar la dinámica espacio-temporal del proteoma durante el desarrollo de los vertebrados.
Introduction
Nuestra comprensión de la diferenciación celular y la génesis de tejidos y órganos es el resultado de décadas de elaboradas pantallas específicas de genes y sus productos. Aumentar nuestro conocimiento de todas las biomoléculas y sus cantidades durante eventos celulares importantes ayudaría a desentrañar los mecanismos moleculares que controlan el patrón espacial y temporal del plan corporal de los vertebrados. Las tecnologías que permiten la amplificación molecular y la secuenciación ahora pueden informar rutinariamente sobre un gran número de genes y transcripciones, apoyando estudios basados en hipótesis en investigación biológica básica y traslacional. Para comprender los sistemas en desarrollo, una relación compleja entre la transcripción y la traducción aboga por el análisis directo de múltiples proteínas y sus modificaciones postraduccionales. La proteómica global utilizando sistemas biológicos in vitro, como células madre pluripotentes inducidas, comenzó a delinear mecanismos de inducción tisular 1,2. En organismos complejos, como el embrión vertebrado, el desarrollo se basa en gradientes morfógenos en el contexto del espacio y el tiempo3. De ello se deduce que obtener conocimiento de los cambios proteómicos a medida que las células se diferencian para formar tejidos especializados, como los tejidos neurales, ofrece una clave para desbloquear programas moleculares que controlan el desarrollo normal y defectuoso y guiar la terapéutica de próxima generación.
La rana vertebrada sudafricana (Xenopus laevis) es un modelo bien establecido en biología celular y del desarrollo, neuro y regenerativa. El Premio Nobel de Fisiología o Medicina 2012 de Sir John Gurdon por el descubrimiento de la pluripotencia del núcleo somático destacó la importancia de este modelo para los descubrimientos en estudios básicos y traslacionales. Los embriones de Xenopus se desarrollan externamente a la madre, facilitando así la manipulación directa de células, clones celulares y expresión génica en varias etapas de desarrollo. La pigmentación asimétrica y las divisiones celulares estereotipadas permitieron el trazado de mapas de destino reproducibles del embrión de 7,8 células de 16-6 y 32 células. Para la proteómica basada en espectrometría de masas de alta resolución (HRMS), las ventajas adicionales del modelo incluyen un tamaño relativamente grande (~ 1 mm de diámetro), que produce un contenido abundante de proteínas para el análisis (~ 130 μg en embriones en etapa de escisión temprana, ~ 10 μg de contenido de proteína en células individuales del embrión de 16 células)9,10.
En la actualidad, HRMS es la tecnología líder de elección para detectar proteínas. Esta tecnología permite la detección y cuantificación directa, sensible y específica de múltiples, generalmente cientos a miles de proteínas diferentes11. La proteómica ascendente de HRMS implica una serie de pasos interconectados. Después de la extracción de la muestra de célula/tejido, las proteínas se digieren con una enzima proteolítica, como la tripsina (proteómica ascendente). Los péptidos resultantes se separan en función de sus diferentes propiedades fisicoquímicas, incluida la hidrofobicidad (cromatografía líquida de fase inversa, LC), la carga neta (cromatografía de intercambio iónico), el tamaño (cromatografía de exclusión de tamaño) o la movilidad electroforética (electroforesis capilar, CE). Los péptidos se cargan (ionizan), típicamente usando ionización por electrospray (ESI), y los iones peptídicos se detectan y secuencian a través de la fragmentación en fase gaseosa por HRMS en tándem. Los datos peptídicos resultantes se asignan al proteoma del organismo que se está estudiando. Con la intensidad de la señal de iones peptídicos específicos de proteínas (proteotípicos) que se correlacionan con la concentración, la cuantificación de proteínas se puede realizar sin etiqueta o basada en etiquetas (cuantificación de multiplexación). La proteómica HRMS proporciona un rico recurso de información sobre el estado molecular del sistema en estudio, lo que permite la generación de hipótesis y estudios funcionales de seguimiento.
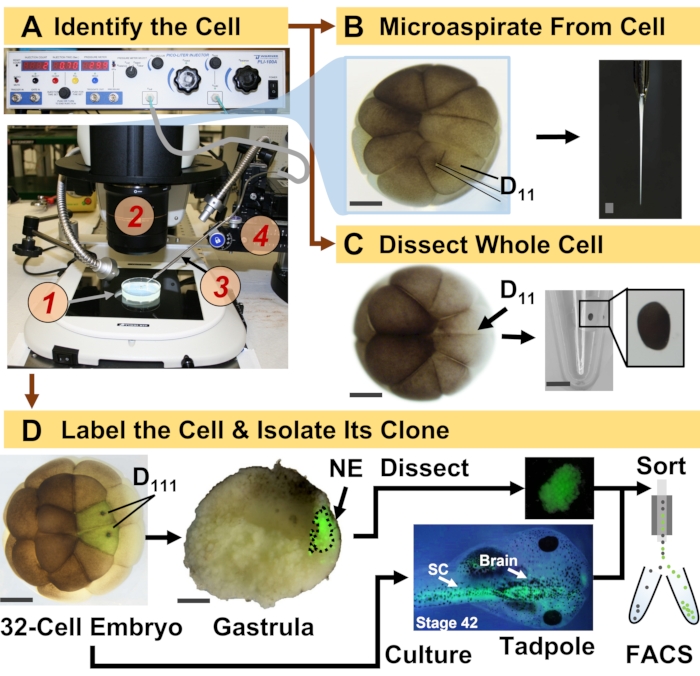
Figura 1: Proteómica escalable espaciotemporalmente que permite la proteómica HRMS guiada por linaje celular en el embrión en desarrollo (rana). (A) Visualización de la muestra (1) utilizando un microscopio estereoscópico (2) para la inyección de una célula identificada (recuadro), utilizando una micropipeta fabricada (3) bajo control por una etapa de traslación (4). (B) Muestreo subcelular de la célula D11 izquierda identificada en un embrión de 16 células. (C) Disección de una célula D11 completa de un embrión de 16 células. (D) Trazado fluorescente (verde) de las progenies D111 izquierda y derecha de un embrión de 32 células para guiar la disección del ectodermo neural (NE) en la gástrula (etapa 10) y el aislamiento del tejido descendente del renacuajo utilizando FACS. Barras de escala: 200 μm para embriones, 1,25 mm para el vial. Las figuras fueron adaptadas con permiso de las referencias 15,19,21,59. Haga clic aquí para ver una versión más grande de esta figura.
El protocolo presentado aquí permite la cuantificación basada en HRMS de un gran número de proteínas en células / tejidos identificados en embriones de X. laevis en desarrollo. El enfoque se basa en la identificación precisa de células, mapas de destino celular reproducibles y metodologías establecidas para rastrear linajes celulares en este modelo biológico 6,7,8. Como se muestra en la Figura 1, estudiamos proteomas de células individuales empleando disección de células enteras o micromuestreo capilar para aspirar el contenido celular. El monitoreo del linaje de una célula nos permite estudiar la evolución espacio-temporal del proteoma a medida que las células forman tejidos durante la gastrulación. La progenie celular se marca fluorescentemente inyectando un fluoróforo conjugado con dextrano inerte o ARNm para la proteína fluorescente (por ejemplo, proteína fluorescente verde o GFP). La progenie etiquetada se aísla en los puntos de tiempo de desarrollo deseados. Durante la gastrulación, los clones celulares que están estrechamente agrupados pueden aislarse por disección. Después de la gastrulación, los clones celulares pueden distribuirse dentro del embrión debido a movimientos migratorios y pueden aislarse de tejidos disociados mediante clasificación celular activada por fluorescencia (FACS). Las proteínas en estas células y tejidos se miden a través de la proteómica ascendente empleando HPLC o CE para la separación y ESI tándem HRMS para la identificación. La proteómica HRMS guiada por linaje celular es escalable a diferentes tamaños y linajes celulares dentro del embrión y es específica, sensible y cuantitativa. A través de ejemplos seleccionados que se muestran aquí, también demostramos que este protocolo es escalable y ampliamente adaptable a diferentes tipos de células y linajes celulares.
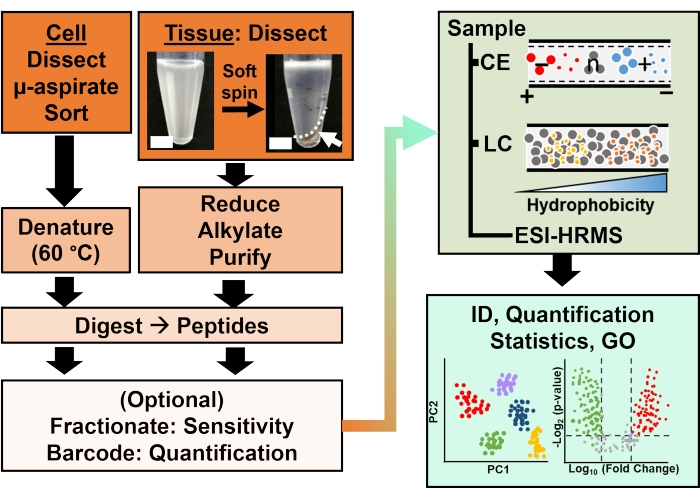
Figura 2: El flujo de trabajo bioanalítico. La microdisección y la aspiración capilar, o FACS, facilitaron el muestreo del contenido de proteínas celulares y clonales. Agotamiento de abundantes proteínas vitelinas y separación por electroforesis capilar (CE) o cromatografía líquida de nanoflujo (LC) mejora la sensibilidad de identificación (ID) utilizando espectrometría de masas de alta resolución (HRMS) por ionización por electrospray (ESI). La cuantificación reveló desregulación, proporcionando nueva información para estudios basados en hipótesis junto con la información disponible de la ontología génica (GO). Las figuras fueron adaptadas con permiso de la referencia15. Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
Este protocolo permite la caracterización de la expresión de proteínas en linajes celulares identificados en embriones de la especie Xenopus. Derivada de HRMS, la metodología combina una especificidad exquisita en la identificación molecular, la capacidad de detección de múltiples proteínas sin sondas moleculares (generalmente cientos a miles de proteínas diferentes) y la capacidad de cuantificación. La adaptabilidad a las herramientas y flujos de trabajo clásicos en biología celular y del desarrollo…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Agradecemos a Jie Li (Universidad de Maryland, College Park) por sus valiosos debates sobre la disociación embrionaria y el sistema de control de los bienes sobre el terreno. Agradecemos a Vi M. Quach y Camille Lombard-Banek por su ayuda con la preparación de muestras y la recopilación de datos en estudios previos que ejemplifican las aplicaciones proteómicas que se destacan en este protocolo. Partes de este trabajo fueron apoyadas por la National Science Foundation bajo el número de premio IOS-1832968 CAREER (a P.N.), los Institutos Nacionales de Salud bajo el número de premio R35GM124755 (a P.N.), el Programa de Asociación de la Universidad de Maryland-Instituto Nacional del Cáncer (a P.N.) y los premios de investigación de la Fundación COSMOS Club (a A.B.B. y L.R.P.).
Materials
| Acetonitrile (LC-MS-grade) | Fisher Scientific | A955 | |
| Agarose | ThermoFisher Scientific | R0492 | |
| Ammonium bicarbonate | Fisher Scientific | A643-500 | |
| Analytical Column | Thermo Scientific | 164941 | |
| Analytical microbalance | Mettler-Toledo | XSE105DU | |
| Automatic peptide fractionation platform | Agilent | 1260 Infinity II | |
| Borosilicate Capillaries | Sutter Instruments Co. | B100-50-10 | |
| Borosilicate Capillaries (for making Emmitters) | Sutter Instruments | B100-75-10 | |
| C18 spin columns (for desalting) | ThermoFisher Scientific | 89870 | |
| Camera ro monitor electrospray | Edmund Optics Inc. | EO-2018C | |
| Combretastatin A4 | Millipore Sigma | C7744 | |
| Commercial CESI system | AB SCIEX | CESI | |
| (Cyclohexylamino)-1-propanesulfonic acid (CAPS) | VWR | 97061-492 | |
| Cytochalasin D | Millipore Sigma | C8273 | |
| Dextran, Alexa Fluor 488; 10,000 MW, Anionic, Fixable | ThermoFisher Scientific | D22910 | |
| Diothiothreitol | Fisher Scientific | FERR0861 | |
| Dumont #5 Forceps | Fine Science Tools | 11252-30 | |
| EDTA | Fisher Scientific | AAJ62786AP | |
| Epifluorescence light source | Lumencore | AURA III | |
| Eppendorf LoBing microcentrifuge tubes: protein | Fisher Scientific | 13-698-793 | |
| Formic acid (LC-MS-grade) | Fisher Scientific | A117-50 | |
| Freezer (-20 °C) | Fisher Scientific | 97-926-1 | |
| Freezer (-80 °C) | Thermo Scientific | TSX40086A | |
| Fused silica capillary | Molex | 1088150596 | |
| Heat Block | Benchmark | BSH300 | |
| High pressure liquid Chromatography System | ThermoFisher Scientific | Dionex Ultimate 3000 RSLC nanosystem | |
| High voltage power supply | Spellman | CZE1000R | |
| High-resolution Mass Spectrometer | ThermoFisher Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | |
| HPLC caps | Thermo Scientific | C4013-40A | |
| HPLC Vials | Thermo Scientific | C4013-11 | |
| Illuminator e.g. Goosenecks | Nikon | C-FLED2 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide | Fisher Scientific | AC122275000 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456-4 | |
| Microcapillary puller | Suttor Instruments | P-2000 | |
| Microinjector | Warner Instrument, Handem, CT | PLI-100A | |
| Micropippette puller | Sutter Instruments Co. | P-1000 | |
| MS data analysis software, commercial | ProteomeDiscoverer | ||
| MS data analysis software, opensource | MaxQuant | ||
| non-idet 40 substitute | Millipore Sigma | 11754599001 | |
| Petri dish 60 mm and 80 mm | Fisher Scientific | S08184 | |
| Pierce 10 µL bed Zip-tips (for desalting) | ThermoFisher Scientific | 87782 | |
| Pierce bicinchoninic acid protein assay kit | ThermoFisher Scientific | 23225 | |
| Pierce quantitative colorimetric peptide assay | ThermoFisher Scientific | 23275 | |
| Pierce Trypsin Protease (MS Grade) | Fisher Scientific | PI90058 | |
| Protein LoBind vials | Eppendorf | 0030108434 , 0030108442 |
|
| Refrigerated Centrifuge | Eppendorf | 5430R | |
| Refrigerated Incubator | Thermo Scientific | PR505755R/3721 | |
| sodium isethionate | Millipore Sigma | 220078 | |
| sodium pyrophosphate | Sigma Aldrich | 221368-100G | |
| Stainless steel BGE vial | Custom-Built | ||
| Stainless steel sample vials | Custom-Built | ||
| Stereomicroscope (objective 10x) | Nikon | SMZ 1270, SZX18 | |
| Sucrose | VWR | 97063-790 | |
| Syringe pumps (2) | Harvard Apparatus | 704506 | |
| Syringes (gas-tight): 500–1000 µL | Hamilton | 1750TTL | |
| Transfer pipettes (Plastic, disposable) | Fisher Scientific | 13-711-7M | |
| Trap Column | Thermo Scientific | 164750 | |
| Tris-HCl (1 M solution) | Fisher Scientific | AAJ22638AP | |
| Vacuum concentrator capable of operation at 4–10 °C | Labconco | 7310022 | |
| Vortex-mixer | Benchmark | BS-VM-1000 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| XYZ translation stage | Thorlabs | PT3 | |
| XYZ translation stage | Custom-Built |
References
- Shoemaker, L. D., Kornblum, H. I. Neural Stem Cells (NSCs) and Proteomics. Molecular & Cellular Proteomics. 15 (2), 344-354 (2016).
- Cervenka, J., et al. Proteomic characterization of human neural stem cells and their secretome during in vitro differentiation. Frontiers in Cellular Neuroscience. 14, 612560 (2021).
- Christian, J. L. Morphogen gradients in development: From form to function. Wiley Interdisciplinary Reviews. Developmental Biology. 1 (1), 3-15 (2012).
- Gurdon, J. B., Elsdale, T. R., M, F. Sexually mature individuals of Xenopus laevis from the transplantation of single somatic nuclei. Nature. 182, 64-65 (1958).
- Harland, R. M., Grainger, R. M. Xenopus research: metamorphosed by genetics and genomics. Trends in Genetics. 27 (12), 507-515 (2011).
- Moody, S. A. Fates of the blastomeres of the 16-cell stage Xenopus embryo. Developmental Biology. 119 (2), 560-578 (1987).
- Moody, S. A. Fates of the blastomeres of the 32-cell stage Xenopus embryo. Developmental Biology. 122 (2), 300-319 (1987).
- Dale, L., Slack, J. M. W. Fate map for the 32-cell stage of Xenopus laevis. Development. 99 (4), 527-551 (1987).
- Sun, L. L., et al. Single cell proteomics using frog (Xenopus laevis) blastomeres isolated from early stage embryos, which form a geometric progression in protein content. Analytical Chemistry. 88 (13), 6653-6657 (2016).
- Lombard-Banek, C., Moody, S. A., Nemes, P. Single-cell mass spectrometry for discovery proteomics: quantifying translational cell heterogeneity in the 16-cell frog (Xenopus) embryo. Angewandte Chemie-International Edition. 55 (7), 2454-2458 (2016).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early development of Xenopus laevis: A laboratory manual. , (2000).
- Briggs, J. A., et al. The dynamics of gene expression in vertebrate embryogenesis at single-cell resolution. Science. 360 (6392), (2018).
- Gupta, M., Sonnett, M., Ryazanova, L., Presler, M., Wuhr, M., Vleminckx, K. Quantitative proteomics of xenopus embryos I, sample preparation. Xenopus. Methods in Molecular Biology. 1865, 175-194 (2018).
- Baxi, A. B., Lombard-Banek, C., Moody, S. A., Nemes, P. Proteomic characterization of the neural ectoderm fated cell clones in the Xenopus laevis embryo by high-resolution mass spectrometry. ACS Chemical Neuroscience. 9 (8), 2064-2073 (2018).
- Moody, S. A. Cell lineage analysis in Xenopus embryos. Methods in Molecular Biology. 135, 331-347 (2000).
- Sater, A. K., Moody, S. A. Using Xenopus to understand human diseases and developmental disorders. Genesis. 55 (1-2), 1-14 (2017).
- Lombard-Banek, C., Choi, S. B., Nemes, P., Allbritton, N. L., Kovarik, M. L. . Enzyme Activity in Single Cells. Methods in Enzymology. 628, 263-292 (2019).
- Lombard-Banek, C., Moody, S. A., Nemes, P. High-sensitivity mass spectrometry for probing gene translation in single embryonic cells in the early frog (Xenopus) embryo. Frontiers in Cell and Developmental Biology. 4, 11 (2016).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. Microprobe capillary electrophoresis mass spectrometry for single-cell metabolomics in live frog (Xenopus laevis) embryos. Journal of Visualized Experiments: JoVE. (130), e56956 (2017).
- Lombard-Banek, C., Moody, S. A., Manzin, M. C., Nemes, P. Microsampling capillary electrophoresis mass spectrometry enables single-cell proteomics in complex tissues: developing cell clones in live Xenopus laevis and zebrafish embryos. Analytical Chemistry. 91 (7), 4797-4805 (2019).
- Klein, S. L. The first cleavage furrow demarcates the dorsal-ventral axis in Xenopus embryos. Developmental Biology. 120 (1), 299-304 (1987).
- Karimi, K., et al. Xenbase: a genomic, epigenomic and transcriptomic model organism database. Nucleic Acids Research. 46 (1), 861-868 (2018).
- Kakebeen, A. D., Chitsazan, A. D., Wills, A. E. Tissue disaggregation and isolation of specific cell types from transgenic Xenopus appendages for transcriptional analysis by FACS. Developmental Dynamics. 250 (9), 1381-1392 (2021).
- Garcia, B. A. What does the future hold for top down mass spectrometry. Journal of the American Society for Mass Spectrometry. 21 (2), 193-202 (2010).
- Toby, T. K., Fornelli, L., Kelleher, N. L. Progress in top-down proteomics and the analysis of proteoforms. Annual Review of Analytical Chemistry. (Palo Alto Calif). 9 (1), 499-519 (2016).
- Zhang, Z. B., Dubiak, K. M., Huber, P. W., Dovichi, N. J. Miniaturized filter-aided sample preparation (MICRO-FASP) method for high throughput, ultrasensitive proteomics sample preparation reveals proteome asymmetry in Xenopus laevis Embryos. Analytical Chemistry. 92 (7), 5554-5560 (2020).
- Wisniewski, J. R., Becher, D. . Microbial Proteomics: Methods and Protocols.Methods in Molecular Biology. 1841, 3-10 (2018).
- Hughes, C. S., et al. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9, 882 (2018).
- Wessel, D., Flugge, U. I. A method for the quantitative recovery of protein in dilute-solution in the presence of detergents and lipids. Analytical Biochemistry. 138 (1), 141-143 (1984).
- Jiang, L., He, L., Fountoulakis, M. Comparison of protein precipitation methods for sample preparation prior to proteomic analysis. Journal of Chromatography A. 1023 (2), 317-320 (2004).
- Hildonen, S., Halvorsen, T. G., Reubsaet, L. Why less is more when generating tryptic peptides in bottom-up proteomics. Proteomics. 14 (17-18), 2031-2041 (2014).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19, 161 (2018).
- Drouin, N., et al. Capillary electrophoresis-mass spectrometry at trial by metabo-ring: effective electrophoretic mobility for reproducible and robust compound annotation. Analytical Chemistry. 92 (20), 14103-14112 (2020).
- Sun, L. L., Zhu, G. J., Zhang, Z. B., Mou, S., Dovichi, N. J. Third-generation electrokinetically pumped sheath-flow nanospray interface with improved stability and sensitivity for automated capillary zone electrophoresis-mass spectrometry analysis of complex proteome digests. Journal of Proteome Research. 14 (5), 2312-2321 (2015).
- DeLaney, K., Sauer, C. S., Vu, N. Q., Li, L. J. Recent advances and new perspectives in capillary electrophoresis-mass spectrometry for single cell "omics". Molecules. 24 (1), 21 (2019).
- Nemes, P., Rubakhin, S. S., Aerts, J. T., Sweedler, J. V. Qualitative and quantitative metabolomic investigation of single neurons by capillary electrophoresis electrospray ionization mass spectrometry. Nature Protocols. 8 (4), 783-799 (2013).
- Choi, S. B., Zamarbide, M., Manzini, M. C., Nemes, P. Tapered-tip capillary electrophoresis nano-electrospray ionization mass spectrometry for ultrasensitive proteomics: the mouse cortex. Journal of the American Society for Mass Spectrometry. 28 (4), 597-607 (2017).
- Pino, L. K., Rose, J., O’Broin, A., Shah, S., Schilling, B. Emerging mass spectrometry-based proteomics methodologies for novel biomedical applications. Biochemical Society Transactions. 48 (5), 1953-1966 (2020).
- Chen, C., Hou, J., Tanner, J. J., Cheng, J. L. Bioinformatics methods for mass spectrometry-based proteomics data analysis. International Journal of Molecular Sciences. 21 (8), 25 (2020).
- Peshkin, L., et al. On the relationship of protein and mRNA dynamics in vertebrate embryonic development. Developmental Cell. 35 (3), 383-394 (2015).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Gygi, S. P., et al. Quantitative analysis of complex protein mixtures using isotope-coded affinity tags. Nature Biotechnology. 17 (10), 994-999 (1999).
- Thompson, A., et al. Tandem mass tags: A novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75 (8), 1895-1904 (2003).
- Mi, H. Y., et al. PANTHER version 16: a revised family classification, tree-based classification tool, enhancer regions and extensive api. Nucleic Acids Research. 49, 394-403 (2021).
- Schmidt, E., et al. . On the Move Federated Workshops. , 710-719 (2006).
- Deutsch, E. W., et al. Trans-Proteomic pipeline, a standardized data processing pipeline for large-scale reproducible proteomics informatics. Proteomics Clinical Applications. 9 (7-8), 745-754 (2015).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Demsar, J., et al. Orange: Data mining toolbox in Python. Journal of Machine Learning Research. 14, 2349-2353 (2013).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. Journal of Proteome Research. 8 (5), 2144-2156 (2009).
- Jensen, L. J., et al. STRING 8 – a global view on proteins and their functional interactions in 630 organisms. Nucleic Acids Research. 37, 412-416 (2009).
- Schweppe, D. K., Huttlin, E. L., Harper, J. W., Gygi, S. P. BioPlex display: an interactive suite for large-scale AP-MS protein-protein interaction data. Journal of Proteome Research. 17 (1), 722-726 (2018).
- Hornbeck, P. V., et al. PhosphoSitePlus, 2014: mutations, PTMs and recalibrations. Nucleic Acids Research. 43, 512-520 (2015).
- Letunic, I., Khedkar, S., Bork, P. SMART: recent updates, new developments and status in 2020. Nucleic Acids Research. 49, 458-460 (2021).
- Lombard-Banek, C., et al. In vivo subcellular mass spectrometry enables proteo-metabolomic single-cell systems biology in a chordate embryo developing to a normally behaving tadpole (X. laevis). Angewandte Chemie-International Edition. 60 (23), 12852-12858 (2021).
- Lombard-Banek, C., Reddy, S., Moody, S. A., Nemes, P. Label-free quantification of proteins in single embryonic cells with neural fate in the cleavage-stage frog (Xenopus laevis) embryo using capillary electrophoresis electrospray ionization high-resolution mass spectrometry (CE-ESI-HRMS). Molecular & Cellular Proteomics. 15 (8), 2756-2768 (2016).
- Saha-Shah, A., et al. Single cell proteomics by data-independent acquisition to study embryonic asymmetry in Xenopus laevis. Analytical Chemistry. 91 (14), 8891-8899 (2019).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. In situ microprobe single-cell capillary electrophoresis mass spectrometry: metabolic reorganization in single differentiating cells in the live vertebrate (Xenopus laevis) embryo. Analytical Chemistry. 89 (13), 7069-7076 (2017).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).

