Zellliniengesteuerte Massenspektrometrie-Proteomik im sich entwickelnden (Frosch-)Embryo
Summary
In dieser Arbeit beschreiben wir eine massenspektrometriebasierte proteomische Charakterisierung von Zelllinien mit bekannten Gewebeschicksalen im Embryo von Xenopus laevis im Wirbeltier.
Abstract
Die Charakterisierung molekularer Vorgänge bei der Entstehung von Geweben und Organen in Zellen eröffnet das Potenzial, die normale Entwicklung besser zu verstehen und effiziente Heilmittel für Krankheiten zu entwickeln. Technologien, die eine genaue Identifizierung und Quantifizierung verschiedener Arten und einer großen Anzahl von Proteinen ermöglichen, würden noch fehlende Informationen über molekulare Mechanismen liefern, die die Entwicklung von Geweben und Organismen in Raum und Zeit orchestrieren. Hier stellen wir ein Massenspektrometrie-basiertes Protokoll vor, das die Messung von Tausenden von Proteinen in identifizierten Zelllinien in Xenopus laevis (Frosch) Embryonen ermöglicht. Der Ansatz baut auf reproduzierbaren Zellschicksalskarten und etablierten Methoden auf, um Zellen und ihre Nachkommen (Klone) aus diesem Modell der Wirbeltierentwicklung zu identifizieren, fluoreszierend zu markieren, zu verfolgen und zu beproben. Nach der Entnahme des Zellinhalts mittels Mikroprobenahme oder der Isolierung von Zellen durch Dissektion oder fluoreszenzaktivierte Zellsortierung werden Proteine extrahiert und für die Bottom-up-Proteomik-Analyse verarbeitet. Flüssigkeitschromatographie und Kapillarelektrophorese werden verwendet, um eine skalierbare Trennung für die Proteindetektion und -quantifizierung mit hochauflösender Massenspektrometrie (HRMS) zu ermöglichen. Repräsentative Beispiele werden für die proteomische Charakterisierung von Nervengewebe-Schicksalszellen angeführt. Die zellliniengesteuerte HRMS-Proteomik ist an verschiedene Gewebe und Organismen anpassbar. Es ist sensitiv, spezifisch und quantitativ genug, um in die räumlich-zeitliche Dynamik des Proteoms während der Wirbeltierentwicklung zu blicken.
Introduction
Unser Verständnis der Zelldifferenzierung und der Genese von Geweben und Organen ist das Ergebnis jahrzehntelanger aufwendiger gezielter Screenings von Genen und ihren Produkten. Ein besseres Wissen über alle Biomoleküle und ihre Mengen während wichtiger zellulärer Ereignisse würde dazu beitragen, molekulare Mechanismen zu entschlüsseln, die die räumliche und zeitliche Musterung des Körperbauplans von Wirbeltieren steuern. Technologien, die molekulare Amplifikation und Sequenzierung ermöglichen, sind nun in der Lage, routinemäßig über eine große Anzahl von Genen und Transkripten zu berichten und hypothesengestützte Studien in der biologischen und translationalen Grundlagenforschung zu unterstützen. Um sich entwickelnde Systeme zu verstehen, spricht eine komplexe Beziehung zwischen Transkription und Translation für die direkte Analyse mehrerer Proteine und ihrer posttranslationalen Modifikationen. Die globale Proteomik unter Verwendung biologischer In-vitro-Systeme, wie z. B. induzierter pluripotenter Stammzellen, begann, die Mechanismen der Gewebeinduktion zu beschreiben 1,2. In komplexen Organismen, wie dem Wirbeltierembryo, beruht die Entwicklung auf Morphogengradienten im Kontext von Raum und Zeit3. Daraus folgt, dass das Wissen über proteomische Veränderungen bei der Differenzierung von Zellen zu spezialisierten Geweben, wie z. B. Nervengeweben, einen Schlüssel zur Entschlüsselung molekularer Programme bietet, die die normale und fehlerhafte Entwicklung steuern, und um Therapeutika der nächsten Generation zu steuern.
Der südafrikanische Krallenfrosch (Xenopus laevis) ist ein etabliertes Modell in der Zell- und Entwicklungs-, Neuro- und regenerativen Biologie. Der Nobelpreisfür Physiologie oder Medizin 2012 von Sir John Gurdon 4,5 für die Entdeckung der Pluripotenz des somatischen Kerns unterstrich die Bedeutung dieses Modells für Entdeckungen in Grundlagen- und translationalen Studien. Xenopus-Embryonen entwickeln sich außerhalb der Mutter, was die direkte Manipulation von Zellen, Zellklonen und Genexpression über verschiedene Entwicklungsstadien hinweg ermöglicht. Asymmetrische Pigmentierung und stereotype Zellteilungen ermöglichten die Kartierung reproduzierbarer Schicksalskarten des 16-6- und 32-zelligen Embryos im 7,8-Stadium. Für die hochauflösende Massenspektrometrie (HRMS)-basierte Proteomik besteht ein weiterer Vorteil des Modells aus der relativ großen Größe (~1 mm Durchmesser), die einen hohen Proteingehalt für die Analyse liefert (~130 μg in Embryonen im frühen Spaltungsstadium, ~10 μg Proteingehalt in einzelnen Zellen des 16-zelligen Embryos)9,10.
Derzeit ist HRMS die führende Technologie der Wahl für den Nachweis von Proteinen. Diese Technologie ermöglicht den direkten, sensitiven und spezifischen Nachweis und die Quantifizierung von mehreren, in der Regel Hunderten bis Tausenden verschiedener Proteine11. Die Bottom-up-Proteomik von HRMS umfasst eine Reihe miteinander verbundener Schritte. Nach der Extraktion aus der Zell-/Gewebeprobe werden Proteine mit einem proteolytischen Enzym, wie z.B. Trypsin, verdaut (Bottom-up-Proteomik). Die resultierenden Peptide werden anhand ihrer unterschiedlichen physikalisch-chemischen Eigenschaften getrennt, einschließlich Hydrophobie (Umkehrphasen-Flüssigkeitschromatographie, LC), Nettoladung (Ionenaustauschchromatographie), Größe (Größenausschlusschromatographie) oder elektrophoretische Mobilität (Kapillarelektrophorese, CE). Die Peptide werden dann aufgeladen (ionisiert), typischerweise durch Elektrospray-Ionisation (ESI), und Peptidionen werden durch Gasphasenfragmentierung mittels Tandem-HRMS detektiert und sequenziert. Die resultierenden Peptiddaten werden auf das Proteom des untersuchten Organismus abgebildet. Mit proteinspezifischer (proteotypischer) Peptidionen-Signalintensität, die mit der Konzentration korreliert, kann die Proteinquantifizierung markierungsfrei oder markierungsbasiert (Multiplexing-Quantifizierung) durchgeführt werden. Die HRMS-Proteomik liefert eine reichhaltige Informationsquelle über den molekularen Zustand des untersuchten Systems, die die Generierung von Hypothesen und anschließende funktionelle Studien ermöglicht.
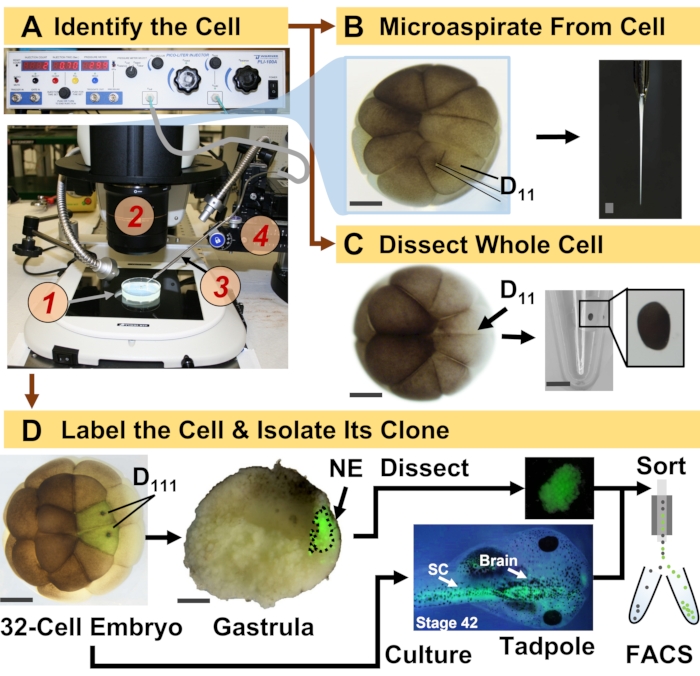
Abbildung 1: Räumlich-zeitlich skalierbare Proteomik, die eine zellliniengesteuerte HRMS-Proteomik im sich entwickelnden (Frosch-)Embryo ermöglicht. (A) Visualisierung der Probe (1) unter Verwendung eines Stereomikroskops (2) zur Injektion einer identifizierten Zelle (Inset) unter Verwendung einer hergestellten Mikropipette (3) unter Kontrolle durch einen Translationstisch (4). (B) Subzelluläre Probenahme der identifizierten linken D11-Zelle in einem 16-zelligen Embryo. (C) Präparation einer ganzenD-11-Zelle aus einem 16-zelligen Embryo. (D) Fluoreszierende (grüne) Nachverfolgung der linken und rechten D111-Nachkommen aus einem 32-zelligen Embryo zur Steuerung der Dissektion des neuralen Ektoderms (NE) in der Gastrula (Stadium 10) und Isolierung des Nachkommengewebes von der Kaulquappe mittels FACS. Maßstabsbalken: 200 μm für Embryonen, 1,25 mm für das Fläschchen. Die Abbildungen wurden mit Genehmigung der Referenzen 15,19,21,59 angepasst. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Das hier vorgestellte Protokoll ermöglicht die HRMS-basierte Quantifizierung einer großen Anzahl von Proteinen in identifizierten Zellen/Geweben in sich entwickelnden X. laevis-Embryonen. Der Ansatz baut auf einer genauen Zellidentifikation, reproduzierbaren Zellschicksalskarten und etablierten Methoden zur Verfolgung von Zelllinien in diesem biologischen Modell auf 6,7,8. Wie in Abbildung 1 gezeigt, untersuchen wir Proteome aus einzelnen Zellen, indem wir Ganzzelldissektion oder Kapillarmikroskopie verwenden, um zellulären Inhalt abzusaugen. Die Überwachung der Abstammungslinie einer Zelle ermöglicht es uns, die raumzeitliche Entwicklung des Proteoms zu untersuchen, wenn Zellen während der Gastrulation Gewebe bilden. Die Nachkommen der Zelle werden fluoreszenzmarkiert, indem ein Fluorophor injiziert wird, der an inertes Dextran oder mRNA für fluoreszierendes Protein (z. B. grün fluoreszierendes Protein oder GFP) konjugiert ist. Die markierten Nachkommen werden zu den gewünschten Entwicklungszeitpunkten isoliert. Während der Gastrulation können Zellklone, die eng geclustert sind, durch Dissektion isoliert werden. Nach der Gastrulation können Zellklone aufgrund von Migrationsbewegungen innerhalb des Embryos verteilt und durch fluoreszenzaktivierte Zellsortierung (FACS) aus dissoziierten Geweben isoliert werden. Proteine in diesen Zellen und Geweben werden mittels Bottom-up-Proteomik unter Verwendung von HPLC oder CE zur Trennung und ESI-Tandem-HRMS zur Identifizierung gemessen. Die zellliniengesteuerte HRMS-Proteomik ist auf verschiedene Zellgrößen und Abstammungslinien innerhalb des Embryos skalierbar und spezifisch, sensitiv und quantitativ. Anhand ausgewählter Beispiele, die hier gezeigt werden, zeigen wir auch, dass dieses Protokoll skalierbar und breit an verschiedene Zelltypen und Zelllinien anpassbar ist.
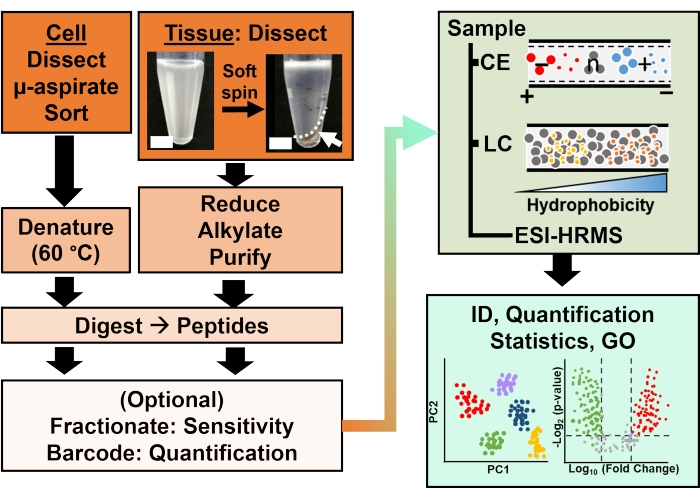
Abbildung 2: Der bioanalytische Arbeitsablauf. Die Mikrodissektion und Kapillaraspiration (FACS) erleichterte die Probenahme des zellulären und klonalen Proteingehalts. Die Depletion von reichlich vorhandenen Dotterproteinen und die Trennung durch Kapillarelektrophorese (CE) oder Nano-Flow-Flüssigkeitschromatographie (LC) verbesserte die Identifikationsempfindlichkeit (ID) unter Verwendung von hochauflösender Massenspektrometrie (HRMS) mit Elektrospray-Ionisation (ESI). Die Quantifizierung ergab eine Dysregulation, die neue Informationen für hypothesengetriebene Studien in Verbindung mit Informationen aus der Genontologie (GO) lieferte. Die Abbildungen wurden mit Genehmigung von Referenz15 angepasst. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Protocol
Representative Results
Discussion
Dieses Protokoll ermöglicht die Charakterisierung der Proteinexpression in identifizierten Zelllinien in Embryonen der Xenopus-Spezies . Die Methodik, die auf HRMS zurückgeht, kombiniert eine hervorragende Spezifität bei der molekularen Identifizierung, die Fähigkeit zur Detektion mehrerer Proteine ohne molekulare Sonden (in der Regel Hunderte bis Tausende verschiedener Proteine) und die Fähigkeit zur Quantifizierung. Die Anpassungsfähigkeit an klassische Werkzeuge und Arbeitsabläufe in der Zell- und Entw…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Wir danken Jie Li (University of Maryland, College Park) für die wertvollen Diskussionen über embryonale Dissoziation und FACS. Wir danken Vi M. Quach und Camille Lombard-Banek für die Unterstützung bei der Probenvorbereitung und Datenerhebung in früheren Studien, die beispielhaft für die proteomischen Anwendungen stehen, die in diesem Protokoll hervorgehoben werden. Teile dieser Arbeit wurden von der National Science Foundation unter der Fördernummer IOS-1832968 CAREER (an P.N.), den National Institutes of Health unter der Fördernummer R35GM124755 (an P.N.), dem University of Maryland-National Cancer Institute Partnership Program (an P.N.) und den Forschungspreisen der COSMOS Club Foundation (an A.B.B. und L.R.P.) unterstützt.
Materials
| Acetonitrile (LC-MS-grade) | Fisher Scientific | A955 | |
| Agarose | ThermoFisher Scientific | R0492 | |
| Ammonium bicarbonate | Fisher Scientific | A643-500 | |
| Analytical Column | Thermo Scientific | 164941 | |
| Analytical microbalance | Mettler-Toledo | XSE105DU | |
| Automatic peptide fractionation platform | Agilent | 1260 Infinity II | |
| Borosilicate Capillaries | Sutter Instruments Co. | B100-50-10 | |
| Borosilicate Capillaries (for making Emmitters) | Sutter Instruments | B100-75-10 | |
| C18 spin columns (for desalting) | ThermoFisher Scientific | 89870 | |
| Camera ro monitor electrospray | Edmund Optics Inc. | EO-2018C | |
| Combretastatin A4 | Millipore Sigma | C7744 | |
| Commercial CESI system | AB SCIEX | CESI | |
| (Cyclohexylamino)-1-propanesulfonic acid (CAPS) | VWR | 97061-492 | |
| Cytochalasin D | Millipore Sigma | C8273 | |
| Dextran, Alexa Fluor 488; 10,000 MW, Anionic, Fixable | ThermoFisher Scientific | D22910 | |
| Diothiothreitol | Fisher Scientific | FERR0861 | |
| Dumont #5 Forceps | Fine Science Tools | 11252-30 | |
| EDTA | Fisher Scientific | AAJ62786AP | |
| Epifluorescence light source | Lumencore | AURA III | |
| Eppendorf LoBing microcentrifuge tubes: protein | Fisher Scientific | 13-698-793 | |
| Formic acid (LC-MS-grade) | Fisher Scientific | A117-50 | |
| Freezer (-20 °C) | Fisher Scientific | 97-926-1 | |
| Freezer (-80 °C) | Thermo Scientific | TSX40086A | |
| Fused silica capillary | Molex | 1088150596 | |
| Heat Block | Benchmark | BSH300 | |
| High pressure liquid Chromatography System | ThermoFisher Scientific | Dionex Ultimate 3000 RSLC nanosystem | |
| High voltage power supply | Spellman | CZE1000R | |
| High-resolution Mass Spectrometer | ThermoFisher Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | |
| HPLC caps | Thermo Scientific | C4013-40A | |
| HPLC Vials | Thermo Scientific | C4013-11 | |
| Illuminator e.g. Goosenecks | Nikon | C-FLED2 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide | Fisher Scientific | AC122275000 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456-4 | |
| Microcapillary puller | Suttor Instruments | P-2000 | |
| Microinjector | Warner Instrument, Handem, CT | PLI-100A | |
| Micropippette puller | Sutter Instruments Co. | P-1000 | |
| MS data analysis software, commercial | ProteomeDiscoverer | ||
| MS data analysis software, opensource | MaxQuant | ||
| non-idet 40 substitute | Millipore Sigma | 11754599001 | |
| Petri dish 60 mm and 80 mm | Fisher Scientific | S08184 | |
| Pierce 10 µL bed Zip-tips (for desalting) | ThermoFisher Scientific | 87782 | |
| Pierce bicinchoninic acid protein assay kit | ThermoFisher Scientific | 23225 | |
| Pierce quantitative colorimetric peptide assay | ThermoFisher Scientific | 23275 | |
| Pierce Trypsin Protease (MS Grade) | Fisher Scientific | PI90058 | |
| Protein LoBind vials | Eppendorf | 0030108434 , 0030108442 |
|
| Refrigerated Centrifuge | Eppendorf | 5430R | |
| Refrigerated Incubator | Thermo Scientific | PR505755R/3721 | |
| sodium isethionate | Millipore Sigma | 220078 | |
| sodium pyrophosphate | Sigma Aldrich | 221368-100G | |
| Stainless steel BGE vial | Custom-Built | ||
| Stainless steel sample vials | Custom-Built | ||
| Stereomicroscope (objective 10x) | Nikon | SMZ 1270, SZX18 | |
| Sucrose | VWR | 97063-790 | |
| Syringe pumps (2) | Harvard Apparatus | 704506 | |
| Syringes (gas-tight): 500–1000 µL | Hamilton | 1750TTL | |
| Transfer pipettes (Plastic, disposable) | Fisher Scientific | 13-711-7M | |
| Trap Column | Thermo Scientific | 164750 | |
| Tris-HCl (1 M solution) | Fisher Scientific | AAJ22638AP | |
| Vacuum concentrator capable of operation at 4–10 °C | Labconco | 7310022 | |
| Vortex-mixer | Benchmark | BS-VM-1000 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| XYZ translation stage | Thorlabs | PT3 | |
| XYZ translation stage | Custom-Built |
References
- Shoemaker, L. D., Kornblum, H. I. Neural Stem Cells (NSCs) and Proteomics. Molecular & Cellular Proteomics. 15 (2), 344-354 (2016).
- Cervenka, J., et al. Proteomic characterization of human neural stem cells and their secretome during in vitro differentiation. Frontiers in Cellular Neuroscience. 14, 612560 (2021).
- Christian, J. L. Morphogen gradients in development: From form to function. Wiley Interdisciplinary Reviews. Developmental Biology. 1 (1), 3-15 (2012).
- Gurdon, J. B., Elsdale, T. R., M, F. Sexually mature individuals of Xenopus laevis from the transplantation of single somatic nuclei. Nature. 182, 64-65 (1958).
- Harland, R. M., Grainger, R. M. Xenopus research: metamorphosed by genetics and genomics. Trends in Genetics. 27 (12), 507-515 (2011).
- Moody, S. A. Fates of the blastomeres of the 16-cell stage Xenopus embryo. Developmental Biology. 119 (2), 560-578 (1987).
- Moody, S. A. Fates of the blastomeres of the 32-cell stage Xenopus embryo. Developmental Biology. 122 (2), 300-319 (1987).
- Dale, L., Slack, J. M. W. Fate map for the 32-cell stage of Xenopus laevis. Development. 99 (4), 527-551 (1987).
- Sun, L. L., et al. Single cell proteomics using frog (Xenopus laevis) blastomeres isolated from early stage embryos, which form a geometric progression in protein content. Analytical Chemistry. 88 (13), 6653-6657 (2016).
- Lombard-Banek, C., Moody, S. A., Nemes, P. Single-cell mass spectrometry for discovery proteomics: quantifying translational cell heterogeneity in the 16-cell frog (Xenopus) embryo. Angewandte Chemie-International Edition. 55 (7), 2454-2458 (2016).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early development of Xenopus laevis: A laboratory manual. , (2000).
- Briggs, J. A., et al. The dynamics of gene expression in vertebrate embryogenesis at single-cell resolution. Science. 360 (6392), (2018).
- Gupta, M., Sonnett, M., Ryazanova, L., Presler, M., Wuhr, M., Vleminckx, K. Quantitative proteomics of xenopus embryos I, sample preparation. Xenopus. Methods in Molecular Biology. 1865, 175-194 (2018).
- Baxi, A. B., Lombard-Banek, C., Moody, S. A., Nemes, P. Proteomic characterization of the neural ectoderm fated cell clones in the Xenopus laevis embryo by high-resolution mass spectrometry. ACS Chemical Neuroscience. 9 (8), 2064-2073 (2018).
- Moody, S. A. Cell lineage analysis in Xenopus embryos. Methods in Molecular Biology. 135, 331-347 (2000).
- Sater, A. K., Moody, S. A. Using Xenopus to understand human diseases and developmental disorders. Genesis. 55 (1-2), 1-14 (2017).
- Lombard-Banek, C., Choi, S. B., Nemes, P., Allbritton, N. L., Kovarik, M. L. . Enzyme Activity in Single Cells. Methods in Enzymology. 628, 263-292 (2019).
- Lombard-Banek, C., Moody, S. A., Nemes, P. High-sensitivity mass spectrometry for probing gene translation in single embryonic cells in the early frog (Xenopus) embryo. Frontiers in Cell and Developmental Biology. 4, 11 (2016).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. Microprobe capillary electrophoresis mass spectrometry for single-cell metabolomics in live frog (Xenopus laevis) embryos. Journal of Visualized Experiments: JoVE. (130), e56956 (2017).
- Lombard-Banek, C., Moody, S. A., Manzin, M. C., Nemes, P. Microsampling capillary electrophoresis mass spectrometry enables single-cell proteomics in complex tissues: developing cell clones in live Xenopus laevis and zebrafish embryos. Analytical Chemistry. 91 (7), 4797-4805 (2019).
- Klein, S. L. The first cleavage furrow demarcates the dorsal-ventral axis in Xenopus embryos. Developmental Biology. 120 (1), 299-304 (1987).
- Karimi, K., et al. Xenbase: a genomic, epigenomic and transcriptomic model organism database. Nucleic Acids Research. 46 (1), 861-868 (2018).
- Kakebeen, A. D., Chitsazan, A. D., Wills, A. E. Tissue disaggregation and isolation of specific cell types from transgenic Xenopus appendages for transcriptional analysis by FACS. Developmental Dynamics. 250 (9), 1381-1392 (2021).
- Garcia, B. A. What does the future hold for top down mass spectrometry. Journal of the American Society for Mass Spectrometry. 21 (2), 193-202 (2010).
- Toby, T. K., Fornelli, L., Kelleher, N. L. Progress in top-down proteomics and the analysis of proteoforms. Annual Review of Analytical Chemistry. (Palo Alto Calif). 9 (1), 499-519 (2016).
- Zhang, Z. B., Dubiak, K. M., Huber, P. W., Dovichi, N. J. Miniaturized filter-aided sample preparation (MICRO-FASP) method for high throughput, ultrasensitive proteomics sample preparation reveals proteome asymmetry in Xenopus laevis Embryos. Analytical Chemistry. 92 (7), 5554-5560 (2020).
- Wisniewski, J. R., Becher, D. . Microbial Proteomics: Methods and Protocols.Methods in Molecular Biology. 1841, 3-10 (2018).
- Hughes, C. S., et al. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9, 882 (2018).
- Wessel, D., Flugge, U. I. A method for the quantitative recovery of protein in dilute-solution in the presence of detergents and lipids. Analytical Biochemistry. 138 (1), 141-143 (1984).
- Jiang, L., He, L., Fountoulakis, M. Comparison of protein precipitation methods for sample preparation prior to proteomic analysis. Journal of Chromatography A. 1023 (2), 317-320 (2004).
- Hildonen, S., Halvorsen, T. G., Reubsaet, L. Why less is more when generating tryptic peptides in bottom-up proteomics. Proteomics. 14 (17-18), 2031-2041 (2014).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19, 161 (2018).
- Drouin, N., et al. Capillary electrophoresis-mass spectrometry at trial by metabo-ring: effective electrophoretic mobility for reproducible and robust compound annotation. Analytical Chemistry. 92 (20), 14103-14112 (2020).
- Sun, L. L., Zhu, G. J., Zhang, Z. B., Mou, S., Dovichi, N. J. Third-generation electrokinetically pumped sheath-flow nanospray interface with improved stability and sensitivity for automated capillary zone electrophoresis-mass spectrometry analysis of complex proteome digests. Journal of Proteome Research. 14 (5), 2312-2321 (2015).
- DeLaney, K., Sauer, C. S., Vu, N. Q., Li, L. J. Recent advances and new perspectives in capillary electrophoresis-mass spectrometry for single cell "omics". Molecules. 24 (1), 21 (2019).
- Nemes, P., Rubakhin, S. S., Aerts, J. T., Sweedler, J. V. Qualitative and quantitative metabolomic investigation of single neurons by capillary electrophoresis electrospray ionization mass spectrometry. Nature Protocols. 8 (4), 783-799 (2013).
- Choi, S. B., Zamarbide, M., Manzini, M. C., Nemes, P. Tapered-tip capillary electrophoresis nano-electrospray ionization mass spectrometry for ultrasensitive proteomics: the mouse cortex. Journal of the American Society for Mass Spectrometry. 28 (4), 597-607 (2017).
- Pino, L. K., Rose, J., O’Broin, A., Shah, S., Schilling, B. Emerging mass spectrometry-based proteomics methodologies for novel biomedical applications. Biochemical Society Transactions. 48 (5), 1953-1966 (2020).
- Chen, C., Hou, J., Tanner, J. J., Cheng, J. L. Bioinformatics methods for mass spectrometry-based proteomics data analysis. International Journal of Molecular Sciences. 21 (8), 25 (2020).
- Peshkin, L., et al. On the relationship of protein and mRNA dynamics in vertebrate embryonic development. Developmental Cell. 35 (3), 383-394 (2015).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Gygi, S. P., et al. Quantitative analysis of complex protein mixtures using isotope-coded affinity tags. Nature Biotechnology. 17 (10), 994-999 (1999).
- Thompson, A., et al. Tandem mass tags: A novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75 (8), 1895-1904 (2003).
- Mi, H. Y., et al. PANTHER version 16: a revised family classification, tree-based classification tool, enhancer regions and extensive api. Nucleic Acids Research. 49, 394-403 (2021).
- Schmidt, E., et al. . On the Move Federated Workshops. , 710-719 (2006).
- Deutsch, E. W., et al. Trans-Proteomic pipeline, a standardized data processing pipeline for large-scale reproducible proteomics informatics. Proteomics Clinical Applications. 9 (7-8), 745-754 (2015).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Demsar, J., et al. Orange: Data mining toolbox in Python. Journal of Machine Learning Research. 14, 2349-2353 (2013).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. Journal of Proteome Research. 8 (5), 2144-2156 (2009).
- Jensen, L. J., et al. STRING 8 – a global view on proteins and their functional interactions in 630 organisms. Nucleic Acids Research. 37, 412-416 (2009).
- Schweppe, D. K., Huttlin, E. L., Harper, J. W., Gygi, S. P. BioPlex display: an interactive suite for large-scale AP-MS protein-protein interaction data. Journal of Proteome Research. 17 (1), 722-726 (2018).
- Hornbeck, P. V., et al. PhosphoSitePlus, 2014: mutations, PTMs and recalibrations. Nucleic Acids Research. 43, 512-520 (2015).
- Letunic, I., Khedkar, S., Bork, P. SMART: recent updates, new developments and status in 2020. Nucleic Acids Research. 49, 458-460 (2021).
- Lombard-Banek, C., et al. In vivo subcellular mass spectrometry enables proteo-metabolomic single-cell systems biology in a chordate embryo developing to a normally behaving tadpole (X. laevis). Angewandte Chemie-International Edition. 60 (23), 12852-12858 (2021).
- Lombard-Banek, C., Reddy, S., Moody, S. A., Nemes, P. Label-free quantification of proteins in single embryonic cells with neural fate in the cleavage-stage frog (Xenopus laevis) embryo using capillary electrophoresis electrospray ionization high-resolution mass spectrometry (CE-ESI-HRMS). Molecular & Cellular Proteomics. 15 (8), 2756-2768 (2016).
- Saha-Shah, A., et al. Single cell proteomics by data-independent acquisition to study embryonic asymmetry in Xenopus laevis. Analytical Chemistry. 91 (14), 8891-8899 (2019).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. In situ microprobe single-cell capillary electrophoresis mass spectrometry: metabolic reorganization in single differentiating cells in the live vertebrate (Xenopus laevis) embryo. Analytical Chemistry. 89 (13), 7069-7076 (2017).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).

