Proteômica por Espectrometria de Massas Guiada por Linhagem Celular no Embrião em Desenvolvimento (Rã)
Summary
Descrevemos aqui uma caracterização proteômica baseada em espectrometria de massa de linhagens celulares com destino tecidual conhecido no embrião do vertebrado Xenopus laevis .
Abstract
A caracterização de eventos moleculares à medida que as células dão origem a tecidos e órgãos aumenta o potencial para entender melhor o desenvolvimento normal e projetar remédios eficientes para doenças. Tecnologias que permitam a identificação e quantificação precisas de diversos tipos e grande número de proteínas forneceriam informações ainda escassas sobre os mecanismos moleculares que orquestram o desenvolvimento de tecidos e organismos no espaço e no tempo. Aqui, apresentamos um protocolo baseado em espectrometria de massas que permite a medição de milhares de proteínas em linhagens celulares identificadas em embriões de Xenopus laevis (rã). A abordagem baseia-se em mapas reprodutíveis de destino celular e métodos estabelecidos para identificar, marcar fluorescentemente, rastrear e amostrar células e suas progênies (clones) a partir deste modelo de desenvolvimento de vertebrados. Após a coleta do conteúdo celular por microamostragem ou isolamento de células por dissecção ou classificação celular ativada por fluorescência, as proteínas são extraídas e processadas para análise proteômica ascendente. Cromatografia líquida e eletroforese capilar são usadas para fornecer separação escalável para detecção e quantificação de proteínas com espectrometria de massas de alta resolução (EMAR). Exemplos representativos são fornecidos para a caracterização proteômica de células adiposas do tecido neural. A proteômica da HRMS guiada por linhagem celular é adaptável a diferentes tecidos e organismos. É suficientemente sensível, específico e quantitativo para perscrutar a dinâmica espaço-temporal do proteoma durante o desenvolvimento dos vertebrados.
Introduction
Nossa compreensão da diferenciação celular e da gênese de tecidos e órgãos é o resultado de décadas de elaboradas telas direcionadas de genes e seus produtos. Aumentar nosso conhecimento de todas as biomoléculas e suas quantidades durante eventos celulares importantes ajudaria a desvendar mecanismos moleculares que controlam o padrão espacial e temporal do plano corporal dos vertebrados. Tecnologias que permitem amplificação molecular e sequenciamento são agora capazes de relatar rotineiramente um grande número de genes e transcritos, apoiando estudos orientados por hipóteses em pesquisa biológica básica e translacional. Para entender os sistemas em desenvolvimento, uma complexa relação entre transcrição e tradução defende a análise direta de múltiplas proteínas e suas modificações pós-traducionais. A proteômica global utilizando sistemas biológicos in vitro, como as células-tronco pluripotentes induzidas, começou a delinear mecanismos de indução tecidual 1,2. Em organismos complexos, como o embrião de vertebrados, o desenvolvimento depende de gradientes morfogênicos no contexto do espaço e do tempo3. Conclui-se que obter conhecimento das mudanças proteômicas à medida que as células se diferenciam para formar tecidos especializados, como tecidos neurais, oferece uma chave para desbloquear programas moleculares que controlam o desenvolvimento normal e defeituoso e orientar a terapêutica de próxima geração.
A rã-de-garra vertebrada sul-africana (Xenopus laevis) é um modelo bem estabelecido em biologia celular e de desenvolvimento, neuro e regenerativa. O Prêmio Nobel de Fisiologia ou Medicina de Sir John Gurdon em 2012 4,5 pela descoberta da pluripotência do núcleo somático destacou a importância desse modelo para descobertasem estudos básicos e translacionais. Os embriões de Xenopus desenvolvem-se externamente à mãe, facilitando assim a manipulação direta de células, clones celulares e expressão gênica ao longo de vários estágios de desenvolvimento. A pigmentação assimétrica e as divisões celulares estereotipadas permitiram o mapeamento de mapas reprodutíveis do destino do embrião de 16-6 e 32 célulasem 7,8 estádios. Para proteômica baseada em espectrometria de massa de alta resolução (EMAR), vantagens adicionais do modelo incluem tamanho relativamente grande (~1 mm de diâmetro), que produz conteúdo proteico abundante para análise (~130 μg em embriões em estágio inicial de clivagem, ~10 μg de conteúdo proteico em células isoladas do embrião de 16 células)9,10.
Atualmente, o HRMS é a tecnologia líder de escolha para detectar proteínas. Essa tecnologia permite a detecção e quantificação direta, sensível e específica de múltiplas, geralmente centenas a milhares de proteínas diferentes11. A proteômica ascendente por HRMS envolve uma série de etapas interconectadas. Após a extração da amostra de célula/tecido, as proteínas são digeridas com uma enzima proteolítica, como a tripsina (proteômica de baixo para cima). Os peptídeos resultantes são separados com base em suas diferentes propriedades físico-químicas, incluindo hidrofobicidade (cromatografia líquida de fase reversa, LC), carga líquida (cromatografia de troca iônica), tamanho (cromatografia de exclusão de tamanho) ou mobilidade eletroforética (eletroforese capilar, CE). Os peptídeos são então carregados (ionizados), tipicamente usando ionização por eletrospray (ESI), e íons peptídeos são detectados e sequenciados via fragmentação em fase gasosa por EMHR tandem. Os dados peptídicos resultantes são mapeados para o proteoma do organismo em estudo. Com a intensidade de sinal do peptídeo específico para proteínas (proteotípicas) correlacionando-se com a concentração, a quantificação da proteína pode ser realizada sem rótulos ou baseada em marcadores (quantificação multiplexação). A proteômica da HRMS fornece um rico recurso de informações sobre o estado molecular do sistema em estudo, permitindo a geração de hipóteses e estudos funcionais de acompanhamento.
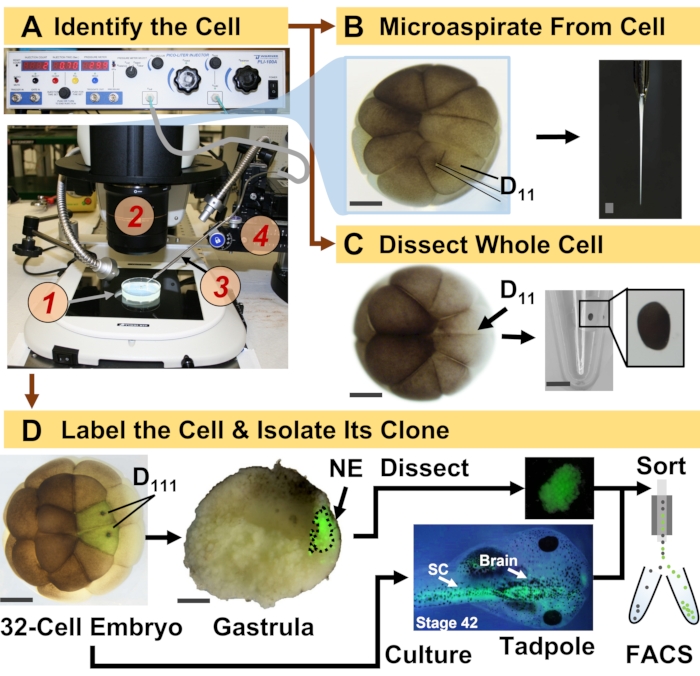
Figura 1: Proteômica espaço-temporalmente escalável permitindo a proteômica de HRMS guiada por linhagem celular no embrião em desenvolvimento (rã). (A) Visualização do espécime (1) utilizando um estereomicroscópio (2) para injeção de uma célula identificada (inset), utilizando uma micropipeta fabricada (3) sob controle por um estágio de translação (4). (B) Amostragem subcelular da célula D 11 esquerda identificada em um embrião de16 células. (C) Dissecção de uma célula D11 inteira de um embrião de 16 células. (D) Traçado fluorescente (verde) das progênies D111 esquerda e direita de um embrião de 32 células para guiar a dissecção do ectoderma neural (NE) na gástrula (estágio 10) e isolamento do tecido descendente do girino usando FACS. Barras de escala: 200 μm para embriões, 1,25 mm para o frasco para injetáveis. As figuras foram adaptadas com permissão das referências 15,19,21,59. Clique aqui para ver uma versão maior desta figura.
O protocolo aqui apresentado permite a quantificação baseada em HRMS de um grande número de proteínas em células/tecidos identificados em embriões de X. laevis em desenvolvimento. A abordagem baseia-se na identificação celular precisa, mapas de destino celular reprodutíveis e metodologias estabelecidas para rastrear linhagens celulares neste modelo biológico 6,7,8. Como mostrado na Figura 1, estudamos proteomas de células isoladas empregando dissecção de células inteiras ou microamostragem capilar para aspirar o conteúdo celular. O monitoramento da linhagem de uma célula nos permite estudar a evolução espaço-temporal do proteoma à medida que as células formam tecidos durante a gastrulação. A progênie celular é marcada fluorescentemente pela injeção de um fluoróforo conjugado a dextran inerte ou mRNA para proteína fluorescente (por exemplo, proteína fluorescente verde ou GFP). A progênie marcada é isolada nos momentos de desenvolvimento desejados. Durante a gastrulação, clones celulares que estão firmemente agrupados podem ser isolados por dissecção. Após a gastrulação, os clones celulares podem ser distribuídos dentro do embrião devido a movimentos migratórios e podem ser isolados de tecidos dissociados por triagem celular ativada por fluorescência (FACS). As proteínas nessas células e tecidos são medidas via proteômica bottom-up empregando HPLC ou CE para separação e ESI tandem HRMS para identificação. A proteômica da HRMS guiada por linhagens celulares é escalável para diferentes tamanhos e linhagens celulares dentro do embrião e é específica, sensível e quantitativa. Através de exemplos selecionados mostrados aqui, também demonstramos que este protocolo é escalável e amplamente adaptável a diferentes tipos de células e linhagens celulares.
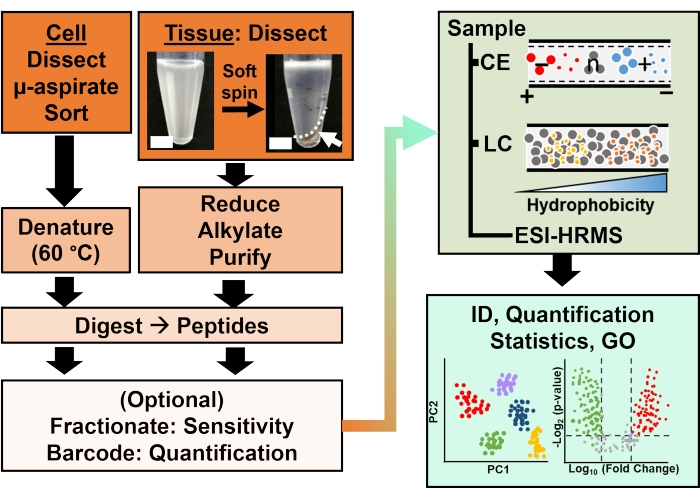
Figura 2: O fluxo de trabalho bioanalítico. A microdissecção e a aspiração capilar, ou FACS, facilitaram a amostragem do conteúdo de proteínas celulares e clonais. A depleção de proteínas vitelínicas abundantes e a separação por eletroforese capilar (CE) ou cromatografia líquida de nanofluxo (LC) aumentaram a sensibilidade de identificação (ID) usando espectrometria de massa de alta resolução (EMRH) com ionização por eletrospray (ESI). A quantificação revelou desregulação, fornecendo novas informações para estudos baseados em hipóteses em conjunto com informações disponíveis na ontologia gênica (GO). As figuras foram adaptadas com permissão da referência15. Clique aqui para ver uma versão maior desta figura.
Protocol
Representative Results
Discussion
Este protocolo possibilita a caracterização da expressão de proteínas em linhagens celulares identificadas em embriões da espécie Xenopus. Derivada do HRMS, a metodologia combina especificidade requintada na identificação molecular, capacidade de detecção de multiproteínas sem sondas moleculares (geralmente centenas a milhares de proteínas diferentes) e capacidade de quantificação. A adaptabilidade a ferramentas clássicas e fluxos de trabalho em (neuro)biologia celular e de desenvolvimento expande…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Somos gratos a Jie Li (University of Maryland, College Park) pelas valiosas discussões sobre dissociação embrionária e FACS. Agradecemos a Vi M. Quach e Camille Lombard-Banek pelo auxílio na preparação de amostras e coleta de dados em estudos anteriores exemplificando as aplicações proteômicas destacadas neste protocolo. Partes deste trabalho foram apoiadas pela National Science Foundation sob o número de prêmio IOS-1832968 CAREER (para P.N.), os Institutos Nacionais de Saúde sob o número de prêmio R35GM124755 (para P.N.), o Programa de Parceria do Instituto Nacional do Câncer da Universidade de Maryland (para P.N.) e os prêmios de pesquisa da Fundação COSMOS Club (para A.B.B. e L.R.P.).
Materials
| Acetonitrile (LC-MS-grade) | Fisher Scientific | A955 | |
| Agarose | ThermoFisher Scientific | R0492 | |
| Ammonium bicarbonate | Fisher Scientific | A643-500 | |
| Analytical Column | Thermo Scientific | 164941 | |
| Analytical microbalance | Mettler-Toledo | XSE105DU | |
| Automatic peptide fractionation platform | Agilent | 1260 Infinity II | |
| Borosilicate Capillaries | Sutter Instruments Co. | B100-50-10 | |
| Borosilicate Capillaries (for making Emmitters) | Sutter Instruments | B100-75-10 | |
| C18 spin columns (for desalting) | ThermoFisher Scientific | 89870 | |
| Camera ro monitor electrospray | Edmund Optics Inc. | EO-2018C | |
| Combretastatin A4 | Millipore Sigma | C7744 | |
| Commercial CESI system | AB SCIEX | CESI | |
| (Cyclohexylamino)-1-propanesulfonic acid (CAPS) | VWR | 97061-492 | |
| Cytochalasin D | Millipore Sigma | C8273 | |
| Dextran, Alexa Fluor 488; 10,000 MW, Anionic, Fixable | ThermoFisher Scientific | D22910 | |
| Diothiothreitol | Fisher Scientific | FERR0861 | |
| Dumont #5 Forceps | Fine Science Tools | 11252-30 | |
| EDTA | Fisher Scientific | AAJ62786AP | |
| Epifluorescence light source | Lumencore | AURA III | |
| Eppendorf LoBing microcentrifuge tubes: protein | Fisher Scientific | 13-698-793 | |
| Formic acid (LC-MS-grade) | Fisher Scientific | A117-50 | |
| Freezer (-20 °C) | Fisher Scientific | 97-926-1 | |
| Freezer (-80 °C) | Thermo Scientific | TSX40086A | |
| Fused silica capillary | Molex | 1088150596 | |
| Heat Block | Benchmark | BSH300 | |
| High pressure liquid Chromatography System | ThermoFisher Scientific | Dionex Ultimate 3000 RSLC nanosystem | |
| High voltage power supply | Spellman | CZE1000R | |
| High-resolution Mass Spectrometer | ThermoFisher Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | |
| HPLC caps | Thermo Scientific | C4013-40A | |
| HPLC Vials | Thermo Scientific | C4013-11 | |
| Illuminator e.g. Goosenecks | Nikon | C-FLED2 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide | Fisher Scientific | AC122275000 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456-4 | |
| Microcapillary puller | Suttor Instruments | P-2000 | |
| Microinjector | Warner Instrument, Handem, CT | PLI-100A | |
| Micropippette puller | Sutter Instruments Co. | P-1000 | |
| MS data analysis software, commercial | ProteomeDiscoverer | ||
| MS data analysis software, opensource | MaxQuant | ||
| non-idet 40 substitute | Millipore Sigma | 11754599001 | |
| Petri dish 60 mm and 80 mm | Fisher Scientific | S08184 | |
| Pierce 10 µL bed Zip-tips (for desalting) | ThermoFisher Scientific | 87782 | |
| Pierce bicinchoninic acid protein assay kit | ThermoFisher Scientific | 23225 | |
| Pierce quantitative colorimetric peptide assay | ThermoFisher Scientific | 23275 | |
| Pierce Trypsin Protease (MS Grade) | Fisher Scientific | PI90058 | |
| Protein LoBind vials | Eppendorf | 0030108434 , 0030108442 |
|
| Refrigerated Centrifuge | Eppendorf | 5430R | |
| Refrigerated Incubator | Thermo Scientific | PR505755R/3721 | |
| sodium isethionate | Millipore Sigma | 220078 | |
| sodium pyrophosphate | Sigma Aldrich | 221368-100G | |
| Stainless steel BGE vial | Custom-Built | ||
| Stainless steel sample vials | Custom-Built | ||
| Stereomicroscope (objective 10x) | Nikon | SMZ 1270, SZX18 | |
| Sucrose | VWR | 97063-790 | |
| Syringe pumps (2) | Harvard Apparatus | 704506 | |
| Syringes (gas-tight): 500–1000 µL | Hamilton | 1750TTL | |
| Transfer pipettes (Plastic, disposable) | Fisher Scientific | 13-711-7M | |
| Trap Column | Thermo Scientific | 164750 | |
| Tris-HCl (1 M solution) | Fisher Scientific | AAJ22638AP | |
| Vacuum concentrator capable of operation at 4–10 °C | Labconco | 7310022 | |
| Vortex-mixer | Benchmark | BS-VM-1000 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| XYZ translation stage | Thorlabs | PT3 | |
| XYZ translation stage | Custom-Built |
References
- Shoemaker, L. D., Kornblum, H. I. Neural Stem Cells (NSCs) and Proteomics. Molecular & Cellular Proteomics. 15 (2), 344-354 (2016).
- Cervenka, J., et al. Proteomic characterization of human neural stem cells and their secretome during in vitro differentiation. Frontiers in Cellular Neuroscience. 14, 612560 (2021).
- Christian, J. L. Morphogen gradients in development: From form to function. Wiley Interdisciplinary Reviews. Developmental Biology. 1 (1), 3-15 (2012).
- Gurdon, J. B., Elsdale, T. R., M, F. Sexually mature individuals of Xenopus laevis from the transplantation of single somatic nuclei. Nature. 182, 64-65 (1958).
- Harland, R. M., Grainger, R. M. Xenopus research: metamorphosed by genetics and genomics. Trends in Genetics. 27 (12), 507-515 (2011).
- Moody, S. A. Fates of the blastomeres of the 16-cell stage Xenopus embryo. Developmental Biology. 119 (2), 560-578 (1987).
- Moody, S. A. Fates of the blastomeres of the 32-cell stage Xenopus embryo. Developmental Biology. 122 (2), 300-319 (1987).
- Dale, L., Slack, J. M. W. Fate map for the 32-cell stage of Xenopus laevis. Development. 99 (4), 527-551 (1987).
- Sun, L. L., et al. Single cell proteomics using frog (Xenopus laevis) blastomeres isolated from early stage embryos, which form a geometric progression in protein content. Analytical Chemistry. 88 (13), 6653-6657 (2016).
- Lombard-Banek, C., Moody, S. A., Nemes, P. Single-cell mass spectrometry for discovery proteomics: quantifying translational cell heterogeneity in the 16-cell frog (Xenopus) embryo. Angewandte Chemie-International Edition. 55 (7), 2454-2458 (2016).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early development of Xenopus laevis: A laboratory manual. , (2000).
- Briggs, J. A., et al. The dynamics of gene expression in vertebrate embryogenesis at single-cell resolution. Science. 360 (6392), (2018).
- Gupta, M., Sonnett, M., Ryazanova, L., Presler, M., Wuhr, M., Vleminckx, K. Quantitative proteomics of xenopus embryos I, sample preparation. Xenopus. Methods in Molecular Biology. 1865, 175-194 (2018).
- Baxi, A. B., Lombard-Banek, C., Moody, S. A., Nemes, P. Proteomic characterization of the neural ectoderm fated cell clones in the Xenopus laevis embryo by high-resolution mass spectrometry. ACS Chemical Neuroscience. 9 (8), 2064-2073 (2018).
- Moody, S. A. Cell lineage analysis in Xenopus embryos. Methods in Molecular Biology. 135, 331-347 (2000).
- Sater, A. K., Moody, S. A. Using Xenopus to understand human diseases and developmental disorders. Genesis. 55 (1-2), 1-14 (2017).
- Lombard-Banek, C., Choi, S. B., Nemes, P., Allbritton, N. L., Kovarik, M. L. . Enzyme Activity in Single Cells. Methods in Enzymology. 628, 263-292 (2019).
- Lombard-Banek, C., Moody, S. A., Nemes, P. High-sensitivity mass spectrometry for probing gene translation in single embryonic cells in the early frog (Xenopus) embryo. Frontiers in Cell and Developmental Biology. 4, 11 (2016).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. Microprobe capillary electrophoresis mass spectrometry for single-cell metabolomics in live frog (Xenopus laevis) embryos. Journal of Visualized Experiments: JoVE. (130), e56956 (2017).
- Lombard-Banek, C., Moody, S. A., Manzin, M. C., Nemes, P. Microsampling capillary electrophoresis mass spectrometry enables single-cell proteomics in complex tissues: developing cell clones in live Xenopus laevis and zebrafish embryos. Analytical Chemistry. 91 (7), 4797-4805 (2019).
- Klein, S. L. The first cleavage furrow demarcates the dorsal-ventral axis in Xenopus embryos. Developmental Biology. 120 (1), 299-304 (1987).
- Karimi, K., et al. Xenbase: a genomic, epigenomic and transcriptomic model organism database. Nucleic Acids Research. 46 (1), 861-868 (2018).
- Kakebeen, A. D., Chitsazan, A. D., Wills, A. E. Tissue disaggregation and isolation of specific cell types from transgenic Xenopus appendages for transcriptional analysis by FACS. Developmental Dynamics. 250 (9), 1381-1392 (2021).
- Garcia, B. A. What does the future hold for top down mass spectrometry. Journal of the American Society for Mass Spectrometry. 21 (2), 193-202 (2010).
- Toby, T. K., Fornelli, L., Kelleher, N. L. Progress in top-down proteomics and the analysis of proteoforms. Annual Review of Analytical Chemistry. (Palo Alto Calif). 9 (1), 499-519 (2016).
- Zhang, Z. B., Dubiak, K. M., Huber, P. W., Dovichi, N. J. Miniaturized filter-aided sample preparation (MICRO-FASP) method for high throughput, ultrasensitive proteomics sample preparation reveals proteome asymmetry in Xenopus laevis Embryos. Analytical Chemistry. 92 (7), 5554-5560 (2020).
- Wisniewski, J. R., Becher, D. . Microbial Proteomics: Methods and Protocols.Methods in Molecular Biology. 1841, 3-10 (2018).
- Hughes, C. S., et al. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9, 882 (2018).
- Wessel, D., Flugge, U. I. A method for the quantitative recovery of protein in dilute-solution in the presence of detergents and lipids. Analytical Biochemistry. 138 (1), 141-143 (1984).
- Jiang, L., He, L., Fountoulakis, M. Comparison of protein precipitation methods for sample preparation prior to proteomic analysis. Journal of Chromatography A. 1023 (2), 317-320 (2004).
- Hildonen, S., Halvorsen, T. G., Reubsaet, L. Why less is more when generating tryptic peptides in bottom-up proteomics. Proteomics. 14 (17-18), 2031-2041 (2014).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19, 161 (2018).
- Drouin, N., et al. Capillary electrophoresis-mass spectrometry at trial by metabo-ring: effective electrophoretic mobility for reproducible and robust compound annotation. Analytical Chemistry. 92 (20), 14103-14112 (2020).
- Sun, L. L., Zhu, G. J., Zhang, Z. B., Mou, S., Dovichi, N. J. Third-generation electrokinetically pumped sheath-flow nanospray interface with improved stability and sensitivity for automated capillary zone electrophoresis-mass spectrometry analysis of complex proteome digests. Journal of Proteome Research. 14 (5), 2312-2321 (2015).
- DeLaney, K., Sauer, C. S., Vu, N. Q., Li, L. J. Recent advances and new perspectives in capillary electrophoresis-mass spectrometry for single cell "omics". Molecules. 24 (1), 21 (2019).
- Nemes, P., Rubakhin, S. S., Aerts, J. T., Sweedler, J. V. Qualitative and quantitative metabolomic investigation of single neurons by capillary electrophoresis electrospray ionization mass spectrometry. Nature Protocols. 8 (4), 783-799 (2013).
- Choi, S. B., Zamarbide, M., Manzini, M. C., Nemes, P. Tapered-tip capillary electrophoresis nano-electrospray ionization mass spectrometry for ultrasensitive proteomics: the mouse cortex. Journal of the American Society for Mass Spectrometry. 28 (4), 597-607 (2017).
- Pino, L. K., Rose, J., O’Broin, A., Shah, S., Schilling, B. Emerging mass spectrometry-based proteomics methodologies for novel biomedical applications. Biochemical Society Transactions. 48 (5), 1953-1966 (2020).
- Chen, C., Hou, J., Tanner, J. J., Cheng, J. L. Bioinformatics methods for mass spectrometry-based proteomics data analysis. International Journal of Molecular Sciences. 21 (8), 25 (2020).
- Peshkin, L., et al. On the relationship of protein and mRNA dynamics in vertebrate embryonic development. Developmental Cell. 35 (3), 383-394 (2015).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Gygi, S. P., et al. Quantitative analysis of complex protein mixtures using isotope-coded affinity tags. Nature Biotechnology. 17 (10), 994-999 (1999).
- Thompson, A., et al. Tandem mass tags: A novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75 (8), 1895-1904 (2003).
- Mi, H. Y., et al. PANTHER version 16: a revised family classification, tree-based classification tool, enhancer regions and extensive api. Nucleic Acids Research. 49, 394-403 (2021).
- Schmidt, E., et al. . On the Move Federated Workshops. , 710-719 (2006).
- Deutsch, E. W., et al. Trans-Proteomic pipeline, a standardized data processing pipeline for large-scale reproducible proteomics informatics. Proteomics Clinical Applications. 9 (7-8), 745-754 (2015).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Demsar, J., et al. Orange: Data mining toolbox in Python. Journal of Machine Learning Research. 14, 2349-2353 (2013).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. Journal of Proteome Research. 8 (5), 2144-2156 (2009).
- Jensen, L. J., et al. STRING 8 – a global view on proteins and their functional interactions in 630 organisms. Nucleic Acids Research. 37, 412-416 (2009).
- Schweppe, D. K., Huttlin, E. L., Harper, J. W., Gygi, S. P. BioPlex display: an interactive suite for large-scale AP-MS protein-protein interaction data. Journal of Proteome Research. 17 (1), 722-726 (2018).
- Hornbeck, P. V., et al. PhosphoSitePlus, 2014: mutations, PTMs and recalibrations. Nucleic Acids Research. 43, 512-520 (2015).
- Letunic, I., Khedkar, S., Bork, P. SMART: recent updates, new developments and status in 2020. Nucleic Acids Research. 49, 458-460 (2021).
- Lombard-Banek, C., et al. In vivo subcellular mass spectrometry enables proteo-metabolomic single-cell systems biology in a chordate embryo developing to a normally behaving tadpole (X. laevis). Angewandte Chemie-International Edition. 60 (23), 12852-12858 (2021).
- Lombard-Banek, C., Reddy, S., Moody, S. A., Nemes, P. Label-free quantification of proteins in single embryonic cells with neural fate in the cleavage-stage frog (Xenopus laevis) embryo using capillary electrophoresis electrospray ionization high-resolution mass spectrometry (CE-ESI-HRMS). Molecular & Cellular Proteomics. 15 (8), 2756-2768 (2016).
- Saha-Shah, A., et al. Single cell proteomics by data-independent acquisition to study embryonic asymmetry in Xenopus laevis. Analytical Chemistry. 91 (14), 8891-8899 (2019).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. In situ microprobe single-cell capillary electrophoresis mass spectrometry: metabolic reorganization in single differentiating cells in the live vertebrate (Xenopus laevis) embryo. Analytical Chemistry. 89 (13), 7069-7076 (2017).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).

