Cell-Lineage guidad masspektrometri proteomik i det utvecklande (groda) embryot
Summary
Här beskrivs en masspektrometribaserad proteomisk karakterisering av cellinjer med kända vävnadsöden i ryggradsdjurens Xenopus laevis embryo.
Abstract
Karakterisering av molekylära händelser som celler ger upphov till vävnader och organ ökar en potential att bättre förstå normal utveckling och utforma effektiva botemedel mot sjukdomar. Teknik som möjliggör noggrann identifiering och kvantifiering av olika typer och ett stort antal proteiner skulle ge fortfarande saknad information om molekylära mekanismer som orkestrerar vävnads- och organismutveckling i rum och tid. Här presenterar vi ett masspektrometribaserat protokoll som möjliggör mätning av tusentals proteiner i identifierade cellinjer i Xenopus laevis (groda) embryon. Tillvägagångssättet bygger på reproducerbara cell-ödeskartor och etablerade metoder för att identifiera, fluorescerande etikett, spåra och provceller och deras avkomma (kloner) från denna modell av ryggradsdjurens utveckling. Efter insamling av cellulärt innehåll med hjälp av mikroprovtagning eller isolering av celler genom dissektion eller fluorescensaktiverad cellsortering extraheras proteiner och bearbetas för proteomisk analys nedifrån och upp. Vätskekromatografi och kapillärelektrofores används för att tillhandahålla skalbar separation för proteindetektion och kvantifiering med högupplösande masspektrometri (HRMS). Representativa exempel ges för proteomisk karakterisering av nervvävnadsfatade celler. Celllinjestyrd HRMS-proteomik kan anpassas till olika vävnader och organismer. Det är tillräckligt känsligt, specifikt och kvantitativt för att kika in i proteomets spatio-temporala dynamik under ryggradsdjurens utveckling.
Introduction
Vår förståelse av celldifferentiering och uppkomsten av vävnader och organ är resultatet av årtionden av utarbetade riktade skärmar av gener och deras produkter. Att öka vår kunskap om alla biomolekyler och deras kvantiteter under viktiga cellulära händelser skulle hjälpa till att avslöja molekylära mekanismer som styr det rumsliga och tidsmässiga mönstret för ryggradsdjurens kroppsplan. Teknologier som möjliggör molekylär amplifiering och sekvensering kan nu rutinmässigt rapportera om ett stort antal gener och transkript, vilket stöder hypotesdrivna studier inom grundläggande biologisk och translationell forskning. För att förstå utvecklingssystem förespråkar ett komplext förhållande mellan transkription och translation direkt analys av flera proteiner och deras posttranslationella modifieringar. Global proteomik som använder in vitro biologiska system, såsom inducerade pluripotenta stamceller, började avgränsa mekanismer för vävnadsinduktion 1,2. I komplexa organismer, såsom ryggradsdjurens embryo, är utvecklingen beroende av morfogengradienter i samband med rum och tid3. Det följer att få kunskap om proteomiska förändringar när celler differentieras för att bilda specialiserade vävnader, såsom neurala vävnader, erbjuder en nyckel för att låsa upp molekylära program som styr normal och defekt utveckling och vägleda nästa generations terapi.
Den sydafrikanska klogrodan (Xenopus laevis) är en väletablerad modell inom cell- och utvecklings-, neuro- och regenerativ biologi. Sir John Gurdons Nobelpris i fysiologi eller medicin 2012 4,5 för upptäckten av pluripotens hos den somatiska kärnan belyste vikten av denna modell för upptäckter i grundläggande och translationella studier. Xenopus-embryon utvecklas externt till modern, vilket underlättar direkt manipulation av celler, cellkloner och genuttryck över olika utvecklingsstadier. Asymmetrisk pigmentering och stereotypa celldelningar möjliggjorde kartläggning av reproducerbara ödeskartor från 16-6 och 32-celliga 7,8-stegs embryo. För högupplöst masspektrometri (HRMS) baserad proteomik inkluderar ytterligare fördelar med modellen relativt stor storlek (~ 1 mm i diameter), vilket ger rikligt proteininnehåll för analys (~ 130 μg i embryon i tidigt klyvningsstadium, ~ 10 μg proteininnehåll i enskilda celler i 16-cellembryot)9,10.
För närvarande är HRMS den ledande tekniken för att detektera proteiner. Denna teknik möjliggör direkt, känslig och specifik detektion och kvantifiering av flera, vanligtvis hundratusentals olika proteiner11. Bottom-up-proteomik av HRMS innebär en serie sammankopplade steg. Efter extraktion från cell/vävnadsprovet spjälkas proteiner med ett proteolytiskt enzym, såsom trypsin (bottom-up proteomik). De resulterande peptiderna separeras baserat på deras olika fysikalisk-kemiska egenskaper, inklusive hydrofobicitet (vätskekromatografi med omvänd fas, LC), nettoladdning (jonbyteskromatografi), storlek (storleksuteslutningskromatografi) eller elektroforetisk rörlighet (kapillärelektrofores, CE). Peptider laddas sedan (joniseras), vanligtvis med användning av elektrosprayjonisering (ESI), och peptidjoner detekteras och sekvenseras via gasfasfragmentering genom tandem HRMS. De resulterande peptiddata mappas till proteomet hos organismen som studeras. Med proteinspecifik (proteotypisk) peptidjonsignalintensitet korrelerad med koncentration kan proteinkvantifiering utföras etikettfri eller etikettbaserad (multiplexeringskvantifiering). HRMS-proteomik ger en rik resurs av information om det molekylära tillståndet i systemet som studeras, vilket möjliggör generering av hypoteser och uppföljande funktionella studier.
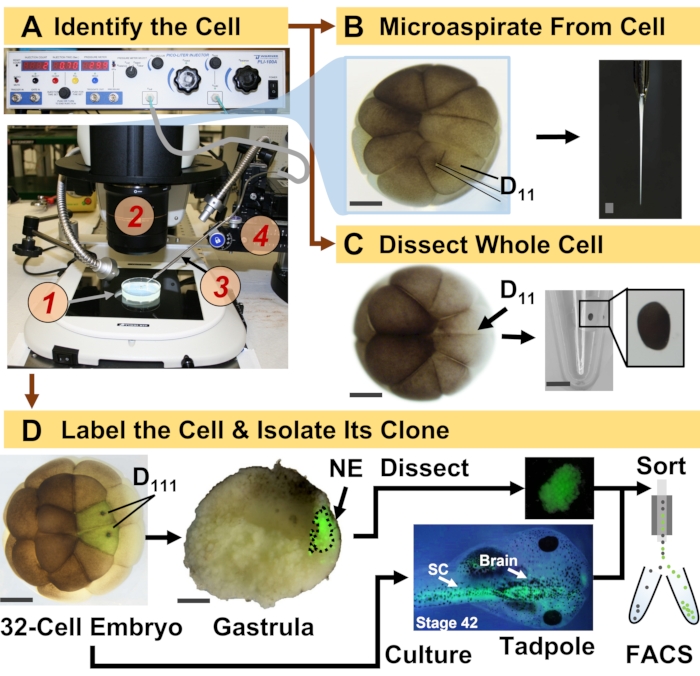
Figur 1: Spatiotemporalt skalbar proteomik som möjliggör cellinjestyrd HRMS-proteomik i det utvecklande (grod) embryot. (A) Visualisering av provet (1) med hjälp av ett stereomikroskop (2) för injektion av en identifierad cell (infälld) med hjälp av en tillverkad mikropipett (3) under kontroll genom ett translationssteg (4). b) Subcellulär provtagning av den identifierade vänstra D11-cellen i ett 16-celligt embryo. c) Dissektion av en hel D11-cell från ett embryo med 16 celler. (D) Fluorescerande (grön) spårning av vänster och höger D111-avkomma från ett 32-celligt embryo för att styra dissektion av neural ektoderm (NE) i gastrula (steg 10) och isolering av den nedstigande vävnaden från grodyngel med hjälp av FACS. Skalstänger: 200 μm för embryon, 1,25 mm för injektionsflaskan. Siffrorna anpassades med tillstånd från referenserna 15,19,21,59. Klicka här för att se en större version av denna figur.
Protokollet som presenteras här möjliggör HRMS-baserad kvantifiering av ett stort antal proteiner i identifierade celler/vävnader i utvecklande X. laevis embryon. Tillvägagångssättet bygger på noggrann cellidentifiering, reproducerbara cellödeskartor och etablerade metoder för att spåra cellinjer i denna biologiska modell 6,7,8. Som visas i figur 1 studerar vi proteom från enskilda celler genom att använda helcellsdissektion eller kapillär mikroprovtagning för att aspirera cellulärt innehåll. Övervakning av en cells härstamning gör det möjligt för oss att studera proteomets spatiotemporala utveckling när celler bildar vävnader under gastrulation. Cellavkomman markeras fluorescerande genom att injicera en fluorofor konjugerad till inert dextran eller mRNA för fluorescerande protein (t.ex. grönt fluorescerande protein eller GFP). Den märkta avkomman isoleras vid önskade utvecklingstidpunkter. Under gastrulation kan cellkloner som är tätt grupperade isoleras genom dissektion. Efter gastrulation kan cellkloner distribueras inom embryot på grund av migrationsrörelser och kan isoleras från dissocierade vävnader genom fluorescensaktiverad cellsortering (FACS). Proteiner i dessa celler och vävnader mäts via bottom-up-proteomik som använder HPLC eller CE för separation och ESI-tandem-HRMS för identifiering. Celllinjestyrd HRMS-proteomik är skalbar till olika cellstorlekar och linjer inom embryot och är specifik, känslig och kvantitativ. Genom utvalda exempel som visas här visar vi också att detta protokoll är skalbart och i stort sett anpassningsbart till olika typer av celler och cellinjer.
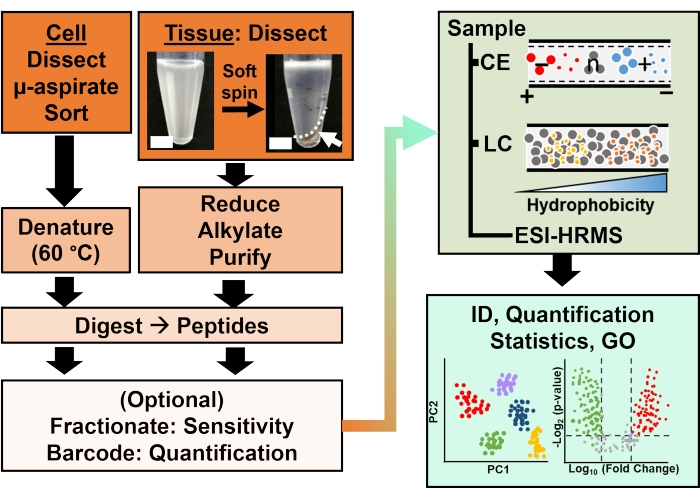
Figur 2: Det bioanalytiska arbetsflödet. Mikrodissektion och kapilläraspiration, eller FACS-underlättad provtagning av cellulärt och klonalt proteininnehåll. Utarmning av rikliga äggula proteiner och separation genom kapillärelektrofores (CE) eller nano-flöde vätskekromatografi (LC), förbättrad identifiering (ID) känslighet med hjälp av elektrospray jonisering (ESI) högupplöst masspektrometri (HRMS). Kvantifiering avslöjade dysregulering, vilket gav ny information för hypotesdrivna studier i kombination med information tillgänglig från genontologi (GO). Siffrorna har anpassats med tillstånd från referens15. Klicka här för att se en större version av denna figur.
Protocol
Representative Results
Discussion
Detta protokoll möjliggör karakterisering av proteinuttryck i identifierade cellinjer i embryon av Xenopus-arten. Metoden bygger på HRMS och kombinerar utsökt specificitet i molekylär identifiering, förmåga för multiproteindetektion utan molekylära sonder (vanligtvis hundratals till tusentals olika proteiner) och en förmåga till kvantifiering. Anpassningsförmåga till klassiska verktyg och arbetsflöden inom cell- och utvecklingsbiologi (neuro) utökar HRMS-proteomiken till spännande tillämpningar,…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vi är tacksamma mot Jie Li (University of Maryland, College Park) för värdefulla diskussioner om embryonal dissociation och FACS. Vi tackar Vi M. Quach och Camille Lombard-Banek för hjälp med provberedning och datainsamling i tidigare studier som exemplifierar de proteomiska tillämpningar som lyfts fram i detta protokoll. Delar av detta arbete stöddes av National Science Foundation under prisnummer IOS-1832968 KARRIÄR (till P.N.), National Institutes of Health under prisnummer R35GM124755 (till P.N.), University of Maryland-National Cancer Institute Partnership Program (till PN) och COSMOS Club Foundation forskningspriser (till A.B.B. och L.R.P.).
Materials
| Acetonitrile (LC-MS-grade) | Fisher Scientific | A955 | |
| Agarose | ThermoFisher Scientific | R0492 | |
| Ammonium bicarbonate | Fisher Scientific | A643-500 | |
| Analytical Column | Thermo Scientific | 164941 | |
| Analytical microbalance | Mettler-Toledo | XSE105DU | |
| Automatic peptide fractionation platform | Agilent | 1260 Infinity II | |
| Borosilicate Capillaries | Sutter Instruments Co. | B100-50-10 | |
| Borosilicate Capillaries (for making Emmitters) | Sutter Instruments | B100-75-10 | |
| C18 spin columns (for desalting) | ThermoFisher Scientific | 89870 | |
| Camera ro monitor electrospray | Edmund Optics Inc. | EO-2018C | |
| Combretastatin A4 | Millipore Sigma | C7744 | |
| Commercial CESI system | AB SCIEX | CESI | |
| (Cyclohexylamino)-1-propanesulfonic acid (CAPS) | VWR | 97061-492 | |
| Cytochalasin D | Millipore Sigma | C8273 | |
| Dextran, Alexa Fluor 488; 10,000 MW, Anionic, Fixable | ThermoFisher Scientific | D22910 | |
| Diothiothreitol | Fisher Scientific | FERR0861 | |
| Dumont #5 Forceps | Fine Science Tools | 11252-30 | |
| EDTA | Fisher Scientific | AAJ62786AP | |
| Epifluorescence light source | Lumencore | AURA III | |
| Eppendorf LoBing microcentrifuge tubes: protein | Fisher Scientific | 13-698-793 | |
| Formic acid (LC-MS-grade) | Fisher Scientific | A117-50 | |
| Freezer (-20 °C) | Fisher Scientific | 97-926-1 | |
| Freezer (-80 °C) | Thermo Scientific | TSX40086A | |
| Fused silica capillary | Molex | 1088150596 | |
| Heat Block | Benchmark | BSH300 | |
| High pressure liquid Chromatography System | ThermoFisher Scientific | Dionex Ultimate 3000 RSLC nanosystem | |
| High voltage power supply | Spellman | CZE1000R | |
| High-resolution Mass Spectrometer | ThermoFisher Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | |
| HPLC caps | Thermo Scientific | C4013-40A | |
| HPLC Vials | Thermo Scientific | C4013-11 | |
| Illuminator e.g. Goosenecks | Nikon | C-FLED2 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide | Fisher Scientific | AC122275000 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456-4 | |
| Microcapillary puller | Suttor Instruments | P-2000 | |
| Microinjector | Warner Instrument, Handem, CT | PLI-100A | |
| Micropippette puller | Sutter Instruments Co. | P-1000 | |
| MS data analysis software, commercial | ProteomeDiscoverer | ||
| MS data analysis software, opensource | MaxQuant | ||
| non-idet 40 substitute | Millipore Sigma | 11754599001 | |
| Petri dish 60 mm and 80 mm | Fisher Scientific | S08184 | |
| Pierce 10 µL bed Zip-tips (for desalting) | ThermoFisher Scientific | 87782 | |
| Pierce bicinchoninic acid protein assay kit | ThermoFisher Scientific | 23225 | |
| Pierce quantitative colorimetric peptide assay | ThermoFisher Scientific | 23275 | |
| Pierce Trypsin Protease (MS Grade) | Fisher Scientific | PI90058 | |
| Protein LoBind vials | Eppendorf | 0030108434 , 0030108442 |
|
| Refrigerated Centrifuge | Eppendorf | 5430R | |
| Refrigerated Incubator | Thermo Scientific | PR505755R/3721 | |
| sodium isethionate | Millipore Sigma | 220078 | |
| sodium pyrophosphate | Sigma Aldrich | 221368-100G | |
| Stainless steel BGE vial | Custom-Built | ||
| Stainless steel sample vials | Custom-Built | ||
| Stereomicroscope (objective 10x) | Nikon | SMZ 1270, SZX18 | |
| Sucrose | VWR | 97063-790 | |
| Syringe pumps (2) | Harvard Apparatus | 704506 | |
| Syringes (gas-tight): 500–1000 µL | Hamilton | 1750TTL | |
| Transfer pipettes (Plastic, disposable) | Fisher Scientific | 13-711-7M | |
| Trap Column | Thermo Scientific | 164750 | |
| Tris-HCl (1 M solution) | Fisher Scientific | AAJ22638AP | |
| Vacuum concentrator capable of operation at 4–10 °C | Labconco | 7310022 | |
| Vortex-mixer | Benchmark | BS-VM-1000 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| XYZ translation stage | Thorlabs | PT3 | |
| XYZ translation stage | Custom-Built |
References
- Shoemaker, L. D., Kornblum, H. I. Neural Stem Cells (NSCs) and Proteomics. Molecular & Cellular Proteomics. 15 (2), 344-354 (2016).
- Cervenka, J., et al. Proteomic characterization of human neural stem cells and their secretome during in vitro differentiation. Frontiers in Cellular Neuroscience. 14, 612560 (2021).
- Christian, J. L. Morphogen gradients in development: From form to function. Wiley Interdisciplinary Reviews. Developmental Biology. 1 (1), 3-15 (2012).
- Gurdon, J. B., Elsdale, T. R., M, F. Sexually mature individuals of Xenopus laevis from the transplantation of single somatic nuclei. Nature. 182, 64-65 (1958).
- Harland, R. M., Grainger, R. M. Xenopus research: metamorphosed by genetics and genomics. Trends in Genetics. 27 (12), 507-515 (2011).
- Moody, S. A. Fates of the blastomeres of the 16-cell stage Xenopus embryo. Developmental Biology. 119 (2), 560-578 (1987).
- Moody, S. A. Fates of the blastomeres of the 32-cell stage Xenopus embryo. Developmental Biology. 122 (2), 300-319 (1987).
- Dale, L., Slack, J. M. W. Fate map for the 32-cell stage of Xenopus laevis. Development. 99 (4), 527-551 (1987).
- Sun, L. L., et al. Single cell proteomics using frog (Xenopus laevis) blastomeres isolated from early stage embryos, which form a geometric progression in protein content. Analytical Chemistry. 88 (13), 6653-6657 (2016).
- Lombard-Banek, C., Moody, S. A., Nemes, P. Single-cell mass spectrometry for discovery proteomics: quantifying translational cell heterogeneity in the 16-cell frog (Xenopus) embryo. Angewandte Chemie-International Edition. 55 (7), 2454-2458 (2016).
- Zhang, Y. Y., Fonslow, B. R., Shan, B., Baek, M. C., Yates, J. R. Protein analysis by shotgun/bottom-up proteomics. Chemical Reviews. 113 (4), 2343-2394 (2013).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early development of Xenopus laevis: A laboratory manual. , (2000).
- Briggs, J. A., et al. The dynamics of gene expression in vertebrate embryogenesis at single-cell resolution. Science. 360 (6392), (2018).
- Gupta, M., Sonnett, M., Ryazanova, L., Presler, M., Wuhr, M., Vleminckx, K. Quantitative proteomics of xenopus embryos I, sample preparation. Xenopus. Methods in Molecular Biology. 1865, 175-194 (2018).
- Baxi, A. B., Lombard-Banek, C., Moody, S. A., Nemes, P. Proteomic characterization of the neural ectoderm fated cell clones in the Xenopus laevis embryo by high-resolution mass spectrometry. ACS Chemical Neuroscience. 9 (8), 2064-2073 (2018).
- Moody, S. A. Cell lineage analysis in Xenopus embryos. Methods in Molecular Biology. 135, 331-347 (2000).
- Sater, A. K., Moody, S. A. Using Xenopus to understand human diseases and developmental disorders. Genesis. 55 (1-2), 1-14 (2017).
- Lombard-Banek, C., Choi, S. B., Nemes, P., Allbritton, N. L., Kovarik, M. L. . Enzyme Activity in Single Cells. Methods in Enzymology. 628, 263-292 (2019).
- Lombard-Banek, C., Moody, S. A., Nemes, P. High-sensitivity mass spectrometry for probing gene translation in single embryonic cells in the early frog (Xenopus) embryo. Frontiers in Cell and Developmental Biology. 4, 11 (2016).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. Microprobe capillary electrophoresis mass spectrometry for single-cell metabolomics in live frog (Xenopus laevis) embryos. Journal of Visualized Experiments: JoVE. (130), e56956 (2017).
- Lombard-Banek, C., Moody, S. A., Manzin, M. C., Nemes, P. Microsampling capillary electrophoresis mass spectrometry enables single-cell proteomics in complex tissues: developing cell clones in live Xenopus laevis and zebrafish embryos. Analytical Chemistry. 91 (7), 4797-4805 (2019).
- Klein, S. L. The first cleavage furrow demarcates the dorsal-ventral axis in Xenopus embryos. Developmental Biology. 120 (1), 299-304 (1987).
- Karimi, K., et al. Xenbase: a genomic, epigenomic and transcriptomic model organism database. Nucleic Acids Research. 46 (1), 861-868 (2018).
- Kakebeen, A. D., Chitsazan, A. D., Wills, A. E. Tissue disaggregation and isolation of specific cell types from transgenic Xenopus appendages for transcriptional analysis by FACS. Developmental Dynamics. 250 (9), 1381-1392 (2021).
- Garcia, B. A. What does the future hold for top down mass spectrometry. Journal of the American Society for Mass Spectrometry. 21 (2), 193-202 (2010).
- Toby, T. K., Fornelli, L., Kelleher, N. L. Progress in top-down proteomics and the analysis of proteoforms. Annual Review of Analytical Chemistry. (Palo Alto Calif). 9 (1), 499-519 (2016).
- Zhang, Z. B., Dubiak, K. M., Huber, P. W., Dovichi, N. J. Miniaturized filter-aided sample preparation (MICRO-FASP) method for high throughput, ultrasensitive proteomics sample preparation reveals proteome asymmetry in Xenopus laevis Embryos. Analytical Chemistry. 92 (7), 5554-5560 (2020).
- Wisniewski, J. R., Becher, D. . Microbial Proteomics: Methods and Protocols.Methods in Molecular Biology. 1841, 3-10 (2018).
- Hughes, C. S., et al. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9, 882 (2018).
- Wessel, D., Flugge, U. I. A method for the quantitative recovery of protein in dilute-solution in the presence of detergents and lipids. Analytical Biochemistry. 138 (1), 141-143 (1984).
- Jiang, L., He, L., Fountoulakis, M. Comparison of protein precipitation methods for sample preparation prior to proteomic analysis. Journal of Chromatography A. 1023 (2), 317-320 (2004).
- Hildonen, S., Halvorsen, T. G., Reubsaet, L. Why less is more when generating tryptic peptides in bottom-up proteomics. Proteomics. 14 (17-18), 2031-2041 (2014).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19, 161 (2018).
- Drouin, N., et al. Capillary electrophoresis-mass spectrometry at trial by metabo-ring: effective electrophoretic mobility for reproducible and robust compound annotation. Analytical Chemistry. 92 (20), 14103-14112 (2020).
- Sun, L. L., Zhu, G. J., Zhang, Z. B., Mou, S., Dovichi, N. J. Third-generation electrokinetically pumped sheath-flow nanospray interface with improved stability and sensitivity for automated capillary zone electrophoresis-mass spectrometry analysis of complex proteome digests. Journal of Proteome Research. 14 (5), 2312-2321 (2015).
- DeLaney, K., Sauer, C. S., Vu, N. Q., Li, L. J. Recent advances and new perspectives in capillary electrophoresis-mass spectrometry for single cell "omics". Molecules. 24 (1), 21 (2019).
- Nemes, P., Rubakhin, S. S., Aerts, J. T., Sweedler, J. V. Qualitative and quantitative metabolomic investigation of single neurons by capillary electrophoresis electrospray ionization mass spectrometry. Nature Protocols. 8 (4), 783-799 (2013).
- Choi, S. B., Zamarbide, M., Manzini, M. C., Nemes, P. Tapered-tip capillary electrophoresis nano-electrospray ionization mass spectrometry for ultrasensitive proteomics: the mouse cortex. Journal of the American Society for Mass Spectrometry. 28 (4), 597-607 (2017).
- Pino, L. K., Rose, J., O’Broin, A., Shah, S., Schilling, B. Emerging mass spectrometry-based proteomics methodologies for novel biomedical applications. Biochemical Society Transactions. 48 (5), 1953-1966 (2020).
- Chen, C., Hou, J., Tanner, J. J., Cheng, J. L. Bioinformatics methods for mass spectrometry-based proteomics data analysis. International Journal of Molecular Sciences. 21 (8), 25 (2020).
- Peshkin, L., et al. On the relationship of protein and mRNA dynamics in vertebrate embryonic development. Developmental Cell. 35 (3), 383-394 (2015).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513-2526 (2014).
- Gygi, S. P., et al. Quantitative analysis of complex protein mixtures using isotope-coded affinity tags. Nature Biotechnology. 17 (10), 994-999 (1999).
- Thompson, A., et al. Tandem mass tags: A novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75 (8), 1895-1904 (2003).
- Mi, H. Y., et al. PANTHER version 16: a revised family classification, tree-based classification tool, enhancer regions and extensive api. Nucleic Acids Research. 49, 394-403 (2021).
- Schmidt, E., et al. . On the Move Federated Workshops. , 710-719 (2006).
- Deutsch, E. W., et al. Trans-Proteomic pipeline, a standardized data processing pipeline for large-scale reproducible proteomics informatics. Proteomics Clinical Applications. 9 (7-8), 745-754 (2015).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Demsar, J., et al. Orange: Data mining toolbox in Python. Journal of Machine Learning Research. 14, 2349-2353 (2013).
- Oberg, A. L., Vitek, O. Statistical design of quantitative mass spectrometry-based proteomic experiments. Journal of Proteome Research. 8 (5), 2144-2156 (2009).
- Jensen, L. J., et al. STRING 8 – a global view on proteins and their functional interactions in 630 organisms. Nucleic Acids Research. 37, 412-416 (2009).
- Schweppe, D. K., Huttlin, E. L., Harper, J. W., Gygi, S. P. BioPlex display: an interactive suite for large-scale AP-MS protein-protein interaction data. Journal of Proteome Research. 17 (1), 722-726 (2018).
- Hornbeck, P. V., et al. PhosphoSitePlus, 2014: mutations, PTMs and recalibrations. Nucleic Acids Research. 43, 512-520 (2015).
- Letunic, I., Khedkar, S., Bork, P. SMART: recent updates, new developments and status in 2020. Nucleic Acids Research. 49, 458-460 (2021).
- Lombard-Banek, C., et al. In vivo subcellular mass spectrometry enables proteo-metabolomic single-cell systems biology in a chordate embryo developing to a normally behaving tadpole (X. laevis). Angewandte Chemie-International Edition. 60 (23), 12852-12858 (2021).
- Lombard-Banek, C., Reddy, S., Moody, S. A., Nemes, P. Label-free quantification of proteins in single embryonic cells with neural fate in the cleavage-stage frog (Xenopus laevis) embryo using capillary electrophoresis electrospray ionization high-resolution mass spectrometry (CE-ESI-HRMS). Molecular & Cellular Proteomics. 15 (8), 2756-2768 (2016).
- Saha-Shah, A., et al. Single cell proteomics by data-independent acquisition to study embryonic asymmetry in Xenopus laevis. Analytical Chemistry. 91 (14), 8891-8899 (2019).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. In situ microprobe single-cell capillary electrophoresis mass spectrometry: metabolic reorganization in single differentiating cells in the live vertebrate (Xenopus laevis) embryo. Analytical Chemistry. 89 (13), 7069-7076 (2017).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).

