Un marco de código abierto para el cálculo masivo de moléculas terapéuticas basadas en anticuerpos
Summary
En este artículo se describe el uso de una aplicación de software, mAbScale, para el cálculo de masas para terapias con proteínas basadas en anticuerpos monoclonales.
Abstract
Las masas bioterapéuticas son un medio para verificar la identidad y la integridad estructural. La espectrometría de masas (MS) de proteínas intactas o subunidades proteicas proporciona una herramienta analítica sencilla para las diferentes etapas del desarrollo biofarmacéutico. La identidad de la proteína se confirma cuando la masa experimental de la EM está dentro de un rango de error de masa predefinido de la masa teórica. Si bien existen varias herramientas computacionales para el cálculo de los pesos moleculares de proteínas y péptidos, no fueron diseñadas para su aplicación directa a entidades bioterapéuticas, tienen limitaciones de acceso debido a licencias pagas o requieren cargar secuencias de proteínas en servidores host.
Hemos desarrollado una rutina modular de cálculo de masas que permite determinar fácilmente las masas medias o monoisotópicas y las composiciones elementales de las glicoproteínas terapéuticas, incluidos los anticuerpos monoclonales (mAb), los anticuerpos biespecíficos (bsAb) y los conjugados anticuerpo-fármaco (ADC). La naturaleza modular de este marco de cálculo basado en Python permitirá la extensión de esta plataforma a otras modalidades como vacunas, proteínas de fusión y oligonucleótidos en el futuro, y este marco también podría ser útil para la interrogación de datos de espectrometría de masas de arriba hacia abajo. Al crear una aplicación de escritorio independiente de código abierto con una interfaz gráfica de usuario (GUI), esperamos superar las restricciones de uso en entornos donde la información propietaria no se puede cargar en herramientas basadas en la web. En este artículo se describen los algoritmos y la aplicación de esta herramienta, mAbScale, a diferentes modalidades terapéuticas basadas en anticuerpos.
Introduction
En las últimas dos décadas, la bioterapéutica ha evolucionado hasta convertirse en un pilar de la industria farmacéutica moderna. La pandemia de SARS-CoV2 y otras afecciones potencialmente mortales han aumentado aún más la necesidad de un desarrollo más rápido y amplio de moléculas biofarmacéuticas 1,2,3.
El peso molecular bioterapéutico es crítico para la identificación de la molécula, en combinación con otros ensayos analíticos. Las masas de subunidades intactas y reducidas se utilizan a lo largo de los ciclos de vida de descubrimiento y desarrollo como parte de las estrategias de control destinadas a mantener la calidad, como se describe en el QTPP (Quality Target Product Profile)4.
El desarrollo analítico en la industria biofarmacéutica se basa en gran medida en las mediciones de masa para el análisis de masa intacta y la caracterización profunda mediante el mapeo de péptidos o el monitoreo del método multiatributo (MAM). En el centro de estas técnicas que utilizan plataformas modernas de espectrometría de masas (MS) se encuentra la capacidad de proporcionar mediciones de masa precisas de alta resolución (HR/AM). La mayoría de los instrumentos HR/AM producen precisiones de masa en el rango de 0,5-5 ppm, que se escalan con el rango de masa. La capacidad de medir masas con precisión para moléculas grandes intactas permite la identificación rápida y confiable de terapias de moléculas grandes. Dado que la resolución isotópica no puede alcanzarse utilizando las condiciones experimentales típicas para moléculas grandes (>10 kDa), se deben calcular las masas medias para su comparación e identificación 5,6.
Un espectro típico de masa de proteína intacta o subunidad representa el perfil proteoforme general, que contiene información compuesta sobre las diversas formas moleculares resultantes de las modificaciones postraduccionales (PTM) y cualquier diferencia de estructura primaria, como clips o variantes de secuencia. La naturaleza relativamente fácil y de alto rendimiento de estas mediciones las hace atractivas para la caracterización y como controles de monitoreo durante el proceso 7,8. El análisis de datos para estos experimentos generalmente requiere que el usuario defina el espacio de búsqueda de formas moleculares (rango de PTM u otras formas moleculares). En el caso de las proteínas glicosiladas, este espacio de búsqueda está impulsado en gran medida por la heterogeneidad de los glicoformes. Las combinaciones de múltiples PTM, las configuraciones de enlaces disulfuro y otras variaciones a lo largo de la estructura primaria hacen que el cálculo de todas las formas moleculares posibles sea una tarea tediosa. Por lo tanto, el cálculo manual de las posibles formas moleculares es un proceso que consume tiempo y recursos con un alto potencial de error humano.
Aquí, presentamos una herramienta de cálculo de masa que fue desarrollada considerando las características más importantes de las moléculas bioterapéuticas, tales como mAbs, bsAbs, ADCs, etc. La herramienta permite la fácil incorporación de variables de espacio de búsqueda para el cálculo consistente de masas y composiciones elementales. El carácter modular de esta herramienta permitirá seguir desarrollándola y aplicándola al cálculo de masas y a la adaptación de masas para otras modalidades.
El módulo GUI permite al usuario especificar la entrada para el cálculo de masa, como se muestra en la Figura 1; En concreto, el usuario introduce secuencias de aminoácidos de una sola letra para cadenas de anticuerpos ligeros y pesados. Las modificaciones comunes para la ciclación de N-terminal de cadena pesada y el recorte de lisina C-terminal se incluyen como casillas de verificación. Además, la fórmula química/composición elemental se puede agregar/restar de estas cadenas de proteínas a través del cuadro de texto Chem Mod respectivo. Esto permite al usuario la flexibilidad de agregar una composición elemental que incluye múltiples modificaciones postraduccionales o una carga útil de molécula pequeña en el caso de un ADC. Como la mayoría de los anticuerpos monoclonales terapéuticos están diseñados para eliminar los sitios de glicosilación en la cadena ligera, la glicosilación en la cadena ligera se deja opcional y se puede especificar mediante una casilla de verificación en la interfaz gráfica de usuario.
Una variación típica del análisis de masa intacta para anticuerpos es un análisis de masa de subunidades reducidas, en el que la cadena ligera se separa de la cadena pesada mediante la reducción de los enlaces disulfuro entre cadenas. Dependiendo de la fuerza del agente reductor utilizado, los enlaces disulfuro intracadena pueden o no escindirse. Los usuarios tienen la flexibilidad de introducir el número total de enlaces disulfuro en función del subtipo de IgG o en el caso de unADC 9 conjugado con cisteína.
La aplicación calcula las masas de forma ascendente, en la que las composiciones elementales se calculan primero para las cadenas pesadas y ligeras individuales. A continuación, se tiene en cuenta la ciclación N-terminal de cadena pesada (HC) Lys-clipping ajustando las composiciones elementales calculadas. A continuación, se aplican las modificaciones químicas especificadas a las cadenas pesadas y/o ligeras. Dependiendo del tipo de análisis y de los patrones de enlace disulfuro especificados por el usuario, el número de hidrógenos se ajusta para las dos cadenas polipeptídicas. Las masas de HC glicosilado y de cadena ligera (LC) (opcional) se calculan en función de la entrada del usuario. Finalmente, se combinan múltiples masas HC y LC, y los números de enlace disulfuro se actualizan automáticamente para el cálculo de masa intacta.
Con moléculas más grandes, como proteínas intactas, las masas monoisotópicas no se pueden medir debido al defecto de masa aditivo cuando se utilizan espectrómetros de masas con un poder de resolución típico. En cambio, se miden o informan masas nominales o promedio 5,10,11,12,13. Las masas elementales medias pueden variar en función de la fuente utilizada para las masas seleccionadas14,15. Si bien las diferencias en las masas elementales pueden ser pequeñas, pueden sumar valores significativos para los cálculos de peso molecular de moléculas grandes. Las masas elementales medias utilizadas por defecto en la aplicación de software se muestran en la Tabla Suplementaria 1. Para entornos regulados como el campo de la investigación y el desarrollo (I&D) biofarmacéutico, es importante mantener masas moleculares consistentes porque los cambios en las masas pueden implicar cambios en la entidad molecular durante las presentaciones regulatorias. Para permitir la coherencia en el uso de masas elementales, se incluye un diccionario de masas elementales con la herramienta de software como un archivo de texto de valores separados por comas (csv): Element_Mass.csv (Archivo de codificación suplementario 1). Del mismo modo, se incluye una lista curada de composiciones de glicanos que se ven típicamente en mAbs: Glycan.csv (Supplemental Coding File 2). Ambos archivos se guardan en la misma ubicación de carpeta que una aplicación ejecutable y pueden ser modificados por el usuario para usar una lista de masa elemental específica o una biblioteca de glicanos.
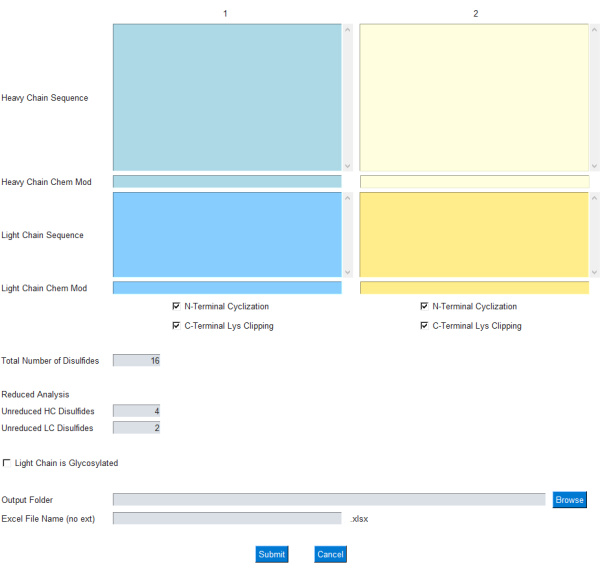
Figura 1: Interfaz gráfica de usuario para la aplicación mAbScale. El módulo GUI permite al usuario especificar la entrada para el cálculo de masa. El usuario introduce secuencias de aminoácidos de una sola letra para las cadenas de anticuerpos ligeros y pesados. Las modificaciones comunes para la ciclación del terminal N de cadena pesada y el recorte de lisina del terminal C se incluyen como casillas de verificación. Las fórmulas químicas/composiciones elementales se pueden sumar/restar a través del cuadro de texto respectivo de Chem Mod . Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
mAbScale proporciona una interfaz de usuario intuitiva con la flexibilidad de alterar los componentes básicos para los cálculos de masa y elementales. Se espera que los usuarios tengan un conocimiento básico de la molécula objetivo para usar la aplicación, derivar masas correctas e interpretar los resultados. Por ejemplo, la hoja de salida de masa intacta o reducida puede ser abrumadora debido a las numerosas filas de masas intactas o reducidas, ya que la base de datos de glicanos predeterminada contiene 88 glicanos…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Los autores agradecen a Robert Schuster por su ayuda con la verificación de datos.
Materials
| Acquity UPLC system | Waters Corp., Milford, MA | N/A | Modular system |
| Antibody-drug conjugate (ADC) | GlaxoSmithKline | N/A | Proprietory molecule |
| BEH 200 SEC column | Waters Corp., Milford, MA | 176003904 | |
| Bispecific mAb | GlaxoSmithKline | N/A | Proprietory molecule |
| Byos | Protein Metrics, Cupertino, CA | https://proteinmetrics.com/byos/ Version 4.5 |
|
| GPMAW | GPMAW | http://www.gpmaw.com/ | |
| LC-MS grade water | Thermo Fisher Scientific, Waltham, MA | W6-1 | |
| mAb standard | Waters Corp., Milford, MA | 186009125 | Waters Humanized mAb Mass Check Standard |
| mAbScale | GlaxoSmithKline | Apache License, Version 2.0 | |
| Xevo G2 Q-TOF mass spectrometer | Waters Corp., Milford, MA | N/A | Modular system |
References
- Reichert, J. M., Valge-Archer, V. E. Development trends for monoclonal antibody cancer therapeutics. Nature Reviews Drug Discovery. 6 (5), 349-356 (2007).
- Kintzing, J. R., Filsinger Interrante, M. V., Cochran, J. R. Emerging strategies for developing next-generation protein therapeutics for cancer treatment. Trends in Pharmacological Sciences. 37 (12), 993-1008 (2016).
- Wang, M. -. Y., et al. SARS-CoV-2: Structure, biology, and structure-based therapeutics development. Frontiers in Cellular and Infection Microbiology. 10, 587269 (2020).
- ICH Q8 (R2) Pharmaceutical Development – Scientific Guideline. European Medicines Agency Available from: https://www.ema.europa.eu/en/ch-q8-r2-pharmaceutical-development-scientific-guideline (2018)
- Donnelly, D. P., et al. Best practices and benchmarks for intact protein analysis for top-down mass spectrometry. Nature Methods. 16 (7), 587-594 (2019).
- Gadgil, H. S., Pipes, G. D., Dillon, T. M., Treuheit, M. J., Bondarenko, P. V. Improving mass accuracy of high performance liquid chromatography/electrospray ionization time-of-flight mass spectrometry of intact antibodies. Journal of the American Society for Mass Spectrometry. 17 (6), 867-872 (2006).
- Beck, A., Sanglier-Cianférani, S., Van Dorsselaer, A. Biosimilar, biobetter, and next generation antibody characterization by mass spectrometry. Analytical Chemistry. 84 (11), 4637-4646 (2012).
- Camperi, J., Goyon, A., Guillarme, D., Zhang, K., Stella, C. Multi-dimensional LC-MS: the next generation characterization of antibody-based therapeutics by unified online bottom-up, middle-up and intact approaches. Analyst. 146 (3), 747-769 (2021).
- Liu, H., May, K. Disulfide bond structures of IgG molecules. mAbs. 4 (1), 17-23 (2012).
- Jakes, C., Füssl, F., Zaborowska, I., Bones, J. Rapid analysis of biotherapeutics using protein a chromatography coupled to orbitrap mass spectrometry. Analytical Chemistry. 93 (40), 13505-13512 (2021).
- Robotham, A. C., Kelly, J. F., Matte, A. Chapter 1 – LC-MS characterization of antibody-based therapeutics: Recent highlights and future prospects. Approaches to the Purification, Analysis and Characterization of Antibody-Based Therapeutics. , 1-33 (2020).
- Valeja, S. G., et al. Unit mass baseline resolution for an intact 148 kDa therapeutic monoclonal antibody by fourier transform ion cyclotron resonance mass spectrometry. Analytical Chemistry. 83 (22), 8391-8395 (2011).
- Fornelli, L., Ayoub, D., Aizikov, K., Beck, A., Tsybin, Y. O. Middle-down analysis of monoclonal antibodies with electron transfer dissociation orbitrap fourier transform mass spectrometry. Analytical Chemistry. 86 (6), 3005-3012 (2014).
- Berglund, M., Wieser, M. E. Isotopic compositions of the elements 2009 (IUPAC Technical Report). Pure and Applied Chemistry. 83 (2), 397-410 (2011).
- Wang, M., et al. The Ame2012 atomic mass evaluation. Chinese Physics C. 36 (12), 1603-2014 (2012).
- Peri, S., Steen, H., Pandey, A. GPMAW–A software tool for analyzing proteins and peptides. Trends in Biochemical Sciences. 26 (11), 687-689 (2001).
- Tipton, J. D., et al. Analysis of intact protein isoforms by mass spectrometry. The Journal of Biological Chemistry. 286 (29), 25451-25458 (2011).
- De Leoz, M. L. A., et al. interlaboratory study on glycosylation analysis of monoclonal antibodies: Comparison of results from diverse analytical methods. Molecular & Cellular Proteomics. 19 (1), 11-30 (2020).
- Cymer, F., Beck, H., Rohde, A., Reusch, D. Therapeutic monoclonal antibody N-glycosylation – Structure, function and therapeutic potential. Biologicals. 52, 1-11 (2018).
- Baker, P. R., Trinidad, J. C., Chalkley, R. J. Modification site localization scoring integrated into a search engine. Molecular & Cellular Proteomics. 10 (7), (2011).
- Chalkley, R. J., Clauser, K. R. Modification site localization scoring: Strategies and performance. Molecular & Cellular Proteomics. 11 (5), 3-14 (2012).

