抗体ベースの治療分子の質量計算のためのオープンソースフレームワーク
Summary
本稿では、モノクローナル抗体ベースのタンパク質治療薬の質量計算のためのソフトウェアアプリケーションmAbScaleの使用について説明します。
Abstract
バイオ医薬品の塊は、同一性と構造的完全性を検証する手段です。インタクトタンパク質またはタンパク質サブユニットの質量分析(MS)は、バイオ医薬品開発のさまざまな段階に容易な分析ツールを提供します。タンパク質の同一性は、MSからの実験質量が理論質量の事前定義された質量誤差範囲内にある場合に確認されます。タンパク質やペプチドの分子量を計算するための計算ツールはいくつか存在しますが、それらはバイオ医薬品企業に直接適用するように設計されていないか、有料ライセンスによるアクセス制限があるか、タンパク質配列をホストサーバーにアップロードする必要があります。
当社は、モノクローナル抗体(mAb)、二重特異性抗体(bsAb)、抗体薬物複合体(ADC)などの治療用糖タンパク質の平均またはモノアイソトピック質量および元素組成を簡単に決定できるモジュール式質量計算ルーチンを開発しました。このPythonベースの計算フレームワークのモジュール性により、将来的にはワクチン、融合タンパク質、オリゴヌクレオチドなどの他のモダリティにもこのプラットフォームを拡張でき、このフレームワークはトップダウン質量分析データの調査にも役立つ可能性があります。グラフィカルユーザーインターフェース(GUI)を備えたオープンソースのスタンドアロンデスクトップアプリケーションを作成することで、Webベースのツールに機密情報をアップロードできない環境での使用に関する制限を克服したいと考えています。本稿では、このツールであるmAbScaleのアルゴリズムと、さまざまな抗体ベースの治療モダリティへの応用について説明します。
Introduction
過去20年間で、バイオ医薬品は現代の製薬業界の主力となるまでに進化しました。SARS-CoV2のパンデミックやその他の生命を脅かす状態により、バイオ医薬品分子のより迅速かつ広範な開発の必要性がさらに高まっています1,2,3。
バイオ医薬品の分子量は、他の分析アッセイと組み合わせて、分子の同定に重要です。インタクトおよび還元されたサブユニット質量は、QTPP(Quality Target Product Profile)4 に記載されているように、品質を維持することを目的とした制御戦略の一環として、創薬および開発ライフサイクル全体を通じて使用されます。
バイオ医薬品業界における分析開発は、インタクト質量分析のための質量測定と、ペプチドマッピングまたはマルチ特性分析法(MAM)モニタリングを用いた詳細な特性評価に大きく依存しています。最新の質量分析(MS)プラットフォームを利用するこれらの技術の中心にあるのは、高分解能の精密質量(HR/AM)測定を提供する機能です。ほとんどのHR/AM装置は、0.5〜5ppmの範囲の質量精度が得られ、質量範囲に応じてスケーリングされます。インタクトな高分子の質量を正確に測定できるため、高分子治療薬を迅速かつ確実に同定できます。高分子(>10 kDa)の一般的な実験条件では同位体分解能を達成できないため、比較と同定のために平均質量を計算する必要があります5,6。
典型的なインタクトまたはサブユニットタンパク質の質量スペクトルは、翻訳後修飾(PTM)から生じるさまざまな分子形態と、クリップや配列バリアントなどの一次構造の違いに関する複合情報を含む、全体的なプロテオフォームプロファイルを表します。これらの測定は比較的簡単でハイスループットなため、特性評価やインプロセスモニタリングコントロールとして魅力的です7,8。これらの実験のデータ解析では、通常、分子形態(PTMまたは他の分子形態の範囲)の探索空間を定義する必要があります。グリコシル化タンパク質の場合、この検索空間は主にグリコフォームの不均一性によって駆動されます。複数のPTM、ジスルフィド結合構成、および一次構造に沿ったその他のバリエーションの組み合わせにより、考えられるすべての分子形態を計算するのは面倒な作業になります。したがって、可能な分子形態の手動計算は、時間とリソースを消費するプロセスであり、人為的ミスの可能性が高くなります。
ここでは、モノクローナル抗体、bsAb、ADCなどのバイオ医薬品分子の最も重要な特徴を考慮して開発された質量計算ツールを紹介します。このツールを使用すると、質量と元素組成の一貫した計算のために探索空間変数を簡単に組み込むことができます。このツールのモジュール性により、さらに開発し、他のモダリティの質量計算や質量マッチングに適用することができます。
GUIモジュールでは、 図1に示すように、質量計算の入力を指定できます。具体的には、ユーザーは軽鎖および重鎖のアミノ酸配列を1文字で入力します。重鎖N末端環化およびC末端リジンクリッピングの一般的な修飾がチェックボックスとして含まれています。さらに、化学式/元素組成は、それぞれの Chem Mod テキストボックスを介してこれらのタンパク質鎖に加算/減算できます。これにより、複数の翻訳後修飾を含む元素組成や、ADCの場合は低分子ペイロードを柔軟に追加できます。ほとんどの治療用モノクローナル抗体は軽鎖のグリコシル化部位を除去するように設計されているため、軽鎖のグリコシル化はオプションであり、GUIのチェックボックスを使用して指定できます。
抗体のインタクト質量分析の典型的なバリエーションは、鎖間ジスルフィド結合を減少させることによって軽鎖を重鎖から分離する還元サブユニット質量分析です。使用される還元剤の強度に応じて、鎖内ジスルフィド結合が切断される場合と切断されない場合があります。ユーザーは、IgGサブタイプに応じて、またはシステイン結合ADC9の場合、ジスルフィド結合の総数を柔軟に入力できます。
このアプリケーションでは、ボトムアップ方式で質量を計算し、元素組成が最初に個々の重鎖と軽鎖について計算されます。次に、計算された元素組成を調整することにより、重鎖(HC)N末端環化Lys-clippingが考慮されます。その後、特定の化学修飾が重鎖および/または軽鎖に適用されます。分析の種類とユーザーが指定したジスルフィド結合パターンに応じて、2つのポリペプチド鎖の水素数が調整されます。グリコシル化HCおよび軽鎖(LC)(オプション)の質量は、ユーザーの入力に基づいて計算されます。最後に、複数の HC 質量と LC 質量が組み合わされ、インタクト質量計算のためにジスルフィド結合数が自動的に更新されます。
インタクトタンパク質などの大きな分子では、一般的な分解能を持つ質量分析計を使用すると、相加的な質量欠陥があるため、モノアイソトピック質量を測定できません。代わりに、公称質量または平均質量が測定または報告されます5,10,11,12,13。平均元素質量は、キュレーション質量に使用されるソースに基づいて変化し得る14,15。元素質量の差は小さいかもしれませんが、高分子分子量の計算では有意な値になることがあります。ソフトウェアアプリケーションでデフォルトで使用される平均元素質量を補足表1に示します。バイオ医薬品の研究開発(R&D)分野のような規制された環境では、質量の変化が規制当局への申請中に分子実体の変化を意味する可能性があるため、一貫した分子質量を維持することが重要です。要素質量の使用に一貫性を持たせるために、要素質量のディクショナリがカンマ区切り値(csv)テキストファイルとしてソフトウェアツールに含まれています:Element_Mass.csv(Supplementary Coding File 1)。同様に、モノクローナル抗体で一般的に見られる糖鎖組成の精選されたリストが含まれています:Glycan.csv(補足コーディングファイル2)。どちらのファイルも実行可能アプリケーションと同じフォルダに保存され、特定の元素質量リストまたは糖鎖ライブラリを使用するようにユーザーが変更できます。
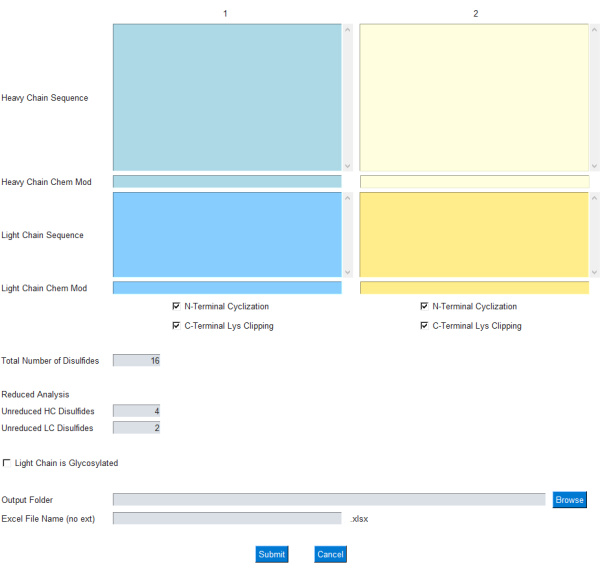
図1:mAbScaleアプリケーションのGUIインターフェース。 GUIモジュールでは、質量計算の入力を指定できます。ユーザーは、軽鎖と重鎖の抗体鎖のアミノ酸配列を1文字で入力します。重鎖N末端環化とC末端リジンクリッピングの一般的な修飾がチェックボックスとして含まれています。化学式/元素組成は、それぞれの ChemMod テキストボックスから追加/削除できます。 この図の拡大版をご覧になるには、ここをクリックしてください。
Protocol
Representative Results
Discussion
mAbScaleは、質量計算と元素計算のビルディングブロックを柔軟に変更できる直感的なユーザーインターフェースを提供します。ユーザーは、アプリケーションを使用し、正しい質量を導き出し、結果を解釈するために、標的分子の基本的な理解を持っていることが期待されます。例えば、デフォルトの糖鎖データベースは、治療用抗体のFc部分に一般的に見出される88個のN-結合型糖鎖を含み?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
著者らは、データ検証に協力してくれたRobert Schuster氏に感謝します。
Materials
| Acquity UPLC system | Waters Corp., Milford, MA | N/A | Modular system |
| Antibody-drug conjugate (ADC) | GlaxoSmithKline | N/A | Proprietory molecule |
| BEH 200 SEC column | Waters Corp., Milford, MA | 176003904 | |
| Bispecific mAb | GlaxoSmithKline | N/A | Proprietory molecule |
| Byos | Protein Metrics, Cupertino, CA | https://proteinmetrics.com/byos/ Version 4.5 |
|
| GPMAW | GPMAW | http://www.gpmaw.com/ | |
| LC-MS grade water | Thermo Fisher Scientific, Waltham, MA | W6-1 | |
| mAb standard | Waters Corp., Milford, MA | 186009125 | Waters Humanized mAb Mass Check Standard |
| mAbScale | GlaxoSmithKline | Apache License, Version 2.0 | |
| Xevo G2 Q-TOF mass spectrometer | Waters Corp., Milford, MA | N/A | Modular system |
References
- Reichert, J. M., Valge-Archer, V. E. Development trends for monoclonal antibody cancer therapeutics. Nature Reviews Drug Discovery. 6 (5), 349-356 (2007).
- Kintzing, J. R., Filsinger Interrante, M. V., Cochran, J. R. Emerging strategies for developing next-generation protein therapeutics for cancer treatment. Trends in Pharmacological Sciences. 37 (12), 993-1008 (2016).
- Wang, M. -. Y., et al. SARS-CoV-2: Structure, biology, and structure-based therapeutics development. Frontiers in Cellular and Infection Microbiology. 10, 587269 (2020).
- ICH Q8 (R2) Pharmaceutical Development – Scientific Guideline. European Medicines Agency Available from: https://www.ema.europa.eu/en/ch-q8-r2-pharmaceutical-development-scientific-guideline (2018)
- Donnelly, D. P., et al. Best practices and benchmarks for intact protein analysis for top-down mass spectrometry. Nature Methods. 16 (7), 587-594 (2019).
- Gadgil, H. S., Pipes, G. D., Dillon, T. M., Treuheit, M. J., Bondarenko, P. V. Improving mass accuracy of high performance liquid chromatography/electrospray ionization time-of-flight mass spectrometry of intact antibodies. Journal of the American Society for Mass Spectrometry. 17 (6), 867-872 (2006).
- Beck, A., Sanglier-Cianférani, S., Van Dorsselaer, A. Biosimilar, biobetter, and next generation antibody characterization by mass spectrometry. Analytical Chemistry. 84 (11), 4637-4646 (2012).
- Camperi, J., Goyon, A., Guillarme, D., Zhang, K., Stella, C. Multi-dimensional LC-MS: the next generation characterization of antibody-based therapeutics by unified online bottom-up, middle-up and intact approaches. Analyst. 146 (3), 747-769 (2021).
- Liu, H., May, K. Disulfide bond structures of IgG molecules. mAbs. 4 (1), 17-23 (2012).
- Jakes, C., Füssl, F., Zaborowska, I., Bones, J. Rapid analysis of biotherapeutics using protein a chromatography coupled to orbitrap mass spectrometry. Analytical Chemistry. 93 (40), 13505-13512 (2021).
- Robotham, A. C., Kelly, J. F., Matte, A. Chapter 1 – LC-MS characterization of antibody-based therapeutics: Recent highlights and future prospects. Approaches to the Purification, Analysis and Characterization of Antibody-Based Therapeutics. , 1-33 (2020).
- Valeja, S. G., et al. Unit mass baseline resolution for an intact 148 kDa therapeutic monoclonal antibody by fourier transform ion cyclotron resonance mass spectrometry. Analytical Chemistry. 83 (22), 8391-8395 (2011).
- Fornelli, L., Ayoub, D., Aizikov, K., Beck, A., Tsybin, Y. O. Middle-down analysis of monoclonal antibodies with electron transfer dissociation orbitrap fourier transform mass spectrometry. Analytical Chemistry. 86 (6), 3005-3012 (2014).
- Berglund, M., Wieser, M. E. Isotopic compositions of the elements 2009 (IUPAC Technical Report). Pure and Applied Chemistry. 83 (2), 397-410 (2011).
- Wang, M., et al. The Ame2012 atomic mass evaluation. Chinese Physics C. 36 (12), 1603-2014 (2012).
- Peri, S., Steen, H., Pandey, A. GPMAW–A software tool for analyzing proteins and peptides. Trends in Biochemical Sciences. 26 (11), 687-689 (2001).
- Tipton, J. D., et al. Analysis of intact protein isoforms by mass spectrometry. The Journal of Biological Chemistry. 286 (29), 25451-25458 (2011).
- De Leoz, M. L. A., et al. interlaboratory study on glycosylation analysis of monoclonal antibodies: Comparison of results from diverse analytical methods. Molecular & Cellular Proteomics. 19 (1), 11-30 (2020).
- Cymer, F., Beck, H., Rohde, A., Reusch, D. Therapeutic monoclonal antibody N-glycosylation – Structure, function and therapeutic potential. Biologicals. 52, 1-11 (2018).
- Baker, P. R., Trinidad, J. C., Chalkley, R. J. Modification site localization scoring integrated into a search engine. Molecular & Cellular Proteomics. 10 (7), (2011).
- Chalkley, R. J., Clauser, K. R. Modification site localization scoring: Strategies and performance. Molecular & Cellular Proteomics. 11 (5), 3-14 (2012).

