Transformation des cellules E. coli en utilisant le chlorure de calcium
English
Share
Overview
Source: Natalia Martin1, Andrew J. Van Alst1, Rhiannon M. LeVeque1, et Victor J. DiRita1
1 Département de microbiologie et de génétique moléculaire, Michigan State University
Les bactéries ont la capacité d’échanger du matériel génétique (acide déoxyribonucléique, ADN) dans un processus connu sous le nom de transfert horizontal de gènes. L’incorporation d’ADN exogène fournit un mécanisme par lequel les bactéries peuvent acquérir de nouveaux traits génétiques qui leur permettent de s’adapter aux conditions environnementales changeantes, telles que la présence d’antibiotiques ou d’anticorps (1) ou de molécules présentes dans les habitats naturels (2). Il existe trois mécanismes de transfert horizontal de gènes : la transformation, la transduction et la conjugaison (3). Ici, nous allons nous concentrer sur la transformation, la capacité des bactéries à prendre l’ADN libre de l’environnement. En laboratoire, le processus de transformation comporte quatre étapes générales : 1) Préparation de cellules compétentes, 2) Incubation de cellules compétentes avec ADN, 3) Récupération des cellules, et 4) Placage des cellules pour la croissance des transformateurs (figure 1).
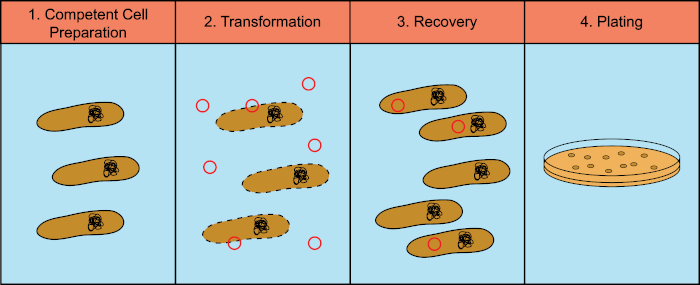
Figure 1 : Étapes générales du processus de transformation. Le processus de transformation comporte quatre étapes générales : 1) Préparation de cellules compétentes, 2) Incubation avec ADN, 3) Récupération des cellules et 4) Cellules de placage pour la croissance des transformateurs.
Pour que la transformation se produise, les bactéries récepteuses doivent être dans un état connu sous le nom de compétence. Certaines bactéries ont la capacité de devenir naturellement compétentes en réponse à certaines conditions environnementales. Cependant, beaucoup d’autres bactéries ne deviennent pas compétentes naturellement, ou les conditions pour ce processus sont encore inconnues. La capacité d’introduire l’ADN dans les bactéries a une gamme d’applications de recherche: pour générer plusieurs copies d’une molécule d’ADN d’intérêt, d’exprimer une grande quantité de protéines, comme un composant dans les procédures de clonage, et d’autres. En raison de la valeur de la transformation à la biologie moléculaire, il existe plusieurs protocoles visant à rendre les cellules artificiellement compétentes lorsque les conditions de compétence naturelle sont inconnues. Deux méthodes principales sont utilisées pour préparer des cellules artificiellement compétentes : 1) par le traitement chimique des cellules et 2) l’exposition des cellules aux impulsions électriques (électroporation). Le premier utilise différents produits chimiques selon la procédure pour créer une attraction entre l’ADN et la surface cellulaire, tandis que le second utilise des champs électriques pour générer des pores dans la membrane cellulaire bactérienne par laquelle les molécules d’ADN peuvent entrer. L’approche la plus efficace pour la compétence chimique est l’incubation avec des cations divalentes, plus particulièrement le calcium (Ca2)(4,5) La compétence induite par le calcium est la procédure qui sera décrite ici (6). Cette méthode est principalement utilisée pour la transformation des bactéries Gram-négatives, et qui sera au centre de ce protocole.
La procédure de transformation chimique implique une série d’étapes dans lesquelles les cellules sont exposées à des cations pour induire la compétence chimique. Ces étapes sont ensuite suivies d’un changement de température – choc thermique – qui favorise l’apport d’ADN étranger par la cellule compétente (7). Les enveloppes de cellules bactériennes sont chargées négativement. Chez les bactéries Gram-négatives comme Escherichia coli, la membrane externe est chargée négativement en raison de la présence de lipopolysaccharide (LPS) (8). Il en résulte la répulsion des molécules d’ADN de même charge négative. Dans l’induction chimique de compétence, les ions de calcium positivement chargés neutralisent cette répulsion de charge permettant l’absorption d’ADN sur la surface de cellules (9). Le traitement du calcium et l’incubation avec de l’ADN se font sur la glace. Par la suite, une incubation à des températures plus élevées (42 oC), le choc thermique, est effectuée. Ce déséquilibre de température favorise encore plus l’apport d’ADN. Les cellules bactériennes doivent être à la phase de croissance mi-exponentielle pour résister au traitement de choc thermique ; dans d’autres stades de croissance, les cellules bactériennes sont trop sensibles à la chaleur, ce qui entraîne une perte de viabilité qui diminue considérablement l’efficacité de la transformation.
Différentes sources d’ADN peuvent être utilisées pour la transformation. Typiquement, les plasmides, petites molécules circulaires d’ADN à double brin, sont utilisés pour la transformation dans la plupart des procédures de laboratoire dans E. coli. Pour que les plasmides soient maintenus dans la cellule bactérienne après la transformation, ils doivent contenir une origine de réplication. Cela leur permet d’être reproduits dans la cellule bactérienne indépendamment du chromosome bactérien. Toutes les cellules bactériennes ne se transforment pas au cours de la procédure de transformation. Ainsi, la transformation donne un mélange de cellules transformées et de cellules non transformées. Pour distinguer entre ces deux populations, une méthode de sélection pour identifier les cellules qui ont acquis le plasmide est utilisée. Les plasmides contiennent habituellement des marqueurs sélectionnables, qui sont des gènes codant un trait qui confère un avantage pour la croissance (c.-à-d. résistance à un antibiotique ou chimique ou sauvetage d’une auotrophie de croissance). Après la transformation, les cellules bactériennes sont plaquées sur des supports sélectifs, ce qui ne permet que la croissance des cellules transformées. Dans le cas des cellules transformées avec un plasmide conférant une résistance à un antibiotique donné, les médias sélectifs seront des supports de croissance contenant cet antibiotique. Plusieurs méthodes différentes peuvent être utilisées pour confirmer que les colonies cultivées dans les milieuis sélectifs sont des transformateurs (c’est-à-dire ont incorporé le plasmide). Par exemple, les plasmides peuvent être récupérés dans ces cellules à l’aide de méthodes de préparation au plasmide (10) et digérés pour confirmer la taille du plasmide. Alternativement, la colonie PCR peut être utilisée pour confirmer la présence du plasmide d’intérêt (11).
L’objectif de cette expérience est de préparer des cellules chimiquement compétentes E. coli DH5MD, en utilisant une adaptation de la procédure de chlorure de calcium (12), et de les transformer avec le plasmide pUC19 pour déterminer l’efficacité de la transformation. La souche E. coli DH5Estest une souche couramment utilisée dans des applications de biologie moléculaire. En raison de son génotype, en particulier le recA1 et endA1, cette souche permet une stabilité accrue de l’insertion et d’améliorer la qualité de l’ADN plasmide dans les préparations ultérieures. Étant donné que l’efficacité de transformation diminue avec l’augmentation de la taille de l’ADN, le plasmide pUC19 a été utilisé dans ce protocole en raison de sa petite taille (2686 bp) (voir https://www.mobitec.com/cms/products/bio/04_vector_sys/standard_cloning_vectors.html pour un carte vectorielle). pUC19 confère une résistance à l’ampicilline et donc, c’était l’antibiotique utilisé pour la sélection.
Procedure
Results
Although TE is dependent on many factors, non-commercial competent cell preparations, like this one, normally yield 106 to 107 transformants per microgram of plasmid. Therefore, this preparation, with a TE = 2.46 x 108 cfu/µg, yielded a TE well beyond the expected range. Additional protocols are available for making supercompetent cells when higher transformation efficiencies are required for a given application (13).
Analysis of the digestion of the plasmid DNA recovered from the transformed cells indicated that this plasmid has the expected size of pUC19 DNA (2686 bp).
Applications and Summary
Transformation is a powerful method for introducing exogenous DNA into bacterial cells that is key to many molecular biology applications in the laboratory. Additionally, it plays a major role in nature by allowing bacterial cells to exchange genetic material that could result in increased genetic variation and allow for acquisition of different beneficial traits for survival under a wide range of conditions. Many bacterial strains encode the genes required for natural competence. However, the conditions in which these genes are induced are still unknown. Further research is required to determine these conditions.
References
- Croucher, N. J. et al. Rapid pneumococcal evolution in response to clinical interventions. Science. 331 (6016):430-434. (2011)
- Borgeaud, S. et al. The type VI secretion system of Vibrio cholerae fosters horizontal gene transfer. Science. 347(6217):63-67. (2015)
- Burmeister, A. R. Horizontal Gene Transfer. Evol Med Public Health. 2015 (1):193-194. (2015)
- Weston A, Brown MG, Perkins HR, Saunders JR, Humphreys GO. Transformation of Escherichia coli with plasmid deoxyribonucleic acid: calcium-induced binding of deoxyribonucleic acid to whole cells and to isolated membrane fractions. J Bacteriol. 145 (2):780-7. (1981)
- Dagert M, Ehrlich SD. Prolonged incubation in calcium chloride improves the competence of Escherichia coli cells. Gene. 6 (1):23-8. (1979)
- Asif A, Mohsin H, Tanvir R, and Rehman Y. Revisiting the Mechanisms Involved in Calcium Chloride Induced Bacterial Transformation. Front Microbiol. 8:2169. (2017)
- Panja S, Aich P, Jana B, Basu T. How does plasmid DNA penetrate cell membranes in artificial transformation process of Escherichia coli? Mol Membr Biol. 25 (5):411-22. (2008)
- Silhavy, TJ, Kahne D, Walker S. The Bacterial Cell Envelope. Cold Spring Harb Perspect Biol. 2 (5): a000414. (2010)
- Panja S, Aich P, Jana B, Basu T. (2008) Plasmid DNA binds to the core oligosaccharide domain of LPS molecules of E. coli cell surface in the CaCl2-mediated transformation process. Biomacromolecules. 9 (9):2501-9.
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Plasmid Purification. JoVE, Cambridge, MA. (2018)
- Bergkessel M and Guthrie C. Colony PCR. Methods in Enzymology. 529: 299-309. (2013)
- Sambrook J and Russell DW. Molecular Cloning A Laboratory Manual. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY.Protocol 25 (1.116-118). (2001)
- Wirth R, Friesenegger A, Fiedler S. Transformation of various species of gram-negative bacteria belonging to 11 different genera by electroporation. Molecular & General Genetics. 216 (1): 175-7. (1989)
Transcript
Bacteria are remarkably adaptable and one mechanism which facilitates this adaptation is their ability to take in external DNA molecules. One type of DNA that bacteria can uptake is called a plasmid, a circular piece of DNA that frequently contains useful information, such as antibiotic resistance genes. The process of bacteria being modified by new genetic information incorporated from an external source is referred to as transformation. Transformation can easily be performed in the laboratory using Escherichia coli, or E. coli.
In order to be transformed, E. coli cells must first be made competent, which means capable of taking in DNA molecules from their environment. The protocol for accomplishing this is surprisingly simple, a short incubation of the cells in a calcium chloride solution. This incubation causes the cells to become permeable to DNA molecules. After the cells are pelleted by centrifugation, the supernatant is removed. The plasmid DNA is now added to the competent cells. After incubating the cells with DNA, the mix is briefly heated to 42 degrees Celsius, followed by rapid cooling on ice. This heat shock causes the DNA to be transferred across the cell’s wall and membranes. The cells are then incubated in fresh media. Then, the bacteria are placed at 37 degrees to allow them to reseal their membranes and express resistant proteins.
Those cells which have taken in the plasmids will faithfully copy the DNA and pass it to their progeny and express any proteins that might be encoded by it, including antibiotic resistance mediators. Those resistance genes can be used as selectable markers to identify bacteria which have been successfully transformed because cells that have not taken up the plasmid will not express the resistance gene product. This means that when the cells are plated on a solid medium which contains the appropriate antibiotic, only cells that have taken up the plasmid will grow. Transformation of the cells in a growing colony can be further confirmed by culturing those cells in liquid media overnight to increase the yield before extracting the DNA from the sample. Once the DNA is isolated, a diagnostic restriction enzyme digest can be carried out. Because restriction enzymes cut DNA in predictable locations, running these digests on a gel should show a predictable pattern if the desired plasmid was successfully transformed. For example, if pUC19 is prepared and cut with the restriction enzyme HindIII, a single band of 2686 nucleotides should be seen on the gel.
In this lab, you will transform E. coli strain DH-5 Alpha with pUC19, and then confirm the successful transformation by DNA gel electrophoresis.
Before starting the procedure, put on the appropriate personal protective equipment, including a lab coat and gloves. Next, sterilize the workspace with 70% ethanol.
Now, prepare chemically competent cells by depositing a loopfull of bacteria onto a sterile LB agar plate and streaking the bacteria with a new loop. Then, incubate the plate at 37 degrees Celsius overnight. The next day, sterilize the bench top with 70% ethanol again, and remove the plate from the incubator.
Inoculate a single, well-isolated colony into 3 milliliters of LB broth in a tube with a sterile loop. Then, grow the culture at 37 degrees Celsius overnight, with shaking at 210 RPM. The next day, measure the optical density of the overnight culture with a spectrophotometer. Then, add 100 milliliters of LB broth to a one-liter flask, and inoculate it with the overnight culture at an optical density of 0. 01. Now, incubate the culture at 37 degrees Celsius with shaking, and check the OD600 every 15 to 20 minutes until the culture reaches mid-exponential growth phase.
After approximately three hours, transfer 50 milliliters of the culture to two ice-cold polypropylene bottles. Then, place the bottles back on ice for 20 minutes to cool. Next, recover the cells via centrifugation. Discard the supernatants and place the bottles upside down on a paper towel. Next, resuspend the bacterial pellet in five milliliters of ice-cold calcium chloride magnesium chloride solution and swirl carefully until the pellet has dissolved completely. Then, add another 25 milliliters of the solution to the dissolved bacterial pellet. Resuspend the other bacterial pellet as previously demonstrated. After this, repeat the centrifugation, and remove the supernatants.
If the competent cells are going to be directly transformed, resuspend each bacterial pellet in two milliliters of an ice-cold 0.1 molar calcium chloride solution by swirling the tubes carefully. To begin the transformation procedure, transfer 50 microliters of competent cells to two labeled 1.5 milliliter polypropylene tubes. Then, add one microliter of pUC19 plasmid DNA to one of the tubes. Mix gently, avoiding bubble formation, and incubate both tubes for 30 minutes on ice. After incubation, transfer the tubes to a heat block and incubate at 42 degrees Celsius for 45 seconds. Immediately transfer the tubes to ice, and incubate for two minutes. Now, add 950 microliters of SOC media to each tube and incubate them for one hour at 37 degrees Celsius to allow the bacteria to recover, and express the antibiotic resistant marker encoded in the plasmid.
To make a 1 to 100 dilution, add 990 microliters of SOC media and 10 microliters of cell suspension to a 1.5 milliliter tube. Then, make a 1 to 10 dilution by adding 900 microliters of SOC media and 100 microliters of cell suspension to a 1.5 milliliter tube. Next, plate 100 microliters of the diluted cell suspensions and 100 microliters of the negative control, onto separate selective plates containing ampicillin using a spreader and incubate the plates at 37 degrees Celsius for 12 to 16 hours. After incubation, count the colony-forming units, or CFUs, per plate, obtained through transformation, and record these data. To verify that the transformants have the pUC19 plasmid, pick a single, well-isolated colony from a plate with a sterile loop, and introduce it to a tube containing 3 milliliters of LB broth. Then, incubate the culture at 37 degrees Celsius with shaking, overnight. The next day, use a DNA mini prep kit to isolate DNA from 3 milliliters of the culture, according to the manufacturer’s instructions. After completing the DNA mini prep, digest the 1 microgram of purified pUC19 with a restriction enzyme at 37 degrees Celsius for 1 hour. Now, load 20 microliters of a molecular weight ladder, 1 microgram of digested plasmid DNA, and 1 microgram of undigested plasmid DNA into consecutive wells of a 1% agarose gel containing 1 microgram per milliliter ethidium bromide. Then, run the gel for 1 hour at 95 volts. Finally, visualize the gel with a UV illuminator.
In this experiment, E. coli DH5 Alpha chemically competent cells were prepared using an adaptation of the calcium chloride procedure, and then transformed with the plasmid pUC19 to determine transformation efficiency. To calculate the transformation efficiency, use the recorded CFU counts for the 1 in 100 and 1 in 10 dilutions, and any other dilutions with CFU counts between 30 and 300. First, the recorded CFU count, 246 in this example, is divided by the amount of DNA, .0001 micrograms here, that was plated. Then, this number is divided by the dilution factor used to give the transformation efficiency in CFUs per microgram. In this example, a 1 to 10 dilution was used and 100 microliters of a 1 milliliter solution was plated, giving a final dilution factor of 0.01. In the undigested plasmid lane, the circular DNA may appear as two or three different bands of varying brightness. This is because the circular, uncut DNA may exist in several different conformation states, such as supercoiled, open circle, or more linear, and each of these move through the gel at different rates. Analysis of the recovered plasmid DNA digestion indicated that the plasmid used has an expected size of pUC19 DNA, 2,686 base pairs.
