적응 염화칼슘 절차를 이용한 E.coli 세포의 형질변환
English
Share
Overview
출처: 나탈리아 마틴1,앤드류 J. 반 알스트1,리안논 M. 레베크1,빅터 J. 디리타1
1 미시간 주립 대학 미생물학 및 분자 유전학학과
박테리아는 수평 유전자 전달로 알려진 과정에서 유전 물질(DeoxyriboNucleic acid, DNA)을 교환할 수 있는 능력을 갖는다. 외인성 DNA를 통합하는 것은 박테리아가 자연적인 서식지에서 발견되는 항생제 또는항체(1)또는 분자의 존재와 같은 변화하는 환경 조건에 적응할 수 있는 새로운 유전적 특성을 획득할 수 있는 메커니즘을제공한다(2). 수평 유전자 전달의 세 가지 메커니즘이 있다: 변환, 변환, 및 컨쥬게이션(3). 여기에서 우리는 변화, 박테리아가 환경에서 무료 DNA를 취할 수있는 능력에 초점을 맞출 것입니다. 실험실에서, 변환 과정은 네 가지 일반적인 단계를 갖는다: 1) 유능한 세포의 준비, 2) DNA를 가진 유능한 세포의 배양, 3) 세포의 회수, 및 4) 변압제의 성장을 위한 세포의 도금 (도 1).
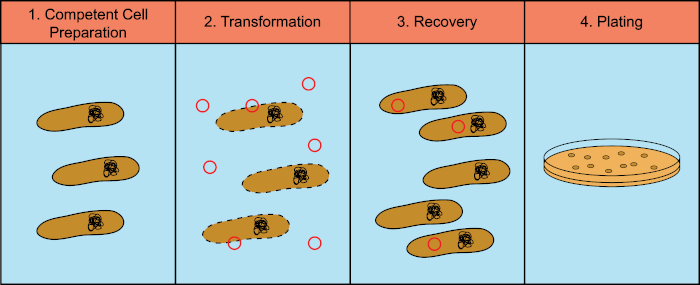
그림 1: 변환 프로세스의 일반적인 단계입니다. 변형 과정에는 4가지 일반적인 단계가 있습니다: 1) 유능한 세포의 준비, 2) DNA를 가진 배양, 3) 세포의 회수 및 4) 변압제의 성장을 위한 도금 세포.
변환이 발생하기 위해, 받는 사람 박테리아는 능력으로 알려진 상태에 있어야합니다. 일부 박테리아는 특정 환경 조건에 대 한 응답으로 자연스럽 게 유능 될 수 있는 능력. 그러나, 많은 다른 박테리아 자연스럽 게 유능한 되지 않습니다., 또는이 과정에 대 한 조건은 아직 알 수 없습니다. 박테리아에 DNA를 소개하는 기능은 연구 응용 프로그램의 범위가 있다: 관심있는 DNA 분자의 여러 사본을 생성, 단백질의 많은 양을 표현하기 위해, 복제 절차의 구성 요소로, 그리고 다른 사람. 분자 생물학으로의 변환의 가치 때문에, 자연적인 역량을 위한 조건이 알려지지 않은 때 세포를 인위적으로 유능하게 하기 위한 몇몇 프로토콜이 있습니다. 두 가지 주요 방법은 인위적으로 유능한 세포를 준비하는 데 사용됩니다 : 1) 세포의 화학 적 처리를 통해 2) 전기 펄스 (전기 포기)에 세포를 노출. 전자는 DNA와 세포 표면 사이 매력을 만들기 위하여 절차에 따라 다른 화학제품을 이용하고, 후자는 DNA 분자가 입력할 수 있는 세균성 세포막에 있는 기공을 생성하기 위하여 전기장을 이용합니다. 화학적 역량에 대한 가장 효율적인 접근법은 이산양이양해, 특히 칼슘(Ca2+)(4,5)칼슘 유도 능력은 여기에 기재될 절차이다(6). 이 방법은 주로 그람 음성 박테리아의 변환에 사용되며, 그것은이 프로토콜의 초점이 될 것입니다.
화학 적 변환의 절차는 세포가 화학 적 능력을 유도하기 위해 양이온에 노출되는 일련의 단계를 포함한다. 이러한 단계는 이어서 온도 변화-열 충격-유능한 세포에 의해 외국 DNA의 섭취를 선호(7). 세균세포 봉투는 음전하로 충전됩니다. 대장균과같은 그람 음성 박테리아에서, 외부 막은 리포폴리사카라이드(LPS)(8)의 존재로 인해음전하된다. 이것은 유사하게 부정적인 충전된 DNA 분자의 반발 귀착됩니다. 화학적 능력 유도에서 양전하 칼슘 이온은 이 전하 반발을 중화시켜 세포 표면에 DNA 흡광도를 가능하게한다(9). DNA를 가진 칼슘 처리 그리고 잠복은 얼음에서 행해지습니다. 그 후, 더 높은 온도 (42°C)에서 인큐베이션, 열 충격이 수행됩니다. 이 온도 불균형은 DNA 섭취를 더 선호합니다. 세균 세포는 열 충격 처리를 견딜 수 있는 중간 기하급수적인 성장 단계에 있을 필요가 있습니다; 다른 성장 단계에서 세균 세포는 열에 너무 민감하여 생존율이 저하되어 변환 효율이 크게 감소합니다.
다른 DNA 소스는 변환을 위해 사용될 수 있습니다. 전형적으로, 플라스미드, 작은 원형, 이중 좌초 DNA 분자는 대장균의대부분의 실험실 절차에서 변형에 사용됩니다. 플라스미드가 변형 후 세균 세포에서 유지되려면 복제의 기원을 포함해야 합니다. 이것은 그(것)들이 세균성 염색체에서 독립적으로 세균성 세포에서 복제될 수 있습니다. 모든 세균 세포가 변형 절차 중에 변형되는 것은 아닙니다. 따라서, 변환은 변형된 세포와 비변형 세포의 혼합물을 산출한다. 이들 두 집단을 구별하기 위해 플라스미드를 획득한 세포를 식별하는 선택 방법이 사용된다. 플라스미드는 일반적으로 성장에 대한 이점을 부여하는 특성을 코딩하는 유전자인 선택 가능한 마커를 포함합니다(즉, 항생제 또는 화학 물질또는 성장 보조요법으로부터의 구조). 변환 후, 세균 세포는 변형 된 세포의 성장을 허용하는 선택적 매체에 도금됩니다. 주어진 항생제에 대한 내성을 부여하는 플라스미드로 변형된 세포의 경우, 선택적 미디어는 그 항생제를 함유하는 성장 매체가 될 것이다. 선택적 매체에서 자란 식민지가 변압제(즉, 플라스미드를 통합한 경우)임을 확인하는 몇 가지 다른 방법을 사용할 수 있다. 예를 들어, 플라스미드는 플라스미드 제제방법(10)을이세포로부터 회수하고 플라스미드 크기를 확인하기 위해 소화될 수 있다. 대안적으로, 콜로니 PCR은 관심의 플라스미드(11)의존재를 확인하는 데 사용할 수 있습니다.
본 실험의 목적은 대장균 DH5α가 화학적으로 유능한 세포를 준비하고, 염화칼슘시술(12)의적응을 이용하여, 플라스미드 pUC19로 변환하여 변환 효율을 결정하는 것이다. 대장균 균주 DH5α는 분자 생물학 응용 분야에서 일반적으로 사용되는 변형입니다. 그것의 유전자형 때문에, 특히 recA1 및 endA1,이 긴장은 증가한 삽입 안정성을 허용하고 후속 준비에 있는 plasmid DNA의 질을 향상합니다. DNA의 크기가 증가함에 따라 변환 효율이 감소하기 때문에, 플라스미드 pUC19는 작은 크기(2686 bp)로 인해 이 프로토콜에 사용되었다(벡터 맵에 대한 https://www.mobitec.com/cms/products/bio/04_vector_sys/standard_cloning_vectors.html 참조). pUC19는 암피실린에 대한 내성을 부여하므로, 이것은 선택에 사용되는 항생제였다.
Procedure
Results
Although TE is dependent on many factors, non-commercial competent cell preparations, like this one, normally yield 106 to 107 transformants per microgram of plasmid. Therefore, this preparation, with a TE = 2.46 x 108 cfu/µg, yielded a TE well beyond the expected range. Additional protocols are available for making supercompetent cells when higher transformation efficiencies are required for a given application (13).
Analysis of the digestion of the plasmid DNA recovered from the transformed cells indicated that this plasmid has the expected size of pUC19 DNA (2686 bp).
Applications and Summary
Transformation is a powerful method for introducing exogenous DNA into bacterial cells that is key to many molecular biology applications in the laboratory. Additionally, it plays a major role in nature by allowing bacterial cells to exchange genetic material that could result in increased genetic variation and allow for acquisition of different beneficial traits for survival under a wide range of conditions. Many bacterial strains encode the genes required for natural competence. However, the conditions in which these genes are induced are still unknown. Further research is required to determine these conditions.
References
- Croucher, N. J. et al. Rapid pneumococcal evolution in response to clinical interventions. Science. 331 (6016):430-434. (2011)
- Borgeaud, S. et al. The type VI secretion system of Vibrio cholerae fosters horizontal gene transfer. Science. 347(6217):63-67. (2015)
- Burmeister, A. R. Horizontal Gene Transfer. Evol Med Public Health. 2015 (1):193-194. (2015)
- Weston A, Brown MG, Perkins HR, Saunders JR, Humphreys GO. Transformation of Escherichia coli with plasmid deoxyribonucleic acid: calcium-induced binding of deoxyribonucleic acid to whole cells and to isolated membrane fractions. J Bacteriol. 145 (2):780-7. (1981)
- Dagert M, Ehrlich SD. Prolonged incubation in calcium chloride improves the competence of Escherichia coli cells. Gene. 6 (1):23-8. (1979)
- Asif A, Mohsin H, Tanvir R, and Rehman Y. Revisiting the Mechanisms Involved in Calcium Chloride Induced Bacterial Transformation. Front Microbiol. 8:2169. (2017)
- Panja S, Aich P, Jana B, Basu T. How does plasmid DNA penetrate cell membranes in artificial transformation process of Escherichia coli? Mol Membr Biol. 25 (5):411-22. (2008)
- Silhavy, TJ, Kahne D, Walker S. The Bacterial Cell Envelope. Cold Spring Harb Perspect Biol. 2 (5): a000414. (2010)
- Panja S, Aich P, Jana B, Basu T. (2008) Plasmid DNA binds to the core oligosaccharide domain of LPS molecules of E. coli cell surface in the CaCl2-mediated transformation process. Biomacromolecules. 9 (9):2501-9.
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Plasmid Purification. JoVE, Cambridge, MA. (2018)
- Bergkessel M and Guthrie C. Colony PCR. Methods in Enzymology. 529: 299-309. (2013)
- Sambrook J and Russell DW. Molecular Cloning A Laboratory Manual. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY.Protocol 25 (1.116-118). (2001)
- Wirth R, Friesenegger A, Fiedler S. Transformation of various species of gram-negative bacteria belonging to 11 different genera by electroporation. Molecular & General Genetics. 216 (1): 175-7. (1989)
Transcript
Bacteria are remarkably adaptable and one mechanism which facilitates this adaptation is their ability to take in external DNA molecules. One type of DNA that bacteria can uptake is called a plasmid, a circular piece of DNA that frequently contains useful information, such as antibiotic resistance genes. The process of bacteria being modified by new genetic information incorporated from an external source is referred to as transformation. Transformation can easily be performed in the laboratory using Escherichia coli, or E. coli.
In order to be transformed, E. coli cells must first be made competent, which means capable of taking in DNA molecules from their environment. The protocol for accomplishing this is surprisingly simple, a short incubation of the cells in a calcium chloride solution. This incubation causes the cells to become permeable to DNA molecules. After the cells are pelleted by centrifugation, the supernatant is removed. The plasmid DNA is now added to the competent cells. After incubating the cells with DNA, the mix is briefly heated to 42 degrees Celsius, followed by rapid cooling on ice. This heat shock causes the DNA to be transferred across the cell’s wall and membranes. The cells are then incubated in fresh media. Then, the bacteria are placed at 37 degrees to allow them to reseal their membranes and express resistant proteins.
Those cells which have taken in the plasmids will faithfully copy the DNA and pass it to their progeny and express any proteins that might be encoded by it, including antibiotic resistance mediators. Those resistance genes can be used as selectable markers to identify bacteria which have been successfully transformed because cells that have not taken up the plasmid will not express the resistance gene product. This means that when the cells are plated on a solid medium which contains the appropriate antibiotic, only cells that have taken up the plasmid will grow. Transformation of the cells in a growing colony can be further confirmed by culturing those cells in liquid media overnight to increase the yield before extracting the DNA from the sample. Once the DNA is isolated, a diagnostic restriction enzyme digest can be carried out. Because restriction enzymes cut DNA in predictable locations, running these digests on a gel should show a predictable pattern if the desired plasmid was successfully transformed. For example, if pUC19 is prepared and cut with the restriction enzyme HindIII, a single band of 2686 nucleotides should be seen on the gel.
In this lab, you will transform E. coli strain DH-5 Alpha with pUC19, and then confirm the successful transformation by DNA gel electrophoresis.
Before starting the procedure, put on the appropriate personal protective equipment, including a lab coat and gloves. Next, sterilize the workspace with 70% ethanol.
Now, prepare chemically competent cells by depositing a loopfull of bacteria onto a sterile LB agar plate and streaking the bacteria with a new loop. Then, incubate the plate at 37 degrees Celsius overnight. The next day, sterilize the bench top with 70% ethanol again, and remove the plate from the incubator.
Inoculate a single, well-isolated colony into 3 milliliters of LB broth in a tube with a sterile loop. Then, grow the culture at 37 degrees Celsius overnight, with shaking at 210 RPM. The next day, measure the optical density of the overnight culture with a spectrophotometer. Then, add 100 milliliters of LB broth to a one-liter flask, and inoculate it with the overnight culture at an optical density of 0. 01. Now, incubate the culture at 37 degrees Celsius with shaking, and check the OD600 every 15 to 20 minutes until the culture reaches mid-exponential growth phase.
After approximately three hours, transfer 50 milliliters of the culture to two ice-cold polypropylene bottles. Then, place the bottles back on ice for 20 minutes to cool. Next, recover the cells via centrifugation. Discard the supernatants and place the bottles upside down on a paper towel. Next, resuspend the bacterial pellet in five milliliters of ice-cold calcium chloride magnesium chloride solution and swirl carefully until the pellet has dissolved completely. Then, add another 25 milliliters of the solution to the dissolved bacterial pellet. Resuspend the other bacterial pellet as previously demonstrated. After this, repeat the centrifugation, and remove the supernatants.
If the competent cells are going to be directly transformed, resuspend each bacterial pellet in two milliliters of an ice-cold 0.1 molar calcium chloride solution by swirling the tubes carefully. To begin the transformation procedure, transfer 50 microliters of competent cells to two labeled 1.5 milliliter polypropylene tubes. Then, add one microliter of pUC19 plasmid DNA to one of the tubes. Mix gently, avoiding bubble formation, and incubate both tubes for 30 minutes on ice. After incubation, transfer the tubes to a heat block and incubate at 42 degrees Celsius for 45 seconds. Immediately transfer the tubes to ice, and incubate for two minutes. Now, add 950 microliters of SOC media to each tube and incubate them for one hour at 37 degrees Celsius to allow the bacteria to recover, and express the antibiotic resistant marker encoded in the plasmid.
To make a 1 to 100 dilution, add 990 microliters of SOC media and 10 microliters of cell suspension to a 1.5 milliliter tube. Then, make a 1 to 10 dilution by adding 900 microliters of SOC media and 100 microliters of cell suspension to a 1.5 milliliter tube. Next, plate 100 microliters of the diluted cell suspensions and 100 microliters of the negative control, onto separate selective plates containing ampicillin using a spreader and incubate the plates at 37 degrees Celsius for 12 to 16 hours. After incubation, count the colony-forming units, or CFUs, per plate, obtained through transformation, and record these data. To verify that the transformants have the pUC19 plasmid, pick a single, well-isolated colony from a plate with a sterile loop, and introduce it to a tube containing 3 milliliters of LB broth. Then, incubate the culture at 37 degrees Celsius with shaking, overnight. The next day, use a DNA mini prep kit to isolate DNA from 3 milliliters of the culture, according to the manufacturer’s instructions. After completing the DNA mini prep, digest the 1 microgram of purified pUC19 with a restriction enzyme at 37 degrees Celsius for 1 hour. Now, load 20 microliters of a molecular weight ladder, 1 microgram of digested plasmid DNA, and 1 microgram of undigested plasmid DNA into consecutive wells of a 1% agarose gel containing 1 microgram per milliliter ethidium bromide. Then, run the gel for 1 hour at 95 volts. Finally, visualize the gel with a UV illuminator.
In this experiment, E. coli DH5 Alpha chemically competent cells were prepared using an adaptation of the calcium chloride procedure, and then transformed with the plasmid pUC19 to determine transformation efficiency. To calculate the transformation efficiency, use the recorded CFU counts for the 1 in 100 and 1 in 10 dilutions, and any other dilutions with CFU counts between 30 and 300. First, the recorded CFU count, 246 in this example, is divided by the amount of DNA, .0001 micrograms here, that was plated. Then, this number is divided by the dilution factor used to give the transformation efficiency in CFUs per microgram. In this example, a 1 to 10 dilution was used and 100 microliters of a 1 milliliter solution was plated, giving a final dilution factor of 0.01. In the undigested plasmid lane, the circular DNA may appear as two or three different bands of varying brightness. This is because the circular, uncut DNA may exist in several different conformation states, such as supercoiled, open circle, or more linear, and each of these move through the gel at different rates. Analysis of the recovered plasmid DNA digestion indicated that the plasmid used has an expected size of pUC19 DNA, 2,686 base pairs.
