Het analyseren van Protein Dynamics gebruik van waterstof Exchange Massaspectrometrie
Summary
Eiwitconformatie en dynamiek zijn de sleutel tot het begrijpen van de relatie tussen eiwit structuur en functie. Waterstofwisselproces combinatie met hoge-resolutie massaspectrometrie is een veelzijdige methode om de conformationele dynamica van proteïnen en karakteriseren eiwit-ligand en eiwit-eiwit interacties, zoals contact interfaces en allosterische effecten.
Abstract
Alle cellulaire processen afhankelijk van de functionaliteit van eiwitten. Hoewel de functionaliteit van een bepaald eiwit is het rechtstreekse gevolg van de unieke aminozuursequentie wordt alleen gerealiseerd door het vouwen van de polypeptideketen tot een gedefinieerde driedimensionale opstelling of meer algemeen in een ensemble van interconverting conformaties. Onderzoek naar het verband tussen eiwit conformatie en functionaliteit is dus essentieel voor een volledig begrip van hoe eiwitten in staat zijn hun grote verscheidenheid aan taken vervullen. Een mogelijkheid om vormveranderingen een eiwit ondergaat tijdens het doorlopen van de functionele cyclus te bestuderen waterstof-1 H / 2 H-uitwisseling in combinatie met hoge-resolutie massaspectrometrie (HX-MS). HX-MS is een veelzijdige en robuuste methode die een nieuwe dimensie toevoegt aan structurele informatie verkregen door bijvoorbeeld kristallografie. Het wordt gebruikt om eiwitten te bestuderen en uitklappen binding van kleine molecule liganden, eiwit-eiwit interacties, conformationele veranderingen in verband met katalyse enzym en allosterie. Bovendien wordt HX-MS vaak gebruikt wanneer de hoeveelheid eiwit zeer beperkt of kristallisatie van het eiwit niet haalbaar. Hier bieden wij een algemeen protocol voor het bestuderen eiwitdynamica met HX-MS en beschrijven als een voorbeeld hoe de interactie grensvlak van twee eiwitten in een complex te onthullen.
Introduction
Het aantal kristalstructuren van eiwitten en eiwitcomplexen afgelopen jaren snel gestegen. Zij presenteren waardevolle momentopnamen van de structurele organisatie van deze eiwitten en vormen een basis voor de structuur-functie analyse. De dynamiek van eiwitten en conformationele veranderingen, die essentieel zijn voor hun functies, zelden onthuld door röntgenkristallografie. Cryo-elektronenmicroscopie, daarentegen, kan eiwitten en eiwitcomplexen vangen in verschillende conformaties, maar algemeen niet beneden oplossen conformationele veranderingen in secondaire structuur niveau 1. Conformationele dynamica van eiwitten in oplossing bij atomaire gegevens kunnen alleen worden opgelost door NMR, maar deze methode is gelimiteerd tot eiwitten relatief kleine grootte (gewoonlijk ≤ 30 kDa) en heeft hoge concentraties van eiwitten (≥ 100 uM), die experimenten belemmert met oligomerisatie of aggregatie gevoelig eiwitten 2. Een methode dieis in staat om een brug tussen hoge-resolutie X-ray kristallografie en cryo-elektronenmicroscopie en die wordt niet beperkt door proteïne grootte of concentratie amide waterstof-1 H / 2 H-uitwisseling (HX) in combinatie met massaspectrometrie (MS). De laatste jaren methode ontwikkeld om een waardevol analytisch instrument voor de analyse van eiwit dynamica, eiwitvouwing, eiwitstabiliteit en conformatieveranderingen 3-5. De moleculaire basis van deze methode is de labiele aard ruggengraat amide waterstof in eiwitten, die wisselen met deuterium atomen wanneer het eiwit in een D2O oplossing. De latere toename van eiwitmassa loop van de tijd wordt gemeten met een hoge-resolutie MS.
Kortom ongestructureerde peptiden HX alleen afhankelijk van de temperatuur, katalysatorconcentratie (OH -, H 3 O + dwz pH, zie figuur 3) en aminozuur-zijketens van aangrenzende residuen door inductieve, katkatalytische en sterische effecten. Deze effecten op de intrinsieke chemische wisselkoers k ch zijn elegant gekwantificeerd door Bai et al.. 6 en een programma beschikbaar is (met dank Z. Zhang), die k l berekent voor elk aminozuur in een polypeptide afhankelijk van pH en temperatuur. Bij neutrale pH en omgevingstemperaturen k l ligt in de orde van 10 1 -10 3 sec -1. In opgevouwen eiwitten HX kan 2-9 ordes van grootte trager vooral door waterstofbinding in de secundaire structuur en in mindere mate door beperkte toegang gehydrateerde OH – ionen om het interieur van een strak gevouwen eiwit. HX in natieve eiwitten impliceert dus gedeeltelijke of globale ontvouwen, chemische uitwisseling en opnieuw vouwen om de natuurlijke toestand volgens vergelijking (1) en de waargenomen wisselkoersen k obs afhangen van de opening tarief k op, de slotkoers k cl en de intrinsieke chemische uitwisseling rate k ch volgens vergelijking (2).
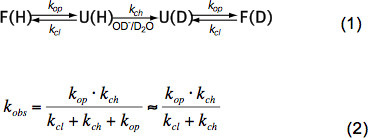
Onder natuurlijke toestand omstandigheden k op veel kleiner is dan k l en kan worden verwaarloosd in de noemer. Er zijn twee extreme uitwisseling regimes genoemd EX1 en EX2. Als de k cl is veel kleiner dan k l (EX1) is het waargenomen aantal is nagenoeg gelijk aan de opening tarief en HX staat directe observatie van het ontvouwen van een structureel element. Een dergelijke uitwisseling regime, waar alle amide protonen uitwisseling in een keer bij het openen van het structurele element, is gemakkelijk in MS door een bimodale verdeling van de isotoop pieken 7. Als k cl veel groter is dan k l (EX2) de waargenomen evenredig is met k ch waarbij de evenredigheidsconstante is gelijk aan het vouwen ontvouwen evenwichten k u = k op </sub> / K cl. Onder deze omstandigheden veel openen en sluiten gebeurtenissen nodig voordat alle amide protonen ruil voor deuteronen, leiden tot een geleidelijke stijging van de gemiddelde massa terwijl de isotopische verdeling blijft ongeveer gelijk. Het regime EX2 maakt de bepaling van de vrije energie van ontvouwing ΔG u en derhalve de stabiliteit van een constructiedeel. Onder natuurlijke toestand staat de EX2 regime is het meest gebruikelijk. Verhoging van de pH en de toevoeging van chaotrope middelen kan de uitwisseling mechanisme verschuiven naar EX1. Daarom kan HX-MS worden gebruikt thermodynamische staand en kinetische parameters van eiwitvouwing en conformatieveranderingen.
Zoals hierboven vermeld HX intrinsiek pH en temperatuur afhankelijke en uitwisseling halfwaardetijd van een geheel aan oplosmiddel blootgestelde proton van de hoofdketen amidegroep tussen 5-400 msec bij fysiologische pH (pH 7,6) en 30 ° C, maar 10 minuten naar> 15 uur met een gemiddelde van> 2 uur bij pH 2,9 en 0 °C (behalve het proton van de eerste ruggengraat amidebinding van een polypeptide, die wisselt met een halfwaardetijd van ca.. 1-2 min). Onder dergelijke trage uitwisseling omstandigheden is het mogelijk om het monster te verteren met proteasen (bijv. pepsine) die actief zijn onder deze omstandigheden, met uit verlies van alle in de opgenomen deuteronen gegevens. Sinds de introductie van peptische digestie onder langzaam uitwisselen omstandigheden kan niet alleen de totale HX kinetiek van full-length eiwitten geanalyseerd maar HX kan worden gelokaliseerd op specifieke gebieden 8,9. Ruimtelijke resolutie is momenteel beperkt tot de omvang van de peptische fragmenten, die in het algemeen tussen 10-30 residuen. Kan echter overlappende fragmenten te wijten aan de niet-specifieke aard van splitsing in pepsine tot een toename van ruimtelijke resolutie. Bovendien werden verscheidene andere proteasen actief bevonden onder quench omstandigheden echter veel minder efficiënt dan pepsine 10. Verdere increase van ruimtelijke resolutie kan worden bereikt door versnippering van peptiden in de gasfase door methoden die de deutereringsgraad patroon zoals electron capture dissociatie (ECD), elektron overdracht dissociatie (ETD) en infrarood multiphoton dissociatie (IRMPD) 11-13 bewaard. Deze technieken Teneinde het verlies van ruimtelijke resolutie door intramoleculaire proton migratie ("scrambling"), die wordt waargenomen door botsing geïnduceerde dissociatie (CID) de meest gebruikte techniek fragmentatie. Deze werkwijzen vereisen optimalisatie voor elke individuele peptide en is dus nog steeds zeer uitdagend.
HX-MS is gebruikt om eiwit-ligand en eiwit-eiwit interacties van virale capside assemblage 14-17 analyseren. Eiwit ontvouwen en opnieuw vouwen en de temperatuur geïnduceerde conformationele veranderingen werden onderzocht 7,18,19. Fosforylering en enkel aminozuur-mutatie-gerelateerde conformatie verandert 16,20 en nucleotide-geïnduceerde veranderingen werden geanalyseerd 21,22. Daarom lijkt deze methode bij uitstek geschikt voor montage en dynamica van moleculaire machines te analyseren. Een aantrekkelijke kandidaat, wiens mechanisme is van groot algemeen belang, is de Hsp90 chaperonne complex.
Protocol
Representative Results
Discussion
Binding van een interactie partner aan een eiwit onvermijdelijk veroorzaakt veranderingen in solvent toegankelijkheid op de bindingsplaats. Bovendien zijn veel eiwitten ondergaan dynamische conformatieveranderingen na binding die andere regio beïnvloeden dan de werkelijke bindende interface. HX-MS is een robuuste methode om deze veranderingen te monitoren en is zelfs in staat om het openbaren van conformationele veranderingen in eiwitten op tijdschalen die andere methoden niet kunnen dekken.
<p class="jove_content"…Disclosures
The authors have nothing to disclose.
Acknowledgements
Wij danken M. Boysen voor commentaar op het manuscript. Dit project werd gefinancierd door de Deutsche Forschungsgemeinschaft (SFB638 en MA 1278/4-1 te MPM en Cluster of Excellence: CellNetworks EXC 81/1). MPM is onderzoeker van de Cluster of Excellence: CellNetworks.
Materials
| Reagent/Equipment | Company | Catalogue number | Comments (optional) |
| maXis QTOF | Bruker | ||
| nanoAcquity UPLC | Waters Corp. | ||
| Shimadzu 10AD-VP | Shimadzu | ||
| 6-port Valve EPC6W with microelectric actuator | Valco | #EPC6W | |
| Injection valve (manual) | Rheodyne | #7725 | |
| Poros AL20 media | Applied Biosystems | #1-6029-06 | |
| Poros R2 | Applied Biosystems | #1-1118-02 | |
| Pepsin | Sigma | #P6887 | use fresh pepsin |
| Microbore (1 mm) | IDEX | #C-128 | |
| Microbore (2 mm) | IDEX | #C-130B | |
| Acquity UPLC BEH C8 Column | Waters Corp. | #186002876 | |
| Thermomixer | Eppendorf | #5355000.011 | |
| Tubing (various diameters) | IDEX | ||
| Fittings | IDEX | #PK-110 with PK-100 |
References
- Saibil, H. R. Conformational changes studied by cryo-electron microscopy. Nat. Struct. Biol. 7 (9), 711-714 (2000).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Hoofnagle, A. N., Resing, K. A., Ahn, N. G. Protein analysis by hydrogen exchange mass spectrometry. Annu. Rev. Biophys. Biomol. Struct. 32, 1-25 (2003).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass. Spectrom. Rev. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. -. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chem Soc. Rev. 40 (3), 1224-1210 (2011).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Rist, W., Jørgensen, T. J. D., Roepstorff, P., Bukau, B., Mayer, M. P. Mapping temperature-induced conformational changes in the Escherichia coli heat shock transcription factor sigma 32 by amide hydrogen exchange. The Journal of biological chemistry. 278 (51), 51415-51421 (1074).
- Englander, J. J., Rogero, J. R., Englander, S. W. Protein hydrogen exchange studied by the fragment separation method. Anal Biochem. 147 (1), 234-244 (1985).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Cravello, L., Lascoux, D., Forest, E. Use of different proteases working in acidic conditions to improve sequence coverage and resolution in hydrogen/deuterium exchange of large proteins. Rapid Commun. Mass Spectrom. : RCM. 17 (21), 2387-2393 (2003).
- Rand, K. D., Zehl, M., Jensen, O. N., Jørgensen, T. J. D. Protein hydrogen exchange measured at single-residue resolution by electron transfer dissociation mass spectrometry. Anal chem. 81 (14), 5577-5584 (2009).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Electron capture dissociation of electrosprayed protein ions for spatially resolved hydrogen exchange measurements. J. Am. Chem. Soc. 130 (35), 11574-11575 (2008).
- Yamada, N., Suzuki, E. -. I., Hirayama, K. Identification of the interface of a large protein-protein complex using H/D exchange and Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 16 (4), 293-299 (2002).
- Lee, T., Hoofnagle, A. N., et al. Docking motif interactions in MAP kinases revealed by hydrogen exchange mass spectrometry. Mol. Cell. 14 (1), 43-55 (2004).
- Hasan, A., Smith, D. L., Smith, J. B. Alpha-crystallin regions affected by adenosine 5′-triphosphate identified by hydrogen-deuterium exchange. Biochem. 41 (52), 15876-15882 (2002).
- Lanman, J., Lam, T. T., Emmett, M. R., Marshall, A. G., Sakalian, M., Prevelige, P. E. Key interactions in HIV-1 maturation identified by hydrogen-deuterium exchange. Nat. Struct. Mol. Biol. 11 (7), 676-677 (2004).
- Wang, L., Lane, L. C., Smith, D. L. Detecting structural changes in viral capsids by hydrogen exchange and mass spectrometry. Protein Sci. 10 (6), 1234-1243 (2001).
- Pan, H., Raza, A. S., Smith, D. L. Equilibrium and kinetic folding of rabbit muscle triosephosphate isomerase by hydrogen exchange mass spectrometry. J. Mol. Biol. 336 (5), 1251-1263 (2004).
- Mazon, H., Marcillat, O., Forest, E., Smith, D. L., Vial, C. Conformational dynamics of the GdmHCl-induced molten globule state of creatine kinase monitored by hydrogen exchange and mass spectrometry. Biochem. 43 (17), 5045-5054 (2004).
- Lanman, J., Lam, T. T., et al. Identification of novel interactions in HIV-1 capsid protein assembly by high-resolution mass spectrometry. J. Mol. Biol. 325 (4), 759-772 (2003).
- Rist, W., Graf, C., Bukau, B., Mayer, M. P. Amide hydrogen exchange reveals conformational changes in hsp70 chaperones important for allosteric regulation. J. Biol. chem. 281 (24), 16493-16501 (2006).
- Graf, C., Stankiewicz, M., Kramer, G., Mayer, M. P. Spatially and kinetically resolved changes in the conformational dynamics of the Hsp90 chaperone machine. EMBO J. 28 (5), 602-613 (2009).
- Lee, C. -. T., Graf, C., Mayer, F. J., Richter, S. M., Mayer, M. P. Dynamics of the regulation of Hsp90 by the co-chaperone Sti1. EMBO J. 31 (6), 1518-1528 (2012).
- Lou, X., Kirchner, M., et al. Deuteration distribution estimation with improved sequence coverage for HX/MS experiments. Bioinformatics. 26 (12), 1535-1541 (2010).
- Kreshuk, A., Stankiewicz, M., Lou, X., Kirchner, M., Hamprecht, F. A., Mayer, M. P. Automated detection and analysis of bimodal isotope peak distributions in H/D exchange mass spectrometry using HeXicon. Intl. J. mass spectrom. 302 (1-3), 125-131 (2011).
- Walters, B. T., Ricciuti, A., Mayne, L., Englander, S. W. Minimizing back exchange in the hydrogen exchange-mass spectrometry experiment. J. Am. Soc. Mass Spectrom. 23 (12), 2132-2139 (2012).
- Weis, D. D., Wales, T. E., Engen, J. R., Hotchko, M., Ten Eyck, L. F. Identification and characterization of EX1 kinetics in H/D exchange mass spectrometry by peak width analysis. J. Am. Soc. Mass Spectrom. 17 (11), 1498-1509 (2006).
- Fang, J., Rand, K. D., Beuning, P. J., Engen, J. R. False EX1 signatures caused by sample carryover during HX MS analyses. Intl. J. Mass Spectrom. 302 (1-3), 19-25 (2011).
- Deng, Y., Smith, D. L. Identification of unfolding domains in large proteins by their unfolding rates. Biochem. 37 (18), 6256-6262 (1998).
- Wales, T. E., Engen, J. R. Partial unfolding of diverse SH3 domains on a wide timescale. J. Mol. Biol. 357 (5), 1592-1604 (2006).
- Pan, Y., Piyadasa, H., O’Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by h/d exchange and covalent labeling: the glycerol facilitator. J. Mol. Biol. 416 (3), 400-413 (2012).
- Hebling, C. M., Morgan, C. R., Stafford, D. W., Jorgenson, J. W., Rand, K. D., Engen, J. R. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Anal chem. 82 (13), 5415-5419 (2010).
- Abzalimov, R. R., Kaplan, D. A., Easterling, M. L., Kaltashov, I. A. Protein conformations can be probed in top-down HDX MS experiments utilizing electron transfer dissociation of protein ions without hydrogen scrambling. J. Am. Soc. Mass Spectrom. 20 (8), 1514-1517 (2009).

