El análisis de dinámica de proteínas utilizando hidrógeno Intercambio de Espectrometría de Masas
Summary
Conformación de la proteína y la dinámica son claves para entender la relación entre estructura y función de proteínas. Intercambio de hidrógeno acoplada a espectrometría de masas de alta resolución es un método versátil para estudiar la dinámica conformacionales de proteínas, así como la caracterización de la proteína-ligando y proteína-proteína interacciones, incluidas las interfaces de contacto y los efectos alostéricos.
Abstract
Todos los procesos celulares dependen de la funcionalidad de las proteínas. Aunque la funcionalidad de una proteína dada es la consecuencia directa de su secuencia de aminoácidos única, que sólo se realiza por el plegamiento de la cadena de polipéptido en una única disposición tridimensional definido o más comúnmente en un conjunto de conformaciones interconverting. La investigación de la relación entre la conformación de la proteína y su función es, por tanto, esencial para una comprensión completa de cómo las proteínas son capaces de cumplir con su gran variedad de tareas. Una posibilidad para estudiar los cambios conformacionales una proteína experimenta mientras avanza a través de su ciclo funcional es hidrógeno H-1/2 H-intercambio en combinación con espectrometría de masas de alta resolución (HX-MS). HX-EM es un método versátil y robusta que añade una nueva dimensión a la información estructural obtenida por ejemplo, cristalografía. Se utiliza para estudiar el plegamiento de proteínas y el despliegue, la unión de pequeña molligandos ecule, las interacciones proteína-proteína, cambios conformacionales ligados a la catálisis enzimática y allostery. Además, HX-MS se utiliza a menudo cuando la cantidad de proteína es muy limitada o la cristalización de la proteína no es factible. Aquí les ofrecemos un protocolo general para el estudio de la dinámica de proteínas con HX-MS y describimos como un ejemplo de cómo mostrar la interfaz de interacción de dos proteínas en un complejo.
Introduction
El número de estructuras cristalinas de proteínas y complejos de proteínas aumentó rápidamente en los últimos años. Presentan instantáneas invaluables de la organización estructural de estas proteínas y proporcionan una base para el análisis de la estructura-función. Sin embargo, la dinámica de las proteínas y los cambios conformacionales, que son esenciales para sus funciones, rara vez se revelaron mediante cristalografía de rayos X. Cryo-microscopia electrónica, por otro lado, es capaz de capturar complejos de proteínas y de proteínas en diferentes conformaciones, pero en general no puede resolver cambios conformacionales a nivel de la estructura secundaria 1. Dinámica conformacional de proteínas en solución a detalles atómicos sólo pueden ser resueltas por RMN, pero este método todavía se limita a proteínas de tamaños relativamente pequeños (generalmente ≤ 30 kDa) y las necesidades de altas concentraciones de proteínas (≥ 100 micras), lo que dificulta experimentos con oligomerización o de agregación de proteínas propensas 2. Un método quees capaz de tender un puente entre la alta resolución de la cristalografía de rayos X y microscopía electrónica de crio-y que no está limitado por el tamaño de la proteína o la concentración es amida de hidrógeno-1 H / 2 H-intercambio (HX) en combinación con espectrometría de masas (MS). En los últimos años este método se ha desarrollado a una herramienta analítica valiosa para el análisis de la dinámica de proteínas, plegamiento de proteínas, estabilidad de la proteína y cambios conformacionales 3-5. La base molecular de este método es la naturaleza lábil de la cadena principal hidrógenos amida en proteínas, que se intercambio con átomos de deuterio cuando la proteína se coloca en un D2O solución. El posterior aumento de la masa de la proteína con el tiempo se mide con alta resolución de la EM.
En péptidos no estructurados cortos sólo HX depende de la temperatura, concentración de catalizador (OH -, H 3 O + es decir, pH, véase la Figura 3) y cadenas laterales de aminoácidos de residuos adyacentes debido a inductiva, gatoefectos alytic y estéricos. Estos efectos sobre el intrínseca tasa de intercambio químico K CH han sido elegantemente cuantificado por Bai et al. 6 y un programa está disponible (por cortesía de Z. Zhang), que calcula k CH para cada aminoácido dentro de un polipéptido dependiente de pH y la temperatura. A pH neutro y temperaturas ambiente K es CH en el orden de 10 1 -10 3 seg -1. En proteínas plegadas HX puede ser 2-9 ordenes de magnitud más lento debido principalmente a enlaces de hidrógeno en la estructura secundaria y en un grado menor debido al acceso restringido de hidratadas iones OH – al interior de una proteína fuertemente plegada. HX en las proteínas nativas, por tanto, implica desarrollo, intercambio químico parcial o global y replegado al estado nativo de acuerdo con la ecuación (1) y los tipos de cambio observados k obs dependen de la tasa de apertura op k, los k cl tasa de cierre y el intercambio químico intrínseca raTE CH K según la ecuación (2).
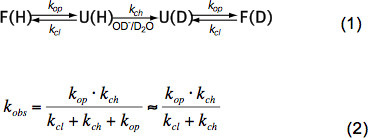
En condiciones de estado nativos K OP es mucho menor que K CH y se puede despreciar en el denominador. Hay dos regímenes cambiarios extremos llamados EX1 y EX2. Si el K CL es mucho menor que K CH (EX1) la tasa observada es prácticamente igual a la tasa de apertura y HX permite la observación inmediata del despliegue de un elemento estructural. Tal régimen de cambio, donde todos los protones amida de cambio a la vez después de la apertura del elemento estructural, es fácilmente observable en la EM por una distribución bimodal de los picos de los isótopos 7. Si k CL es mucho mayor que K CH (EX2) la tasa observada es proporcional a k CH mediante el cual la constante de proporcionalidad es igual a los equilibrios de plegado-desplegado constante K U = K OP </sub> K / cl. En estas condiciones, muchos eventos de apertura y cierre son necesarios antes de todo el intercambio de protones de amida de deuterones, dando lugar a un aumento gradual de la masa media, mientras que la distribución isotópica sigue siendo más o menos el mismo. El régimen EX2 permite la determinación de la energía libre de despliegue Delta G u y, por tanto, la estabilidad de un elemento estructural. Bajo condición de estado nativo régimen EX2 es más común. Aumento del pH y la adición de agentes caotrópicos puede cambiar el mecanismo de intercambio de EX1. Por lo tanto, HX-MS se puede utilizar para explorar termodinámico así como los parámetros cinéticos de plegamiento de la proteína y cambios conformacionales.
Como se mencionó anteriormente HX es intrínsecamente dependiente de pH y la temperatura y el cambio de la vida media de un protón completamente expuestos al disolvente del grupo amida del eje central es de entre 5-400 mseg a pH fisiológico (pH 7,6) y 30 ° C, pero 10 min a> 15 h con un promedio de> 2 horas a pH 2,9 y 0 °C (excepto para el protón de la primera enlace amida columna vertebral de un polipéptido, que intercambia con una vida media de ca. 1-2 min). Bajo tales condiciones lento intercambio es posible digerir la muestra usando proteasas (por ejemplo pepsina) que son activas en estas condiciones, con sin perder toda la información contenida en los deuterones incorporados. Desde la introducción de la digestión péptica en condiciones de intercambio lento, no sólo la cinética global HX de proteínas de longitud completa pueden ser analizados pero HX se pueden localizar en regiones específicas 8,9. La resolución espacial se limita actualmente al tamaño de los fragmentos generados pépticas, que es en general entre 10-30 residuos. Sin embargo, la superposición de fragmentos creados debido a la naturaleza no específica de la escisión por la pepsina podrían conducir a un aumento de la resolución espacial. Además, se encontraron varias otras proteasas para ser activa en condiciones de templado, sin embargo, mucho menos eficiente que la pepsina 10. Además increse de resolución espacial se puede llegar por la fragmentación de péptidos en fase gaseosa por métodos que conservan el patrón deuteración tales como la disociación de captura de electrones (ECD), la transferencia de electrones de disociación (ETD) y disociación multifotónica infrarroja (IRMPD) 11-13. Estas técnicas evitan la pérdida de la resolución espacial debido a la migración intramolecular de protones ("aleatorización"), que se observa por la disociación inducida por colisión (CID) la técnica de fragmentación más comúnmente utilizado. Sin embargo, estos métodos requieren la optimización para cada péptido individual y es por lo tanto todavía bastante difícil.
HX-MS se ha utilizado para analizar la proteína-ligando y proteína-proteína interacciones, incluyendo el montaje de la cápside viral 14-17. Proteína despliegue y replegamiento y se investigaron temperatura inducidas cambios conformacionales 7,18,19. La fosforilación y solo aminoácido conformacional relacionados mutación cambia 16,20 y nucleotSe analizaron los cambios ide-inducidos 21,22. Por lo tanto, este método parece idealmente adecuado para analizar el montaje y la dinámica de máquinas moleculares. Un candidato atractivo, cuyo mecanismo es de gran interés general, es el complejo chaperona Hsp90.
Protocol
Representative Results
Discussion
La unión de una pareja de interacción de una proteína inevitablemente provoca cambios en la accesibilidad de disolvente en el sitio de unión. Además, muchas proteínas sufren cambios conformacionales a vinculantes dinámicos, que afectan a otras regiones de la interfaz de unión real. HX-MS es un método robusto para monitorear estos cambios y es incluso capaz de revelar los cambios conformacionales en proteínas en escalas de tiempo que otros métodos no pueden cubrir.
Para llevar a ca…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Damos las gracias a M. Boysen para comentarios sobre el manuscrito. Este proyecto fue financiado por la Deutsche Forschungsgemeinschaft (SFB638 y MA 1278/4-1 a MPM, y Cluster de Excelencia: CellNetworks EXC 81/1). MPM es investigador del Cluster de Excelencia: CellNetworks.
Materials
| Reagent/Equipment | Company | Catalogue number | Comments (optional) |
| maXis QTOF | Bruker | ||
| nanoAcquity UPLC | Waters Corp. | ||
| Shimadzu 10AD-VP | Shimadzu | ||
| 6-port Valve EPC6W with microelectric actuator | Valco | #EPC6W | |
| Injection valve (manual) | Rheodyne | #7725 | |
| Poros AL20 media | Applied Biosystems | #1-6029-06 | |
| Poros R2 | Applied Biosystems | #1-1118-02 | |
| Pepsin | Sigma | #P6887 | use fresh pepsin |
| Microbore (1 mm) | IDEX | #C-128 | |
| Microbore (2 mm) | IDEX | #C-130B | |
| Acquity UPLC BEH C8 Column | Waters Corp. | #186002876 | |
| Thermomixer | Eppendorf | #5355000.011 | |
| Tubing (various diameters) | IDEX | ||
| Fittings | IDEX | #PK-110 with PK-100 |
References
- Saibil, H. R. Conformational changes studied by cryo-electron microscopy. Nat. Struct. Biol. 7 (9), 711-714 (2000).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Hoofnagle, A. N., Resing, K. A., Ahn, N. G. Protein analysis by hydrogen exchange mass spectrometry. Annu. Rev. Biophys. Biomol. Struct. 32, 1-25 (2003).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass. Spectrom. Rev. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. -. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chem Soc. Rev. 40 (3), 1224-1210 (2011).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Rist, W., Jørgensen, T. J. D., Roepstorff, P., Bukau, B., Mayer, M. P. Mapping temperature-induced conformational changes in the Escherichia coli heat shock transcription factor sigma 32 by amide hydrogen exchange. The Journal of biological chemistry. 278 (51), 51415-51421 (1074).
- Englander, J. J., Rogero, J. R., Englander, S. W. Protein hydrogen exchange studied by the fragment separation method. Anal Biochem. 147 (1), 234-244 (1985).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Cravello, L., Lascoux, D., Forest, E. Use of different proteases working in acidic conditions to improve sequence coverage and resolution in hydrogen/deuterium exchange of large proteins. Rapid Commun. Mass Spectrom. : RCM. 17 (21), 2387-2393 (2003).
- Rand, K. D., Zehl, M., Jensen, O. N., Jørgensen, T. J. D. Protein hydrogen exchange measured at single-residue resolution by electron transfer dissociation mass spectrometry. Anal chem. 81 (14), 5577-5584 (2009).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Electron capture dissociation of electrosprayed protein ions for spatially resolved hydrogen exchange measurements. J. Am. Chem. Soc. 130 (35), 11574-11575 (2008).
- Yamada, N., Suzuki, E. -. I., Hirayama, K. Identification of the interface of a large protein-protein complex using H/D exchange and Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 16 (4), 293-299 (2002).
- Lee, T., Hoofnagle, A. N., et al. Docking motif interactions in MAP kinases revealed by hydrogen exchange mass spectrometry. Mol. Cell. 14 (1), 43-55 (2004).
- Hasan, A., Smith, D. L., Smith, J. B. Alpha-crystallin regions affected by adenosine 5′-triphosphate identified by hydrogen-deuterium exchange. Biochem. 41 (52), 15876-15882 (2002).
- Lanman, J., Lam, T. T., Emmett, M. R., Marshall, A. G., Sakalian, M., Prevelige, P. E. Key interactions in HIV-1 maturation identified by hydrogen-deuterium exchange. Nat. Struct. Mol. Biol. 11 (7), 676-677 (2004).
- Wang, L., Lane, L. C., Smith, D. L. Detecting structural changes in viral capsids by hydrogen exchange and mass spectrometry. Protein Sci. 10 (6), 1234-1243 (2001).
- Pan, H., Raza, A. S., Smith, D. L. Equilibrium and kinetic folding of rabbit muscle triosephosphate isomerase by hydrogen exchange mass spectrometry. J. Mol. Biol. 336 (5), 1251-1263 (2004).
- Mazon, H., Marcillat, O., Forest, E., Smith, D. L., Vial, C. Conformational dynamics of the GdmHCl-induced molten globule state of creatine kinase monitored by hydrogen exchange and mass spectrometry. Biochem. 43 (17), 5045-5054 (2004).
- Lanman, J., Lam, T. T., et al. Identification of novel interactions in HIV-1 capsid protein assembly by high-resolution mass spectrometry. J. Mol. Biol. 325 (4), 759-772 (2003).
- Rist, W., Graf, C., Bukau, B., Mayer, M. P. Amide hydrogen exchange reveals conformational changes in hsp70 chaperones important for allosteric regulation. J. Biol. chem. 281 (24), 16493-16501 (2006).
- Graf, C., Stankiewicz, M., Kramer, G., Mayer, M. P. Spatially and kinetically resolved changes in the conformational dynamics of the Hsp90 chaperone machine. EMBO J. 28 (5), 602-613 (2009).
- Lee, C. -. T., Graf, C., Mayer, F. J., Richter, S. M., Mayer, M. P. Dynamics of the regulation of Hsp90 by the co-chaperone Sti1. EMBO J. 31 (6), 1518-1528 (2012).
- Lou, X., Kirchner, M., et al. Deuteration distribution estimation with improved sequence coverage for HX/MS experiments. Bioinformatics. 26 (12), 1535-1541 (2010).
- Kreshuk, A., Stankiewicz, M., Lou, X., Kirchner, M., Hamprecht, F. A., Mayer, M. P. Automated detection and analysis of bimodal isotope peak distributions in H/D exchange mass spectrometry using HeXicon. Intl. J. mass spectrom. 302 (1-3), 125-131 (2011).
- Walters, B. T., Ricciuti, A., Mayne, L., Englander, S. W. Minimizing back exchange in the hydrogen exchange-mass spectrometry experiment. J. Am. Soc. Mass Spectrom. 23 (12), 2132-2139 (2012).
- Weis, D. D., Wales, T. E., Engen, J. R., Hotchko, M., Ten Eyck, L. F. Identification and characterization of EX1 kinetics in H/D exchange mass spectrometry by peak width analysis. J. Am. Soc. Mass Spectrom. 17 (11), 1498-1509 (2006).
- Fang, J., Rand, K. D., Beuning, P. J., Engen, J. R. False EX1 signatures caused by sample carryover during HX MS analyses. Intl. J. Mass Spectrom. 302 (1-3), 19-25 (2011).
- Deng, Y., Smith, D. L. Identification of unfolding domains in large proteins by their unfolding rates. Biochem. 37 (18), 6256-6262 (1998).
- Wales, T. E., Engen, J. R. Partial unfolding of diverse SH3 domains on a wide timescale. J. Mol. Biol. 357 (5), 1592-1604 (2006).
- Pan, Y., Piyadasa, H., O’Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by h/d exchange and covalent labeling: the glycerol facilitator. J. Mol. Biol. 416 (3), 400-413 (2012).
- Hebling, C. M., Morgan, C. R., Stafford, D. W., Jorgenson, J. W., Rand, K. D., Engen, J. R. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Anal chem. 82 (13), 5415-5419 (2010).
- Abzalimov, R. R., Kaplan, D. A., Easterling, M. L., Kaltashov, I. A. Protein conformations can be probed in top-down HDX MS experiments utilizing electron transfer dissociation of protein ions without hydrogen scrambling. J. Am. Soc. Mass Spectrom. 20 (8), 1514-1517 (2009).

