Hochdurchsatz-Proteinkristallisation mittels Mikrodialyse
Summary
Das vorgestellte Protokoll beschreibt einen einfachen Ansatz für das Screening der Proteinkristallisationsbedingungen und des Kristallwachstums unter Verwendung einer 96-Well-Hochdurchsatz-Dialyseplatte. Der Einsatz von Dialysatorröhrchen für die großskalige Züchtung von Mikrokristallen wird auch für serielle Kristallographie und MicroED-Anwendungen demonstriert.
Abstract
Das Verständnis der Struktur-Funktions-Beziehungen von Makromolekülen, wie z.B. Proteinen, auf molekularer Ebene ist für die Biomedizin und die moderne Arzneimittelforschung von entscheidender Bedeutung. Bis heute ist die Röntgenkristallographie die erfolgreichste Methode, um dreidimensionale Proteinstrukturen mit atomarer Auflösung zu lösen. Mit den jüngsten Fortschritten in der seriellen Kristallographie, entweder unter Verwendung von Röntgenlasern mit freien Elektronen (XFELs) oder Synchrotronlichtquellen, ist die Proteinkristallographie in die nächste Dimension vorgedrungen, wo die Fähigkeit, zeitaufgelöste Daten zu erfassen, wichtige mechanistische Einblicke in das Verhalten biologischer Moleküle bei Raumtemperatur liefert. Dieses Protokoll beschreibt einen einfachen Hochdurchsatz-Workflow (HTP) für das Screening der Kristallisationsbedingungen unter Verwendung einer 96-Well-Dialyseplatte. Diese Platten entsprechen dem Standard der Society for Biomolecular Screening (SBS) und können mit jedem Standard-Kristallisationslabor einfach eingerichtet werden. Sobald die optimalen Bedingungen identifiziert sind, können mit dem Dialysator große Mengen an Kristallen (Hunderte von Mikrokristallen) hergestellt werden. Um die Robustheit und Vielseitigkeit dieses Ansatzes zu validieren, wurden vier verschiedene Proteine kristallisiert, darunter zwei Membranproteine.
Introduction
Im letzten Jahrhundert hat die Röntgenkristallographie entscheidend dazu beigetragen, das Struktur-Funktions-Paradigma biologischer Makromoleküle aufzuklären und zu verstehen. Bis heute ist es eine der erfolgreichsten Methoden zur Aufklärung atomarer Auflösungsstrukturen vieler einzigartig unterschiedlicher Proteine, die für das grundlegende Verständnis der Zellbiochemie, der Medizin und der frühen Wirkstoffforschung von entscheidender Bedeutung sind 1,2. Die Proteinkristallisation bleibt jedoch ein Engpass bei der Untersuchung vieler Proteinziele, insbesondere von Membranproteinen und großen Proteinkomplexen3. Folglich wird die Proteinkristallisation aufgrund der arbeitsintensiven Trial-and-Error-Ansätze fast immer als Kunst angesehen 4,5,6.
Ein Fällungsmittel wird normalerweise in hoher Konzentration zu einer Proteinlösung hinzugefügt, um eine wohlgeordnete, regelmäßige und sich wiederholende Gitteranordnung von Proteinmolekülen, den sogenannten Kristallen, zu bilden. Unter günstigen Bedingungen wie Temperatur, pH-Wert, Konzentration und Fällungsmittel bildet sich schließlich eine übersättigte Lösung, gefolgt von Kristallkeimbildung und Wachstum 7,8. Obwohl es viele Fortschritte bei Kristallisationsversuchsaufbauten gegeben hat, vor allem mit der Entwicklung von Hochdurchsatz-Robotersystemen und der Verfügbarkeit von vorgefertigten “Sparse Matrix”-Bildschirmen, sind die allgemeinen Ansätze zur Proteinkristallisation im Laufe der Jahre weitgehend unverändert geblieben. Zu den gängigen experimentellen Proteinkristallisationstechniken gehören die Dampfdiffusion (hängender Tropfen und sitzender Tropfen)9, die Mikrocharge (unter Öl)10,11, die Diffusion mit freier Schnittstelle (mikrofluidische Geräte)12 und die Dialyse (unter Verwendung von Knöpfen und anderen Techniken)13,14,15. Es existieren jedoch auch andere, speziellere Aufbauten, wie z. B. Mesophasenansätze zur Kristallisation von Membranproteinen16,17. Während die Mehrzahl der in der Proteindatenbank hinterlegten Röntgenproteinstrukturen bisher durch Kristallisation mit Dampfdiffusionsmethoden 6,18 gelöst wurde, scheinen andere Ansätze, wie z.B. die Kristallisation durch Dialyse, zu wenig genutzt zu werden, was wahrscheinlich auf die praktischen Aspekte ihres Versuchsaufbaus zurückzuführen ist.
Die Kristallisation durch Dialyse beruht einfach auf der langsamen Diffusion von gelösten Stoffen (Fällungsmitteln, Ionen, Additiven und Puffern) durch eine semipermeable Membran, die gleichzeitig die Zirkulation von Proteinmolekülen verhindert. Auf diese Weise wird die Proteinlösung langsam ins Gleichgewicht gebracht, wobei das Fällungsmittel die notwendige Konzentration erreicht, um zu kristallisieren. Die Kinetik des Systems hängt von der Temperatur, der Fällmittelkonzentration und dem Molekulargewichtsgrenzwert (MWCO) der Zellulosemembran ab19. Das bisher beliebteste Kristallisationssystem in der Dialyse sind Mikrodialyseknöpfe aus transparenten Acrylplatten. Diese werden in der Regel in Reservoirs getaucht (meist unter Verwendung von Dampfdiffusions-Hängetropfenplatten), die die Kristallisationsfällungslösungen enthalten. Diese Methode mit niedrigerem Durchsatz erfordert jedoch auch eine spezielle Montage, um die Proteinlösung innerhalb der Dialysemembran zu versiegeln, die über der Knopfkammer platziert ist, wie in Abbildung 1 dargestellt. Darüber hinaus sind Luftblasen, die zwischen der Dialysemembran und der Proteinlösung eingeschlossen sind, ein häufiges Problem, das das Kristallwachstum beeinträchtigen. Eine weitere Einschränkung des Verfahrens sind die Probenanforderungen, bei denen im Vergleich zu Dampfdiffusionsmethoden wesentlich höhere Konzentrationen und Volumina erforderlich sind, um die Dialysetasten unterzubringen. Daher wurde die Kristallisation mit Mikrodialyseknöpfen als unattraktiv empfunden, insbesondere für schwierige Ziele wie Membranproteine, deren Reinigungsausbeute frustrierend gering ist. In jüngster Zeit wurden mikrofluidische Geräte entwickelt, um die Proteinkristallisation durch Dialyse zu erleichtern15. Diese Chips wurden auch so konzipiert, dass sie eine hohe Röntgentransparenz bei niedrigem Hintergrund aufweisen, so dass die Chips für die In-situ-Datenerfassung bei Raumtemperatur verwendet werden können, wodurch die Unannehmlichkeiten der Ernte und Kryokühlung von Kristallen entfallen. Trotz dieser Fortschritte ist der Ansatz immer noch sehr durchsatzarm und teuer.
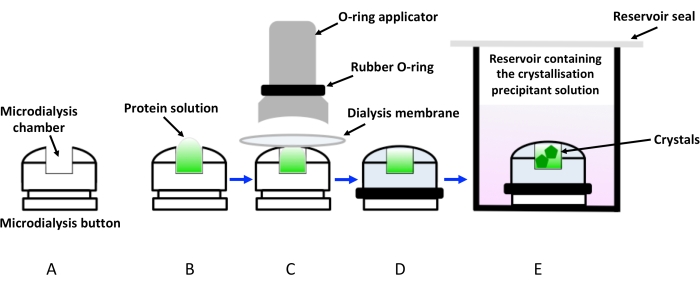
Abbildung 1: Schematische Darstellung der Kristallisation durch Dialyse mit Hilfe von Dialysetasten. (A) Schematische Darstellung eines Kristallisationsdialyseknopfes. (B) Die Proteinlösung wird in die Mikrodialyse-Knopfkammer gegeben. (C) Die Dialysemembran wird mit Hilfe eines Gummirings (O-Rings), der über einen Applikator angebracht wird, am Mikrodialyseknopf gehalten. (D) Der Dialyseknopf kann in das Reservoir mit der Kristallisationslösung (Dialyselösung) eingetaucht werden, wie in (E) gezeigt. Die Durchstechflasche mit dem eingetauchten Dialyseknopf muss verschlossen sein, um eine Verdunstung zu vermeiden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Hier wird ein einfaches Protokoll für das Screening der Proteinkristallisationsbedingungen und des Kristallwachstums unter Verwendung der 96-Well-Hochdurchsatz-Dialyseplatte vorgestellt. Diese Einwegplatten sind so konzipiert, dass sie ähnlich wie die Dampfdiffusionskristallisationsplatten (Pipette und dann Versiegelung) verwendet werden können, wie in Abbildung 2 gezeigt. Die Platten können bis zu 3,2 μl Protein und 350 μl Dialyselösung aufnehmen. Jedes Bohrloch verfügt über eine separate, regenerierte Zellulosemembran, um eine Kreuzkontamination zwischen den Bohrlöchern zu verhindern. Der Aufbau dauert etwa 10 Minuten und erfordert keine spezielle Ausrüstung, außer denen, die in allen Standard-Kristallisationslabors zu finden sind. Vier verschiedene Proteine, darunter zwei Membranproteine, werden verwendet, um diesen Ansatz als effektive Methode für die Hochdurchsatz-Proteinkristallographie (HTP) zu demonstrieren und zu validieren.
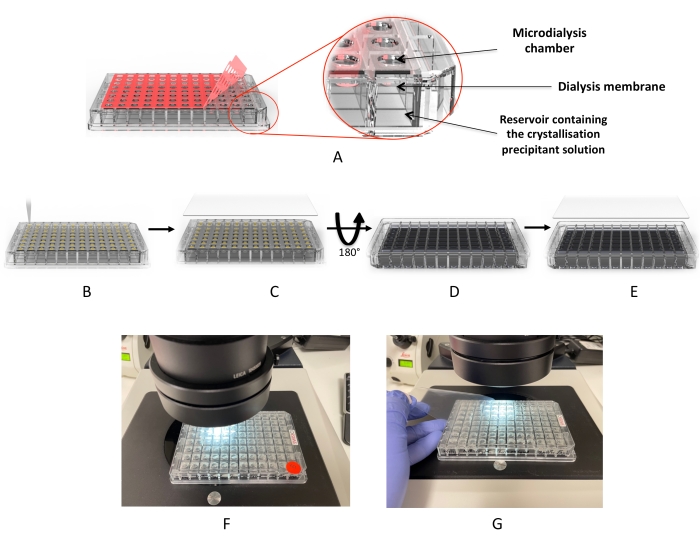
Abbildung 2: Kristallisations-Workflow mit der Mikrodialyseplatte . (A) Entfernen der roten Klebefolie. (B) Abgabe der Proteintröpfchen in jede der Tropfenvertiefungen. (C) Die Vertiefungen werden mit der UV-“Abdeckfolie” abgedeckt. (D) Die Platte wird umgedreht, um die Dialyselösungen (oder das Kristallisationssieb) hinzuzufügen. (E) Die Platte wird versiegelt und inkubiert. (F,G) Mikroskopische Inspektion der Tropfen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Die Verwendung dieses Kristallisationsprotokolls durch Dialyseprotokoll wurde anhand des 0,5-ml-Dialysatorröhrchens (Abbildung 3) für die Herstellung von Mikrokristallen im großen Maßstab (Hunderte bis Tausende) demonstriert, die für modernste Datenerfassungsmethoden wie die serielle Kristallographie an den beiden XFEL-Einrichtungen 20,21,22,23,24 und Synchrotrons25,26,27 geeignet sind , sowie für MicroED28,29,30 Ansätze.
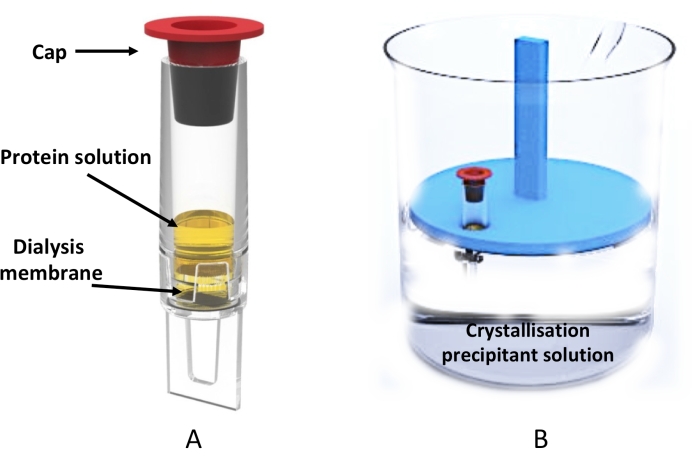
Abbildung 3: Mikrodialysekristallisation im großen Maßstab mit dem Dialysatorröhrchen . (A) Schematische Darstellung des 0,5-ml-Dialysatorröhrchens. (B) Seitenansicht eines Becherglases, das die Kristallisationslösung enthält, und des schwimmenden Röhrchengestells, das ein Dialysatorröhrchen enthält. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Protocol
Representative Results
Discussion
Derzeit ist die Kristallisation durch Dialyse die am wenigsten genutzte Kristallisationsmethode, insbesondere aufgrund des geringen Durchsatzes und der langwierigen Natur der bestehenden Ansätze, wie z. B. der Mikrodialyse mit Knöpfen. Hier folgt ein einfaches, aber robustes Protokoll einem HTP-Workflow für die Züchtung von Proteinkristallen durch die Dialyse durch eine kommerziell erhältliche Mikrodialyseplatte und einen Dialysatorschlauch. Je nach Zielprotein sind die Mikrodialyseplatte und das Dialysatoröhrchen wahlweise mit einer Dialysemembran mit einem MWCO von 3, 5, 10 oder 30 kDa ausgestattet. Das Protokoll kann problemlos in jeder Standard-Kristallisationsanlage eingerichtet werden und hat den großen Vorteil, dass es sowohl auf lösliche als auch auf Membranproteine anwendbar ist. Protein-Protein- und Protein-Nukleinsäure-Komplexe wurden während dieses Protokolls jedoch nicht getestet.
Wie bei jedem Kristallisationsansatz durch Dialyse ist das Volumenverhältnis zwischen Proteinprobe und Dialyselösung entscheidend. In diesem Protokoll wird ein Verhältnis von 1:100 zwischen der Probe und der Dialyselösung empfohlen, aber da die Mikrodialyseplatte eine maximale Kapazität von 350 μl Dialyselösung zulässt, können diese Verhältnisse untersucht werden, um Kristalltreffer zu erhalten. Ein Volumen von 1-2 μL Protein wird im vorgestellten Protokoll beim Aufbau von Kristallisationsplatten verwendet. Dadurch soll sichergestellt werden, dass die Tropfen mit einer manuellen Mehrkanalpipette genau aushärten. Durch den Einsatz von elektronischen Pipetten (Mehrkanal- oder Wiederholungspipetten) oder HTP-Flüssigkeitsdosierrobotern können Tropfen mit geringerem Volumen genau erreicht werden, wodurch die benötigte Proteinmenge gesenkt wird. Darüber hinaus ist es aufgrund der relativ geringen Mengen an Dialysepuffer, die von der Mikrodialyseplatte benötigt werden (im Gegensatz zu anderen konventionellen Dialysemethoden), möglich (ohne den umfangreichen Einsatz von Ressourcen), große chemische Räume zu erkunden, und zwar nicht nur mit kommerziell erhältlichen Kristallisationssieben, sondern auch mit Optimierungsbildschirmen (die um die anfängliche Kristallisationstrefferbedingung herum ausgelegt sind).
Ein kritischer Schritt im vorgestellten HTP-Verfahren ist die rechtzeitige Applikation kleiner Proteinvolumina (0,50-3,2 μl pro Bedingung) auf die Dialyseplatte, um Dehydratisierung und Probenverlust zu begrenzen. Dies kann leicht durch den Einsatz einer Mehrkanalpipette, einer Wiederholungspipette oder eines robotergestützten Kristallisationssystems gemildert werden. Die lange Inkubationszeit, z. B. mehr als 2 Wochen, der Platten bei 20 °C kann zu einer Austrocknung der Proteintröpfchen oder zu einer Schädigung der neu gebildeten Kristalle führen. Die Aufbewahrung der Dialyseplatten in einer Befeuchtungskammer oder einem verschließbaren Beutel kann diesen Effekt abmildern. Darüber hinaus wird die Verwendung steriler Materialien und Techniken empfohlen, um Bakterienwachstum zu vermeiden.
Wie in der Einleitung erwähnt, wurde das Gebiet der Protein-Röntgenkristallographie in jüngster Zeit durch die Entwicklung neuer und bestehender Kristallisationstechniken, moderner Ansätze zur Kristallprobenabgabe, neuer Generationen von Röntgenquellen und neuer ausgeklügelter Methoden zur Datenerfassung und -verarbeitung revolutioniert36 ,37,38. Daher hat sich das Aufkommen der seriellen Mikrokristallographie bei Raumtemperatur, die entweder mit XFELs oder Synchrotronlichtquellen durchgeführt wird, zu einem bemerkenswerten Werkzeug in der Strukturbiologie entwickelt, insbesondere auf dem Gebiet der Membranproteine39. Es sind jedoch Tausende von Mikrokristallen erforderlich, um genügend Daten für eine robuste Strukturlösung zu generieren, was (zumindest mit herkömmlichen Kristallisationsmethoden) keine leichte Aufgabe ist. Das hier beschriebene Dialysekristallisationsverfahren ermöglicht die Herstellung einer großen Anzahl von Mikrokristallen. Nachdem die Kristallisationsbedingungen für die Herstellung von Mikrokristallen (1-10 μm) durch den Einsatz einer Mikrodialyseplatte bestimmt wurden, können mit dem 0,5-ml-Dialysator große Mengen an Mikrokristallen hoher Dichte hergestellt werden (Abbildung 5). Diese Kristalle eignen sich ideal für die Datenerfassung mit festen Targets oder Flüssigstrahl-Probenzufuhrsystemen27,40. Kristalle, die mit dieser Methode gewonnen werden, können auch für MicroED-Anwendungen geeignet sein. Diese müssen jedoch möglicherweise auf eine für diese spezielle Anwendung geeignete Größe und Dicke gefräst werden, da Elektronen viel stärker mit Kristallen wechselwirken als Röntgenphotonen41.
Zusammenfassend lässt sich sagen, dass der hier beschriebene Ansatz der Kristallisation durch Dialyse zu den sich entwickelnden Strategien in der Proteinkristallisation zur Strukturaufklärung beiträgt und das Spektrum der Anstrengungen erweitert, die zur Bestimmung neuer Proteinziele eingesetzt werden können, die bisher mit anderen konventionellen Methoden erfolglos waren.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Wir erkennen die Finanzierung durch das britische Ministerium für Wirtschaft, Energie und Industriestrategie (BEIS) an. Wir danken Alex R. Jones und Mike Shaw vom National Physical Laboratory für ihr Feedback zum Manuskript.
Materials
| 0.2 mL tubes | Thermo Scientific | AB0620 | For aliquoting protein solutions. |
| 0.2 µm syringe filter | Sartorius | 17823———-K | Surfactant-free cellulose acetate filters. For filtering dialysis solutions. |
| 0.22 µm membrane filters | Millipore | GSTF04700 | Membrane filters for filtering large volumes of buffers |
| 12-channel, variable 0.5 – 10 µL Research plus pipette | Eppendorf | 3125000028 | For dispensing protein drops onto the Diaplate. |
| 12-channel, variable 30 – 300 µL Eppendorf Research plus pipette | Eppendorf | 3125000060 | For dispensing dialysis solutions on the Diaplate reservoirs. |
| 20 mL syringe | Fisherbrand | 15889152 | For use with syringe filters. |
| 96 well 2.2 mL deep-well plates | Thermo Scientific | AB0788 | Polypropylene deep-well storage plates; for preparing screens using the Hamilton Microlab STARlet. |
| Centrifuge 5425 | Eppendorf | 5405000565 | With rotor FA-24×2 with a maximum g-force of 21,300 x g. |
| Diacon dialyser | SWISSCI | W72010 | Dialyzer tubes with a regenerated cellulose membrane with a molecular weight cut-off of 10 kDa. Ideal for protein solutions of up to 0.5 mL. |
| Diaplate 96-well plate | SWISSCI | W82010 | Microdialysis plate. The Diaplate consists of two sides with a regenerated cellulose membrane in-between with a molecular weight cut-off of 10 kDa. |
| Falcon 50 mL High Clarity PP Centrifuge Tube | Corning | 352070 | For holding dialysis solutions. |
| Floating rack | SWISSCI | n/a | Included in the Diacon kit |
| Floor-standing vibration-free incubator | Molecular Dimensions | MD5-01 | 400 L temperature-controlled incubator set to 20 °C. |
| Leica M205 C stereo microscope | Leica | Planapo 1.0x objective, 7.8x – 160x zoom range with DMC 4500 camera | |
| Lysozyme from chicken egg white | Sigma Aldrich | 62971 | Lyophilized protein |
| Memgold2 | Molecular Dimensions | MD1-64 | Sparse-matrix screen |
| Microlab STARlet | Hamilton | n/a | Liquid handler system. |
| Reservoir cover film | SWISSCI | n/a | Included in the Diaplate kit |
| Reusable bottle top filter | Thermo Scientific | DS0320-5045 | For fitering large volumes of buffers, for use with 0.22 µm membrane filters |
| Sealing paddle | SWISSCI | n/a | Included in the Diaplate kit |
| Thaumatin from Thaumatococcus daniellii | Sigma Aldrich | T7638 | Lyophilized protein |
| UV cover film | SWISSCI | n/a | Included in the Diaplate kit |
References
- Brooks-Bartlett, J. C., Garman, E. F. The nobel science: One hundred years of crystallography. Interdisciplinary Science Reviews. 40 (3), 244-264 (2015).
- Maveyraud, L., Mourey, L. Protein X-ray crystallography and drug discovery. Molecules. 25 (5), (2020).
- Kwan, T. O. C., Axford, D., Moraes, I. Membrane protein crystallography in the era of modern structural biology. Biochemical Society Transactions. 48 (6), 2505-2524 (2020).
- Birch, J., et al. The fine art of integral membrane protein crystallisation. Methods. 147, 150-162 (2018).
- Gorrec, F., Löwe, J. Automated protocols for macromolecular crystallization at the MRC laboratory of molecular biology. Journal of Visualized Experiments. 131 (131), (2018).
- Govada, L., Chayen, N. E. Choosing the method of crystallization to obtain optimal results. Crystals. 9 (2), 106 (2019).
- Chayen, N. E. Turning protein crystallisation from an art into science. Current Opinion in Structural Biology. 14 (5), 577-583 (2004).
- McPherson, A., Gavira, J. A. Introduction to protein crystallization. Acta Crystallographica Section F: Structural Biology Communications. 70 (1), 2-20 (2014).
- Gulbis, J. Protein crystallography: methods and protocols. Crystallography Reviews. 24 (2), 136-143 (2018).
- D’Arcy, A., Bergfors, T., Cowan-Jacob, S. W., Marsh, M. Microseed matrix screening for optimization in protein crystallization: What have we learned. Acta Crystallographica Section:F Structural Biology Communications. 70 (9), 1117-1126 (2014).
- Shaw Stewart, ., D, P., Kolek, S. A., Briggs, R. A., Chayen, N. E., Baldock, P. F. Random microseeding: a theoretical and practical exploration of seed stability and seeding techniques for successful protein crystallization. Crystal Growth & Design. 11 (8), 3432-3441 (2011).
- Junius, N., et al. A microfluidic device for both on-chip dialysis protein crystallization and in situ X-ray diffraction. Lab on a Chip. 20 (2), 296-310 (2020).
- Junius, N., Vahdatahar, E., Oksanen, E., Ferrer, J. L., Budayova-Spano, M. Optimization of crystallization of biological macromolecules using dialysis combined with temperature control. Journal of Applied Crystallography. 53 (3), 686-698 (2020).
- Vahdatahar, E., Junius, N., Budayova-Spano, M. Optimization of crystal growth for neutron macromolecular crystallography. Journal of Visualized Experiments. 169, (2021).
- Jaho, S., et al. Crystallization of proteins on chip by microdialysis for in situ X-ray diffraction studies. Journal of Visualized Experiments. 170, (2021).
- Liu, W., Cherezov, V. Crystallization of membrane proteins in lipidic mesophases. Journal of Visualized Experiments. 49, 2501 (2011).
- Ujwal, R., Abramson, J. High-throughput crystallization of membrane proteins using the lipidic bicelle method. Journal of Visualized Experiments. 59, (2012).
- Parker, J. L., Newstead, S. Membrane protein crystallisation: Current trends and future perspectives. Advances in Experimental Medicine and Biology. 922. , 61-72 (2016).
- Apostolopoulou, V., Junius, N., Sear, R. P., Budayova-Spano, M. Mixing salts and polyethylene glycol into protein solutions: The effects of diffusion across semi-permeable membranes and of convection. Crystal Growth & Design. 20 (6), 3927-3936 (2020).
- Neutze, R., Wouts, R., Vander Spoel, D., Weckert, E., Hajdu, J. Potential for biomolecular imaging with femtosecond X-ray pulses. Nature. 406 (6797), 752-757 (2000).
- Chapman, H. N., et al. Femtosecond X-ray protein nanocrystallography. Nature. 470 (7332), 73-78 (2011).
- Mizohata, E., Nakane, T., Fukuda, Y., Nango, E., Iwata, S. Serial femtosecond crystallography at the SACLA: breakthrough to dynamic structural biology. Biophysical Reviews. 10 (2), 209-218 (2018).
- Nogly, P., et al. Lipidic cubic phase serial millisecond crystallography using synchrotron radiation. IUCrJ. 2 (2), 168-176 (2015).
- Johansson, L. C., et al. XFEL structures of the human MT2 melatonin receptor reveal the basis of subtype selectivity. Nature. 569 (7755), 289-292 (2019).
- Owen, R. L., et al. Low-dose fixed-target serial synchrotron crystallography. Acta Crystallographica Section D: Structural Biology. 73 (4), 373-378 (2017).
- Weinert, T., et al. Serial millisecond crystallography for routine room-temperature structure determination at synchrotrons. Nature communications. 8 (1), 542 (2017).
- Axford, D., et al. Two states of a light-sensitive membrane protein captured at room temperature using thin-film sample mounts. Acta Crystallographica Section D: Structural Biology. 78 (1), 52-58 (2022).
- Nannenga, B. L., Gonen, T. The cryo-EM method microcrystal electron diffraction (MicroED). Nature Methods. 16 (5), 369-379 (2019).
- Nguyen, C., Gonen, T. Beyond protein structure determination with MicroED. Current Opinion in Structural Biology. 64, 51-58 (2020).
- Mu, X., Gillman, C., Nguyen, C., Gonen, T. An overview of microcrystal electron diffraction (MicroED). Annual Review of Biochemistry. 90, 431-450 (2021).
- Kwan, T. O. C., et al. Selection of biophysical methods for characterisation of membrane proteins. International Journal of Molecular Sciences. 22 (10), 2605 (2019).
- Pos, K. M., Purification Diederichs, K. crystallization and preliminary diffraction studies of AcrB, an inner-membrane multi-drug efflux protein. Acta Crystallographica Section D: Biological Crystallography. 58 (10), 1865-1867 (2002).
- Guan, L., Mirza, O., Verner, G., Iwata, S., Kaback, H. R. Structural determination of wild-type lactose permease. Proceedings of the National Academy of Sciences. 104 (39), 15294-15298 (2007).
- Kwan, T. O. C., Reis, R., Moraes, I. In situ measurements of polypeptide samples by dynamic light scattering: membrane proteins, a case study. Methods in Molecular Biology. , 189-202 (2021).
- Seddon, A. M., Curnow, P., Booth, P. J. Membrane proteins, lipids and detergents: not just a soap opera. Biochimica et Biophysica Acta (BBA)-Biomembranes. (1-2), 105-117 (2004).
- Wickstrand, C., et al. A tool for visualizing protein motions in time-resolved crystallography. Structural Dynamics. 7 (2), 024701 (2020).
- Orville, A. M. Recent results in time resolved serial femtosecond crystallography at XFELs. Current Opinion in Structural Biology. 65, 193-208 (2020).
- Schulz, E. C., Yorke, B. A., Pearson, A. R., Mehrabi, P. Best practices for time-resolved serial synchrotron crystallography. Acta Crystallographica Section D: Structural Biology. 78 (1), 14-29 (2022).
- Neutze, R., Brändén, G., Schertler, G. F. Membrane protein structural biology using X-ray free eletron lasers. Current Opinion in Structural Biology. 33, 115-125 (2015).
- Wierstall, U. Liquid sample delivery techniques for serial femtosecond crystallography. Philosophical Transactions of the Royal Society B: Biological Sciences. 369 (1647), 20130337 (2014).
- Shi, D., Nannenga, B. L., Iadanza, M. G., Gonen, T. Three-dimensional electron crystallography of protein microcrystals. eLife. 2, 01345 (2013).

