Cristalización de proteínas de alto rendimiento mediante microdiálisis
Summary
El protocolo presentado describe un enfoque sencillo para la detección de las condiciones de cristalización de proteínas y el crecimiento de cristales utilizando una placa de diálisis de alto rendimiento de 96 pocillos. El uso de tubos dializadores para el crecimiento a gran escala de microcristales también se demuestra para aplicaciones de cristalografía en serie y MicroED.
Abstract
Comprender las relaciones estructura-función de las macromoléculas, como las proteínas, a nivel molecular es vital para la biomedicina y el descubrimiento de fármacos modernos. Hasta la fecha, la cristalografía de rayos X sigue siendo el método más exitoso para resolver estructuras de proteínas tridimensionales a resolución atómica. Con los recientes avances en cristalografía en serie, ya sea utilizando láseres de electrones libres de rayos X (XFEL) o fuentes de luz sincrotrón, la cristalografía de proteínas ha progresado a la siguiente frontera, donde la capacidad de adquirir datos resueltos en el tiempo proporciona importantes conocimientos mecanicistas sobre el comportamiento de las moléculas biológicas a temperatura ambiente. Este protocolo describe un flujo de trabajo sencillo de alto rendimiento (HTP) para detectar condiciones de cristalización mediante el uso de una placa de diálisis de 96 pocillos. Estas placas siguen el estándar de la Sociedad para el Cribado Biomolecular (SBS) y se pueden configurar fácilmente utilizando cualquier laboratorio de cristalización estándar. Una vez que se identifican las condiciones óptimas, se pueden producir grandes cantidades de cristales (cientos de microcristales) utilizando el dializador. Para validar la robustez y versatilidad de este enfoque, se cristalizaron cuatro proteínas diferentes, incluidas dos proteínas de membrana.
Introduction
Durante el último siglo, la cristalografía de rayos X ha sido fundamental para dilucidar y comprender el paradigma estructura-función de las macromoléculas biológicas. Hasta la fecha, sigue siendo uno de los métodos más exitosos para dilucidar las estructuras de resolución atómica de muchas proteínas singularmente diferentes que son cruciales para la comprensión fundamental de la bioquímica celular, la medicina y el descubrimiento temprano de fármacos 1,2. Sin embargo, la cristalización de proteínas sigue siendo un cuello de botella en el estudio de muchas dianas proteicas, particularmente proteínas de membrana y grandes complejos proteicos3. En consecuencia, la cristalización de proteínas casi siempre se considera un arte debido a los enfoques de ensayo y error intensivos en mano de obra empleados 4,5,6.
Por lo general, se agrega un agente precipitante a una solución de proteína a alta concentración para formar una disposición reticular bien ordenada, regular y repetitiva de moléculas de proteína, conocidas como cristales. En condiciones favorables, como temperatura, pH, concentración y agente precipitante, finalmente se forma una solución sobresaturada, seguida de nucleación cristalina y crecimiento 7,8. Aunque ha habido muchos avances en las configuraciones de prueba de cristalización, predominantemente con el desarrollo de sistemas robóticos de alto rendimiento y la disponibilidad de pantallas de “matriz dispersa” listas para usar, los enfoques generales para la cristalización de proteínas se han mantenido sin cambios a lo largo de los años. Las técnicas experimentales comunes de cristalización de proteínas incluyen la difusión de vapor (gota colgante y gota sentada)9, microlotes (bajo aceite)10,11, difusión de interfaz libre (dispositivos microfluídicos)12 y diálisis (uso de botones y otras técnicas)13,14,15. Sin embargo, también existen otras configuraciones más especializadas, como los enfoques de mesofase para cristalizar proteínas de membrana16,17. Si bien la mayoría de las estructuras de proteínas de rayos X depositadas en el Banco de Datos de Proteínas se han resuelto hasta ahora mediante métodos de cristalización por difusión de vapor 6,18, otros enfoques, como la cristalización por diálisis, parecen estar infrautilizados, probablemente debido a los aspectos prácticos relacionados con su configuración experimental.
La cristalización por diálisis simplemente se basa en la lenta difusión de solutos (precipitantes, iones, aditivos y tampones) a través de una membrana semipermeable que simultáneamente impide que circulen las moléculas de proteínas. De esta manera, la solución proteica se equilibra lentamente, y el precipitante alcanza la concentración necesaria para cristalizar. La cinética del sistema depende de la temperatura, la concentración de precipitante y el corte de peso molecular de la membrana de celulosa (MWCO)19. Hasta la fecha, la configuración de cristalización más popular por diálisis ha sido el uso de botones de microdiálisis hechos de láminas de acrílico transparente. Estos generalmente se sumergen en depósitos (principalmente utilizando placas de gota colgantes de difusión de vapor) que contienen las soluciones precipitantes de cristalización. Sin embargo, este método de menor rendimiento también requiere un ensamblaje específico para sellar la solución de proteína dentro de la membrana de diálisis colocada sobre la cámara del botón, como se ilustra en la Figura 1. Además, las burbujas de aire atrapadas entre la membrana de diálisis y la solución proteica son un problema frecuente que afecta el crecimiento de cristales. Otra limitación del método son los requisitos de la muestra, por lo que se necesitan concentraciones y volúmenes mucho más altos en comparación con los métodos de difusión de vapor, para acomodar los botones de diálisis. Por lo tanto, la cristalización mediante botones de microdiálisis se ha percibido como un método poco atractivo, especialmente para objetivos difíciles como las proteínas de membrana, cuyos rendimientos de purificación son frustrantemente bajos. Recientemente, se han desarrollado dispositivos microfluídicos para facilitar la cristalización de proteínas por diálisis15. Estos chips también han sido diseñados para tener una alta transparencia de rayos X con bajo fondo, lo que permite que los chips se utilicen para la recopilación de datos in situ a temperatura ambiente, eliminando así el inconveniente de cosechar y crioenfriar cristales. A pesar de estos avances, el enfoque sigue siendo de muy bajo rendimiento y costoso.
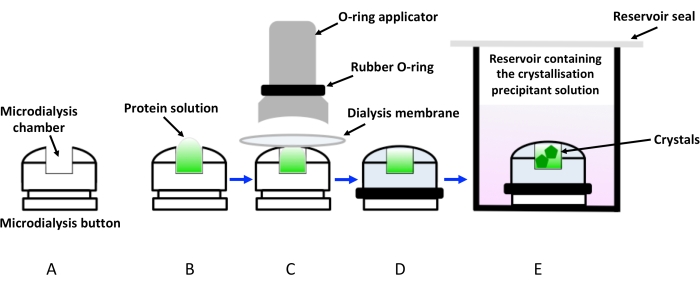
Figura 1: Representación esquemática de la cristalización por diálisis mediante botones de diálisis. (A) Representación esquemática de un botón de diálisis de cristalización. (B) La solución de proteína se agrega a la cámara de botón de microdiálisis. (C) La membrana de diálisis se sujeta al botón de microdiálisis con la ayuda de un anillo de goma (junta tórica) aplicado a través de un aplicador. (D) El botón de diálisis está listo para sumergirse en el depósito que contiene la solución de cristalización (solución de diálisis), como se muestra en (E). El vial que contiene el botón de diálisis sumergido debe sellarse para evitar la evaporación. Haga clic aquí para ver una versión más grande de esta figura.
Aquí, se presenta un protocolo sencillo para detectar las condiciones de cristalización de proteínas y el crecimiento de cristales utilizando la placa de diálisis de alto rendimiento de 96 pocillos. Estas placas desechables están diseñadas para ser utilizadas de manera similar a las placas de cristalización por difusión de vapor (pipeta y luego sello), como se muestra en la Figura 2. Las placas pueden acomodar hasta 3,2 μL de proteína y 350 μL de solución de diálisis. Cada pozo cuenta con una membrana de celulosa regenerada separada para evitar la contaminación cruzada entre los pozos. La configuración tarda alrededor de 10 minutos en completarse y no requiere ningún equipo especializado además de lo que se puede encontrar en todos los laboratorios de cristalización estándar. Se utilizan cuatro proteínas diferentes, incluidas dos proteínas de membrana, para demostrar y validar este enfoque como un método eficaz para la cristalografía de proteínas de alto rendimiento (HTP).
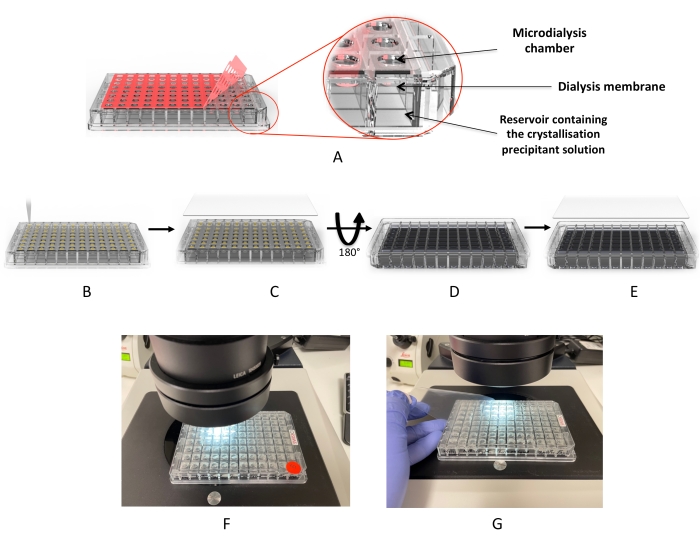
Figura 2: Flujo de trabajo de cristalización utilizando la placa de microdiálisis. (A) Eliminación de la “película de cubierta” adhesiva roja. (B) Dispensar las gotas de proteína en cada uno de los pocillos de gota. (C) Los pozos están cubiertos con la “película de cubierta” UV. (D) La placa se invierte para agregar las soluciones de diálisis (o pantalla de cristalización). (E) La placa se sella e incuba. (F,G) Inspección microscópica de las gotas. Haga clic aquí para ver una versión más grande de esta figura.
El uso de este protocolo de cristalización por diálisis se demostró utilizando el tubo dializador de 0,5 ml (Figura 3) para la producción a gran escala (cientos a miles) de microcristales, adecuados para métodos de recolección de datos de última generación, como la cristalografía en serie en ambas instalaciones XFEL 20,21,22,23,24 y sincrotrones25,26,27 , así como para los enfoques MicroED28,29,30.
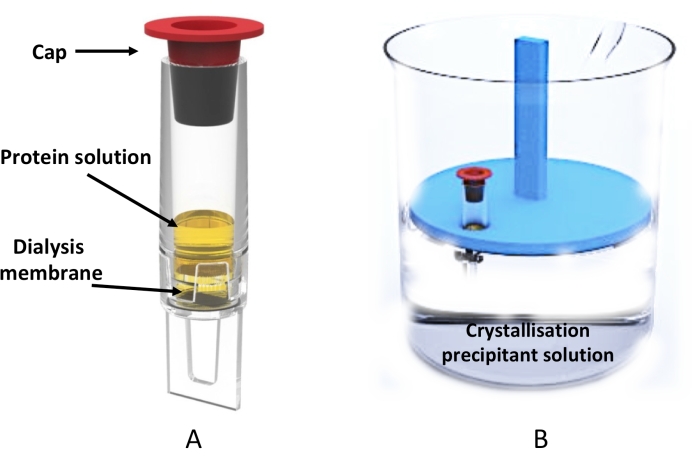
Figura 3: Cristalización de microdiálisis a gran escala utilizando el tubo dializador . (A) Representación esquemática del tubo dializador de 0,5 ml. (B) Vista lateral de un vaso de precipitados que contiene la solución de cristalización y la rejilla de tubos flotantes que sostiene un tubo de dializador. Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
Actualmente, la cristalización por diálisis es el método de cristalización más infrautilizado, debido a la naturaleza tediosa y de bajo rendimiento de los enfoques existentes, como la microdiálisis con botones. Aquí, un protocolo simple pero robusto sigue un flujo de trabajo de HTP para el crecimiento de cristales de proteínas por diálisis a través de una placa de microdiálisis disponible comercialmente y un tubo dializador. Dependiendo de la proteína objetivo, la placa de microdiálisis y el tubo dializador utilizados en el procedimiento vienen con la opción de una membrana de diálisis con un MWCO de 3, 5, 10 o 30 kDa. El protocolo se puede configurar fácilmente en cualquier instalación de cristalización estándar y tiene la gran ventaja de ser aplicable tanto a proteínas solubles como de membrana. Sin embargo, los complejos proteína-proteína y proteína-ácido nucleico no se probaron durante este protocolo.
Como en cualquier enfoque de cristalización por diálisis, la relación de volumen entre la muestra de proteína y la solución de diálisis es crítica. En este protocolo, se recomienda una relación de 1:100 entre la muestra y la solución de diálisis, pero dado que la placa de microdiálisis permite una capacidad máxima de 350 μL de solución de diálisis, estas proporciones se pueden explorar para obtener golpes de cristal. Un volumen de 1-2 μL de proteína se utiliza en el protocolo presentado al configurar placas de cristalización. Esto es para garantizar que las gotas se fijen con precisión con una pipeta manual multicanal. Mediante el uso de pipetas electrónicas (pipetas de dispensación multicanal o de repetición) o robots dispensadores de líquidos HTP, se pueden lograr gotas de volúmenes más bajos con precisión, reduciendo así la cantidad de proteína requerida. Además, debido a los volúmenes relativamente bajos de tampón de diálisis requeridos por la placa de microdiálisis (a diferencia de otros métodos de diálisis convencionales), es posible (sin el uso extensivo de recursos) explorar grandes espacios químicos, no solo utilizando pantallas de cristalización disponibles comercialmente sino también con pantallas de optimización (diseñadas en torno a la condición de impacto de cristalización inicial).
Un paso crítico en el procedimiento de HTP presentado es la aplicación oportuna de pequeños volúmenes de proteína (0.50-3.2 μL por condición) a la placa de diálisis para limitar la deshidratación y la pérdida de muestra. Esto se puede mitigar fácilmente mediante el uso de una pipeta multicanal, una pipeta de dispensación repetida o un sistema de cristalización robótica. El largo tiempo de incubación, como más de 2 semanas, de las placas a 20 °C puede provocar la deshidratación de las gotas de proteína o daños en los cristales recién formados. Mantener las placas de diálisis dentro de una cámara de humidificación o una bolsa sellable puede aliviar este efecto. Además, se recomienda el uso de materiales y técnicas estériles para evitar el crecimiento bacteriano.
Como se mencionó en la introducción, recientemente, con la creciente necesidad de comprender la dinámica estructural de las proteínas para los mecanismos de la enfermedad, las interacciones de unión proteína-ligando y las interacciones proteína-proteína, el campo de la cristalografía de rayos X de proteínas se ha revolucionado mediante el desarrollo de técnicas de cristalización nuevas y existentes, enfoques modernos para la entrega de muestras de cristal, nuevas generaciones de fuentes de rayos X y nuevos métodos sofisticados para la adquisición y procesamiento de datos36 ,37,38. Por lo tanto, el advenimiento de la microcristalografía seriada a temperatura ambiente, ya sea realizada utilizando XFELs o fuentes de luz sincrotrón, se ha convertido en una herramienta notable en biología estructural, específicamente en el campo de las proteínas de membrana39. Sin embargo, se requieren miles de microcristales para generar suficientes datos para una solución de estructura robusta, lo cual no es una tarea fácil (al menos por los métodos de cristalización convencionales). El método de cristalización de diálisis descrito aquí permite la producción de un gran número de microcristales. Una vez que se ha determinado la condición de cristalización para la producción de microcristales (1-10 μm) mediante el uso de una placa de microdiálisis, se pueden producir grandes cantidades de microcristales de alta densidad utilizando el dispositivo dializador de 0,5 ml (Figura 5). Estos cristales son ideales para la recolección de datos utilizando objetivos fijos o sistemas de suministro de muestras de chorro de líquido27,40. Los cristales obtenidos a través de este método también pueden ser apropiados para aplicaciones MicroED. Sin embargo, es posible que sea necesario fresar a un tamaño y grosor adecuados para esta aplicación específica, ya que los electrones interactúan mucho más fuertemente con los cristales que los fotones de rayos X41.
En conclusión, el enfoque de cristalización por diálisis descrito aquí se suma a las estrategias evolutivas en la cristalización de proteínas para la determinación de la estructura y amplía el rango de esfuerzos que se pueden emplear para determinar nuevos objetivos de proteínas que anteriormente no han tenido éxito con otros métodos convencionales.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Reconocemos la financiación del Departamento de Negocios, Energía y Estrategia Industrial (BEIS) del Reino Unido. Agradecemos a Alex R. Jones y Mike Shaw del Laboratorio Nacional de Física por sus comentarios sobre el manuscrito.
Materials
| 0.2 mL tubes | Thermo Scientific | AB0620 | For aliquoting protein solutions. |
| 0.2 µm syringe filter | Sartorius | 17823———-K | Surfactant-free cellulose acetate filters. For filtering dialysis solutions. |
| 0.22 µm membrane filters | Millipore | GSTF04700 | Membrane filters for filtering large volumes of buffers |
| 12-channel, variable 0.5 – 10 µL Research plus pipette | Eppendorf | 3125000028 | For dispensing protein drops onto the Diaplate. |
| 12-channel, variable 30 – 300 µL Eppendorf Research plus pipette | Eppendorf | 3125000060 | For dispensing dialysis solutions on the Diaplate reservoirs. |
| 20 mL syringe | Fisherbrand | 15889152 | For use with syringe filters. |
| 96 well 2.2 mL deep-well plates | Thermo Scientific | AB0788 | Polypropylene deep-well storage plates; for preparing screens using the Hamilton Microlab STARlet. |
| Centrifuge 5425 | Eppendorf | 5405000565 | With rotor FA-24×2 with a maximum g-force of 21,300 x g. |
| Diacon dialyser | SWISSCI | W72010 | Dialyzer tubes with a regenerated cellulose membrane with a molecular weight cut-off of 10 kDa. Ideal for protein solutions of up to 0.5 mL. |
| Diaplate 96-well plate | SWISSCI | W82010 | Microdialysis plate. The Diaplate consists of two sides with a regenerated cellulose membrane in-between with a molecular weight cut-off of 10 kDa. |
| Falcon 50 mL High Clarity PP Centrifuge Tube | Corning | 352070 | For holding dialysis solutions. |
| Floating rack | SWISSCI | n/a | Included in the Diacon kit |
| Floor-standing vibration-free incubator | Molecular Dimensions | MD5-01 | 400 L temperature-controlled incubator set to 20 °C. |
| Leica M205 C stereo microscope | Leica | Planapo 1.0x objective, 7.8x – 160x zoom range with DMC 4500 camera | |
| Lysozyme from chicken egg white | Sigma Aldrich | 62971 | Lyophilized protein |
| Memgold2 | Molecular Dimensions | MD1-64 | Sparse-matrix screen |
| Microlab STARlet | Hamilton | n/a | Liquid handler system. |
| Reservoir cover film | SWISSCI | n/a | Included in the Diaplate kit |
| Reusable bottle top filter | Thermo Scientific | DS0320-5045 | For fitering large volumes of buffers, for use with 0.22 µm membrane filters |
| Sealing paddle | SWISSCI | n/a | Included in the Diaplate kit |
| Thaumatin from Thaumatococcus daniellii | Sigma Aldrich | T7638 | Lyophilized protein |
| UV cover film | SWISSCI | n/a | Included in the Diaplate kit |
References
- Brooks-Bartlett, J. C., Garman, E. F. The nobel science: One hundred years of crystallography. Interdisciplinary Science Reviews. 40 (3), 244-264 (2015).
- Maveyraud, L., Mourey, L. Protein X-ray crystallography and drug discovery. Molecules. 25 (5), (2020).
- Kwan, T. O. C., Axford, D., Moraes, I. Membrane protein crystallography in the era of modern structural biology. Biochemical Society Transactions. 48 (6), 2505-2524 (2020).
- Birch, J., et al. The fine art of integral membrane protein crystallisation. Methods. 147, 150-162 (2018).
- Gorrec, F., Löwe, J. Automated protocols for macromolecular crystallization at the MRC laboratory of molecular biology. Journal of Visualized Experiments. 131 (131), (2018).
- Govada, L., Chayen, N. E. Choosing the method of crystallization to obtain optimal results. Crystals. 9 (2), 106 (2019).
- Chayen, N. E. Turning protein crystallisation from an art into science. Current Opinion in Structural Biology. 14 (5), 577-583 (2004).
- McPherson, A., Gavira, J. A. Introduction to protein crystallization. Acta Crystallographica Section F: Structural Biology Communications. 70 (1), 2-20 (2014).
- Gulbis, J. Protein crystallography: methods and protocols. Crystallography Reviews. 24 (2), 136-143 (2018).
- D’Arcy, A., Bergfors, T., Cowan-Jacob, S. W., Marsh, M. Microseed matrix screening for optimization in protein crystallization: What have we learned. Acta Crystallographica Section:F Structural Biology Communications. 70 (9), 1117-1126 (2014).
- Shaw Stewart, ., D, P., Kolek, S. A., Briggs, R. A., Chayen, N. E., Baldock, P. F. Random microseeding: a theoretical and practical exploration of seed stability and seeding techniques for successful protein crystallization. Crystal Growth & Design. 11 (8), 3432-3441 (2011).
- Junius, N., et al. A microfluidic device for both on-chip dialysis protein crystallization and in situ X-ray diffraction. Lab on a Chip. 20 (2), 296-310 (2020).
- Junius, N., Vahdatahar, E., Oksanen, E., Ferrer, J. L., Budayova-Spano, M. Optimization of crystallization of biological macromolecules using dialysis combined with temperature control. Journal of Applied Crystallography. 53 (3), 686-698 (2020).
- Vahdatahar, E., Junius, N., Budayova-Spano, M. Optimization of crystal growth for neutron macromolecular crystallography. Journal of Visualized Experiments. 169, (2021).
- Jaho, S., et al. Crystallization of proteins on chip by microdialysis for in situ X-ray diffraction studies. Journal of Visualized Experiments. 170, (2021).
- Liu, W., Cherezov, V. Crystallization of membrane proteins in lipidic mesophases. Journal of Visualized Experiments. 49, 2501 (2011).
- Ujwal, R., Abramson, J. High-throughput crystallization of membrane proteins using the lipidic bicelle method. Journal of Visualized Experiments. 59, (2012).
- Parker, J. L., Newstead, S. Membrane protein crystallisation: Current trends and future perspectives. Advances in Experimental Medicine and Biology. 922. , 61-72 (2016).
- Apostolopoulou, V., Junius, N., Sear, R. P., Budayova-Spano, M. Mixing salts and polyethylene glycol into protein solutions: The effects of diffusion across semi-permeable membranes and of convection. Crystal Growth & Design. 20 (6), 3927-3936 (2020).
- Neutze, R., Wouts, R., Vander Spoel, D., Weckert, E., Hajdu, J. Potential for biomolecular imaging with femtosecond X-ray pulses. Nature. 406 (6797), 752-757 (2000).
- Chapman, H. N., et al. Femtosecond X-ray protein nanocrystallography. Nature. 470 (7332), 73-78 (2011).
- Mizohata, E., Nakane, T., Fukuda, Y., Nango, E., Iwata, S. Serial femtosecond crystallography at the SACLA: breakthrough to dynamic structural biology. Biophysical Reviews. 10 (2), 209-218 (2018).
- Nogly, P., et al. Lipidic cubic phase serial millisecond crystallography using synchrotron radiation. IUCrJ. 2 (2), 168-176 (2015).
- Johansson, L. C., et al. XFEL structures of the human MT2 melatonin receptor reveal the basis of subtype selectivity. Nature. 569 (7755), 289-292 (2019).
- Owen, R. L., et al. Low-dose fixed-target serial synchrotron crystallography. Acta Crystallographica Section D: Structural Biology. 73 (4), 373-378 (2017).
- Weinert, T., et al. Serial millisecond crystallography for routine room-temperature structure determination at synchrotrons. Nature communications. 8 (1), 542 (2017).
- Axford, D., et al. Two states of a light-sensitive membrane protein captured at room temperature using thin-film sample mounts. Acta Crystallographica Section D: Structural Biology. 78 (1), 52-58 (2022).
- Nannenga, B. L., Gonen, T. The cryo-EM method microcrystal electron diffraction (MicroED). Nature Methods. 16 (5), 369-379 (2019).
- Nguyen, C., Gonen, T. Beyond protein structure determination with MicroED. Current Opinion in Structural Biology. 64, 51-58 (2020).
- Mu, X., Gillman, C., Nguyen, C., Gonen, T. An overview of microcrystal electron diffraction (MicroED). Annual Review of Biochemistry. 90, 431-450 (2021).
- Kwan, T. O. C., et al. Selection of biophysical methods for characterisation of membrane proteins. International Journal of Molecular Sciences. 22 (10), 2605 (2019).
- Pos, K. M., Purification Diederichs, K. crystallization and preliminary diffraction studies of AcrB, an inner-membrane multi-drug efflux protein. Acta Crystallographica Section D: Biological Crystallography. 58 (10), 1865-1867 (2002).
- Guan, L., Mirza, O., Verner, G., Iwata, S., Kaback, H. R. Structural determination of wild-type lactose permease. Proceedings of the National Academy of Sciences. 104 (39), 15294-15298 (2007).
- Kwan, T. O. C., Reis, R., Moraes, I. In situ measurements of polypeptide samples by dynamic light scattering: membrane proteins, a case study. Methods in Molecular Biology. , 189-202 (2021).
- Seddon, A. M., Curnow, P., Booth, P. J. Membrane proteins, lipids and detergents: not just a soap opera. Biochimica et Biophysica Acta (BBA)-Biomembranes. (1-2), 105-117 (2004).
- Wickstrand, C., et al. A tool for visualizing protein motions in time-resolved crystallography. Structural Dynamics. 7 (2), 024701 (2020).
- Orville, A. M. Recent results in time resolved serial femtosecond crystallography at XFELs. Current Opinion in Structural Biology. 65, 193-208 (2020).
- Schulz, E. C., Yorke, B. A., Pearson, A. R., Mehrabi, P. Best practices for time-resolved serial synchrotron crystallography. Acta Crystallographica Section D: Structural Biology. 78 (1), 14-29 (2022).
- Neutze, R., Brändén, G., Schertler, G. F. Membrane protein structural biology using X-ray free eletron lasers. Current Opinion in Structural Biology. 33, 115-125 (2015).
- Wierstall, U. Liquid sample delivery techniques for serial femtosecond crystallography. Philosophical Transactions of the Royal Society B: Biological Sciences. 369 (1647), 20130337 (2014).
- Shi, D., Nannenga, B. L., Iadanza, M. G., Gonen, T. Three-dimensional electron crystallography of protein microcrystals. eLife. 2, 01345 (2013).

