Sistema de detección de virus de ADN basado en RPA-CRISPR/Cas12A-SPM y Deep Learning
Summary
Presentamos un protocolo que combina la amplificación de la polimerasa recombinasa con un sistema CRISPR/Cas12a para la detección de trazas de virus de ADN y construye una microscopía portátil para teléfonos inteligentes con una clasificación asistida por inteligencia artificial para la detección de virus de ADN en el punto de atención.
Abstract
Presentamos un sistema de detección de virus de ADN rápido, fácil de implementar, altamente sensible, específico de secuencia y en el punto de atención (POC), que combina la amplificación de la polimerasa recombinasa (RPA) y el sistema CRISPR/Cas12a para la detección de trazas de virus de ADN. El ADN objetivo es amplificado y reconocido por RPA y CRISPR/Cas12a por separado, lo que desencadena la actividad de escisión colateral de Cas12a que escinde un reportero de ADN marcado con extintores de fluoróforos y generaliza la fluorescencia. Para la detección de POC, la microscopía portátil de los teléfonos inteligentes está diseñada para tomar imágenes fluorescentes. Además, se despliegan dentro del sistema modelos de aprendizaje profundo para la clasificación binaria de muestras positivas o negativas, logrando una alta precisión. El virus de la rana 3 (FV3, géneros Ranavirus, familia Iridoviridae) se probó como ejemplo para este sistema de detección POC de virus de ADN, y los límites de detección (LoD) pueden alcanzar 10 aM en 40 minutos. Sin operadores cualificados ni instrumentos voluminosos, el RPA-CRISPR/Cas12a-SPM portátil y en miniatura con clasificación asistida por inteligencia artificial (IA) muestra un gran potencial para la detección de virus POC DNA y puede ayudar a prevenir la propagación de dichos virus.
Introduction
En los últimos años, se han producido con frecuencia epidemias de enfermedades infecciosas causadas por diferentes virus, como la epidemia de enfermedad por el virus del Ébola (EVE) en 20141 y 20182, el síndrome respiratorio de Oriente Medio (MERS) en 20153, la epidemia de enfermedad por el virus del Zika en 20154, la enfermedad por coronavirus 2019 (COVID-19) causada por el coronavirus 2 del síndrome respiratorio agudo grave (SARS-CoV-2)5 y la continuación de la viruela del mono causada por el virus de la viruela del mono (MKPV) en 20226. Estos brotes repentinos de enfermedades infecciosas epidémicas causan un gran número de muertes y traen enormes pérdidas económicas y disturbios sociales. Se requiere urgentemente un sistema de detección rápido y preciso para diagnosticar rápidamente la infección y evitar una mayor propagación del virus.
Recientemente, las repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR) y las proteínas asociadas a CRISPR (Cas) han ganado atención mundial y han mostrado resultados prometedores en la detección de ácidos nucleicos 7,8,9,10,11,12,13,14,15 . La proteína CRISPR/Cas12a, guiada por el ARN CRISPR (crRNA), se une y escinde el ADN diana. Esta actividad conduce a la liberación de ADN monocatenario inespecífico (ssDNA), conocido como trans-escisiones, y se puede utilizar para mejorar la señal de detección para la detección de ácidos nucleicos. Algunos métodos de detección tradicionales, como la reacción en cadena de la polimerasa (PCR), la PCR cuantitativa en tiempo real (qPCR) y el ensayo de inmunoabsorción enzimática (ELISA), son complicados, lentos y costosos para la detección en el punto de atención (POC). Nuestro trabajo anterior desarrolló con éxito un sistema de detección automatizado, integrado y rentable para el virus de la peste porcina africana (PPA) basado en la tecnología CRISPR/Cas12a. En este sistema, logramos un límite de detección de 1 pM en un período de tiempo de 2 horas sin necesidad de amplificación. El sistema CRISPR/Cas12a y la amplificación de la polimerasa recombinasa (RPA) se combinan para mejorar la sensibilidad y la especificidad de la detección de trazas de ADN. En comparación con otras técnicas de amplificación isotérmica, RPA tiene un diseño simple y una operación conveniente, ya que tiene un tiempo de reacción más corto sin un equipo sofisticado de control de temperatura.
Para la detección de patógenos POC, se desarrollan instrumentos como la microscopía de teléfono inteligente (SPM), el fluorímetro de mano o las tiras de flujo lateral para las lecturas de resultados 16,17,18. SPM captura imágenes a través de una cámara y las sube a algunas aplicaciones móviles para un análisis rápido de los datos. Dicha microscopía es un sistema de adquisición de señales portátil, barato y en miniatura con alta sensibilidad y ha demostrado ventajas en la detección de patógenos como el H5N1, el virus del Zika y el SARS-CoV-219,20. Por lo tanto, construimos un SPM portátil para captar las señales de fluorescencia desencadenadas por la detección de RPA-CRISPR/Cas12a del virus de ADN objetivo. La sonda reportera de ssDNA que conecta un fluoróforo y un extintor, se escindirá cuando CRISPR/Cas12a reconozca el virus de ADN objetivo, y la fluorescencia emitida por el fluoróforo pueda ser capturada por SPM.
En comparación con el software profesional que se suele utilizar para obtener la información de los resultados de las imágenes de fluorescencia de SPM21, algunos expertos utilizan el aprendizaje automático y el aprendizaje profundo para cuantificar las concentraciones de ADN del virus después de obtener imágenes de fluorescencia22, lo que requiere más tiempo. Cuando se trata de clasificar imágenes médicas, las redes neuronales convencionales (CNN) se utilizan a menudo para aprender características de las imágenes pixeladas en bruto de manera integral 23,24,25,26. Los modelos populares de aprendizaje profundo basados en CNN, como AlexNet, DenseNet-121 y EfficientNet-B7, se han aplicado con éxito en este campo27,28. Sin embargo, la obtención de grandes conjuntos de datos en dominios específicos puede ser un desafío, lo que requiere un aprendizaje por transferencia29,30. Este enfoque entrena previamente un modelo de aprendizaje profundo con un conjunto de datos grande, y el modelo entrenado previamente se usa como punto de partida para una nueva tarea con un conjunto de datos pequeño. Esta técnica puede reducir la necesidad de grandes conjuntos de datos, combatir el sobreajuste y reducir el tiempo de entrenamiento31. En este trabajo, utilizamos modelos de aprendizaje profundo con aprendizaje por transferencia para la clasificación binaria de las imágenes de fluorescencia de las muestras positivas y negativas.
En este método, combinamos RPA y el sistema CRISPR/Cas12a para la detección de trazas de virus de ADN. El ADN objetivo es amplificado y reconocido por RPA y CRISPR/Cas12a por separado, lo que desencadena la actividad de escisión colateral de Cas12a que escinde un reportero de ADN marcado con extintores de fluoróforos y generaliza la fluorescencia. Construimos un SPM portátil para tomar las imágenes fluorescentes para la detección de POC y desarrollamos modelos de aprendizaje profundo para la clasificación binaria. El esquema del sistema de detección POC incorporado se muestra en la Figura 1. Sin operadores cualificados e instrumentos voluminosos, la clasificación asistida por RPA-CRISPR/Cas12a-SPM con inteligencia artificial (IA) muestra un gran potencial para la detección de virus POC ADN.
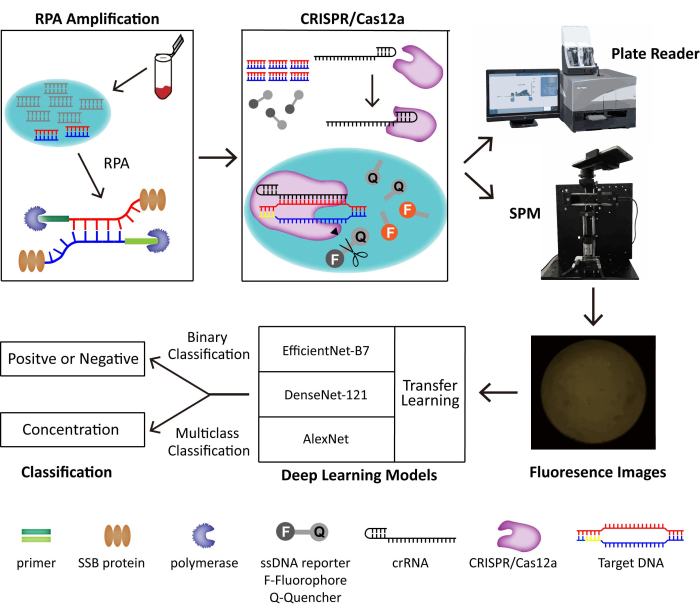
Figura 1: Esquema del sistema de detección RPA-CRISPR/Cas12-SPM junto con la clasificación de IA para las imágenes recopiladas. Los ácidos nucleicos de las muestras de origen animal son liberados por PINDBK. El ADN diana del virus es amplificado y reconocido específicamente por el sistema RPA-CRISPR/Cas12a. CRISPR/Cas12a se une con el crRNA y el complejo Cas12a-crRNA se une con el ADN objetivo, lo que desencadena la escisión colateral de CRISPR/Cas12a en las sondas reporteras de ssDNA. El fluoróforo en el reportero se libera y la fluorescencia es detectada por un lector de placas comercializado o el SPM que construimos. Se utilizan tres modelos diferentes de aprendizaje profundo, incluidos AlexNet, DenseNet-121 y EfficientNet-B7 con aprendizaje por transferencia, para clasificar las imágenes de fluorescencia. Esta figura es reutilizada con permiso de Lei et al.35. Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
En este método, desarrollamos un sistema de detección de virus de ADN POC rápido, fácil de implementar, altamente sensible, específico de secuencia y POC con asistencia de IA. Después de obtener las muestras, se aplica RPA para amplificar la secuencia objetivo, y luego CRISPR/Cas12a puede reconocer el ADN objetivo y liberar fluorescencia, lo que amplía la señal de detección. La microscopía portátil de los teléfonos inteligentes está diseñada para tomar imágenes de fluorescencia, y los modelos de aprendizaj…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Este trabajo cuenta con el apoyo de la Fundación Nacional de Ciencias Naturales de China 31970752, Ciencia, Tecnología e Innovación de la Comisión de JCYJ20190809180003689, JSGG20200225150707332, JSGG20191129110812708, WDZC20200820173710001; Financiación abierta del Laboratorio de la Bahía de Shenzhen, SZBL2020090501004; Fundación de Ciencias Postdoctorales de China 2020M680023; y Administración General de Aduanas de la República Popular China 2021HK007.
Materials
| 20x amplification | OLYMPUS | OPLN20X | |
| 532 nm green laser | Thorlabs | PL201 | with 0.9 mW output power |
| 535 nm cutoff wavelength | chrome | AT535 | |
| 6x DNA loading buffer | Thermo scientific | R0611 | |
| 96-well black microplate | Corning Incorporated | 3603 | Black with flat clear bottom |
| Aspherical lens | Lubang | N/A | |
| Bandpass filter | SEMROCK | FF01-542/27-25 | |
| Bsu DNA Polymerase | ATG Biotechnology | M103 | Large Fragment |
| crRNA | Sangon Biotech | N/A | |
| DNA fragments | Sangon Biotech | N/A | |
| Dichroic holders | Ruicage | N/A | |
| Dichroic mirror | SEMROCK | FF555-Di03-25×36 | with a cutoff wavelength of 535 nm |
| E.Z.N.A Gel Extraction Kit | Omega Biotek | D2500-02 | |
| EnGen Lba Cas12a (Cpf1) | New England Biolabs (Beijing) LTD | M0653T | |
| Filter holders | Ruicage | N/A | |
| Fluorophore-ssDNA-Quencher reporter probes | Sangon Biotech | N/A | TAMRA (carboxy tetramethylrhodamine) as the fluorophore at the 5 ends; BHQ2 (Black Hole Quencher-2) as the quencher at the 3 ends |
| GP32 | ATG Biotechnology | M104 | |
| ImageJ | Open-source | Version 1.53t 24 | Downloaded from https://imagej.nih.gov/ij/ |
| Microplate reader | SPARK, TECAN | N/A | |
| Multi-Block thermal Cycler PCR instrument | LongGene | N/A | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Scientific | ND-2000 | |
| NEBuffer r2.1 | New England Biolabs (Beijing) LTD | B6002S | 10x CRISPR/Cas12a Reaction buffer |
| Oxygen plasma treatment | Electro-Technic Products | N/A | |
| Pathogen Inactivate, Nucleic acid extraction-free, Direct-to-PCR Buffer with Proteinase K (PINDBK) | Ebio | PINDBK -25mL | |
| PCR primer pairs | Sangon Biotech | N/A | |
| PDMS | Dow Corning | Sylgard 184 | |
| RPA primer pairs | Sangon Biotech | N/A | |
| Smartphone | Huawei | Mate10 | |
| Translation stages | Ruicage | N/A | |
| Transmitted neutral density filters | Thorlabs | ND40A | |
| Triplet achromatic lenses | Thorlabs | TRH127-020-A | |
| UvsX | ATG Biotechnology | M105 | |
| UvsY | ATG Biotechnology | M106 |
References
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345 (6202), 1369-1372 (2014).
- The Ebola Outbreak Epidemiology Team. Outbreak of Ebola virus disease in the Democratic Republic of the Congo, April-May 2018: an epidemiological study. Lancet. 392 (10143), 213-221 (2018).
- Zumla, A., Hui, D. S., Perlman, S. Middle East respiratory syndrome. Lancet. 386 (9997), 995-1007 (2015).
- Plourde, A. R., Bloch, E. M. A literature review of Zika virus. Emerg Infect Dis. 22 (7), 1185-1192 (2016).
- Yuan, X., et al. Current and perspective diagnostic techniques for COVID-19. ACS Infect Dis. 6 (8), 1998-2016 (2020).
- Minhaj, F. S., et al. Monkeypox outbreak – nine states, May 2022. MMWR Morb Mortal Wkly Rep. 71 (23), 764-769 (2022).
- Bao, M., et al. Challenges and opportunities for clustered regularly interspaced short palindromic repeats based molecular biosensing. ACS Sens. 6 (7), 2497-2522 (2021).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nat Biotechnol. 38 (7), 870-874 (2020).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), 436-439 (2018).
- Gootenberg, J. S., et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 356 (6336), 438-442 (2017).
- Kellner, M. J., Koob, J. G., Gootenberg, J. S., Abudayyeh, O. O., Zhang, F. SHERLOCK: nucleic acid detection with CRISPR nucleases. Nat Protoc. 14 (10), 2986-3012 (2019).
- Mukama, O., et al. An ultrasensitive and specific point-of-care CRISPR/Cas12 based lateral flow biosensor for the rapid detection of nucleic acids. Biosens Bioelectron. 159, 112143 (2020).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Yin, L., Man, S., Ye, S., Liu, G., Ma, L. CRISPR-Cas based virus detection: Recent advances and perspectives. Biosens Bioelectron. 193, 113541 (2021).
- Dronina, J., Bubniene, U. S., Ramanavicius, A. The application of DNA polymerases and Cas9 as representative of DNA-modifying enzymes group in DNA sensor design (review). Biosens Bioelectron. 175, 112867 (2021).
- Fozouni, P., et al. Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy. Cell. 184 (2), 323-333.e9 (2021).
- Kumar, M., et al. FnCas9-based CRISPR diagnostic for rapid and accurate detection of major SARS-CoV-2 variants on a paper strip. eLife. 10, e67130 (2021).
- Lee, R. A., et al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria. Proc Natl Acad Sci U S A. 117 (41), 25722-25731 (2020).
- Ganguli, A., et al. Hands-free smartphone-based diagnostics for simultaneous detection of Zika, Chikungunya, and Dengue at point-of-care. Biomed Microdevices. 19 (4), 73 (2017).
- Yeo, S. J., et al. Smartphone-based fluorescent diagnostic system for highly pathogenic H5N1 viruses. Theranostics. 6 (2), 231-242 (2016).
- von Chamier, L., et al. Democratising deep learning for microscopy with ZeroCostDL4Mic. Nat Commun. 12 (1), 2276 (2021).
- Shiaelis, N., et al. Virus detection and identification in minutes using single-particle imaging and deep learning. ACS Nano. 17 (1), 697-710 (2020).
- Liu, Y., et al. Mixed-UNet: Refined class activation mapping for weakly-supervised semantic segmentation with multi-scale inference. Front. Comput. Sci. 4, 1036934 (2022).
- Lawrimore, J., Doshi, A., Walker, B., Bloom, K. AI-assisted forward modeling of biological structures. Front Cell Dev Biol. 7, 279 (2019).
- Yang, Y., Hu, Y., Zhang, X., Wang, S. Two-stage selective ensemble of CNN via deep tree training for medical image classification. IEEE Trans Cybern. 52 (9), 9194-9207 (2022).
- Zhang, R., et al. RCMNet: A deep learning model assists CAR-T therapy for leukemia. Comput Biol Med. 150, 106084 (2022).
- Xie, Y., et al. Stroke prediction from electrocardiograms by deep neural network. Multimed Tools Appl. 80, 17291-17297 (2021).
- Wang, J., Zhu, H., Wang, S., Zhang, Y. -. D. A review of deep learning on medical image analysis. Mobile Netw Appl. 26, 351-380 (2021).
- Artoni, P., et al. Deep learning of spontaneous arousal fluctuations detects early cholinergic defects across neurodevelopmental mouse models and patients. Proc Natl Acad Sci U S A. 117 (38), 23298-23303 (2020).
- Li, J., et al. DeepLearnMOR: a deep-learning framework for fluorescence image-based classification of organelle morphology. Plant Physiol. 186 (4), 1786-1799 (2021).
- Yosinski, J., Clune, J., Bengio, Y., Lipson, H. How transferable are features in deep neural networks. Proceedings of the 27th International Conference on Neural Information Processing Systems. 2, 3320-3328 (2014).
- He, Q., et al. High-throughput and all-solution phase African Swine Fever Virus (ASFV) detection using CRISPR-Cas12a and fluorescence based point-of-care system. Biosens Bioelectron. 154, 112068 (2020).
- Krizhevsky, A., Sutskever, I., Hinton, G. E. ImageNet classification with deep convolutional neural networks. Commun. ACM. 60 (6), 84-90 (2017).
- Lawton, S., Viriri, S. Detection of COVID-19 from CT lung scans using transfer learning. Comput Intell Neurosci. 2021, 5527923 (2021).
- Lei, Z., et al. Detection of frog virus 3 by integrating RPA-CRISPR/Cas12a-SPM with deep learning. ACS Omega. 8 (36), 32555-32564 (2023).
- Chen, Z., Huang, J., Zhang, F., Zhou, Y., Huang, H. Detection of shrimp hemocyte iridescent virus by recombinase polymerase amplification assay. Mol Cell Probes. 49, 101475 (2020).
- Fu, X., Sun, J., Ye, Y., Zhang, Y., Sun, X. A rapid and ultrasensitive dual detection platform based on Cas12a for simultaneous detection of virulence and resistance genes of drug-resistant Salmonella. Biosens Bioelectron. 195, 113682 (2022).
- Habimana, J. D., et al. Mechanistic insights of CRISPR/Cas nucleases for programmable targeting and early-stage diagnosis: A review. Biosens Bioelectron. 203, 114033 (2022).
- Liang, Y., Lin, H., Zou, L., Deng, X., Tang, S. Rapid detection and tracking of Omicron variant of SARS-CoV-2 using CRISPR-Cas12a-based assay. Biosens Bioelectron. 205, 114098 (2022).
- Sivaraman, D., Biswas, P., Cella, L. N., Yates, M. V., Chen, W. Detecting RNA viruses in living mammalian cells by fluorescence microscopy. Trends Biotechnol. 29 (7), 307-313 (2011).
- Wang, I. H., Burckhardt, C. J., Yakimovich, A., Greber, U. F. Imaging, tracking and computational analyses of virus entry and egress with the cytoskeleton. Viruses. 10 (4), 166 (2018).

