물 샘플에서 Leptospira 검출을 위한 중합효소 연쇄 반응 및 점-블롯 혼성화
Summary
이 연구에서는 물 샘플의 세 가지 주요 clade에서 Leptospira 를 검출하도록 dot-blot 응용 프로그램을 설계했습니다. 이 방법을 사용하면 항-디곡시제닌 항체에 의해 쉽게 검출되는 디곡시제닌 표지 프로브에 의해 특이적으로 표적화된 최소 DNA 양을 식별할 수 있습니다. 이 접근 방식은 스크리닝 목적에 유용하고 만족스러운 도구입니다.
Abstract
도트 블롯은 운반체 DNA가 있는 상태에서 프로브 교잡에 의해 특이적으로 표적이 되는 최소한의 DNA를 식별할 수 있는 간단하고 빠르며 민감하고 다재다능한 기술입니다. 이는 알려진 양의 DNA를 나일론 멤브레인과 같은 불활성 고체 지지체에 전달하여 도트 블롯 장치를 사용하고 전기 영동 분리 없이 수행하는 것을 기반으로 합니다. 나일론 멤브레인은 높은 핵산 결합력(400 μg/cm2), 고강도의 장점이 있으며 양전하 또는 중성으로 대전됩니다. 사용된 프로브는 디곡시게닌(digoxigenin, DIG)으로 길게 표지된 18-20개의 염기로 구성된 매우 특이적인 ssDNA 단편입니다. 프로브는 렙토스피라 DNA와 결합합니다. 프로브가 표적 DNA와 하이브리드화되면 항-디고시제닌 항체에 의해 검출되어 X선 필름에 드러난 방출을 통해 쉽게 검출할 수 있습니다. 방출이 있는 점은 관심 DNA 조각에 해당합니다. 이 방법은 프로브의 비동위원소 라벨링을 사용하며, 이는 매우 긴 반감기를 가질 수 있습니다. 이 표준 면역 표지의 단점은 동위원소 프로브보다 민감도가 낮다는 것입니다. 그럼에도 불구하고 중합효소 연쇄 반응(PCR)과 도트 블롯 분석을 결합하여 완화됩니다. 이 접근 방식을 통해 표적 염기서열과 검출을 강화할 수 있습니다. 또한, 잘 알려진 표준물질의 연속 희석과 비교할 때 정량적 응용 분야로 사용될 수 있습니다. 물 샘플의 세 가지 주요 clades에서 Leptospira 를 검출하기 위한 dot-blot 응용 프로그램이 여기에 제시되어 있습니다. 이 방법론은 렙토스피나선형 DNA의 존재에 대한 증거를 제공하기 위해 원심분리에 의해 농축된 많은 양의 물에 적용할 수 있습니다. 이것은 일반적인 스크리닝 목적을 위한 가치 있고 만족스러운 도구이며, 물에 존재할 수 있는 다른 배양 불가능한 박테리아에 사용할 수 있어 생태계에 대한 이해를 높일 수 있습니다.
Introduction
인간의 렙토스피라증은 주로 환경적 요인에 의해 발생한다 1,2. 호수, 강 및 하천에 렙토스피라가 존재한다는 것은 야생 동물과 결국 이러한 수역과 접촉할 수 있는 가축 및 생산 동물 사이에서 렙토스피라증이 전염된다는 지표입니다 1,3,4. 또한, 렙토스피라(Leptospira)는 하수, 정체 및 수돗물을 포함한 비자연적 자원에서 확인되었습니다 5,6.
렙토스피라는 전 세계에 분포하는 박테리아 7,8이며, 보존 및 전파에 있어 환경의 역할은 잘 알려져 있습니다. 렙토스피라(Leptospira)는 pH와미네랄이 다양한 음용수9와자연 수역1에서 생존할 수 있다. 또한 증류수(10)에서 장기간 생존할 수 있으며, 일정한 pH(7.8)에서는 최대 152일까지 생존할 수 있다11. 더욱이, 렙토스피라(Leptospira)는 가혹한 조건에서도 생존하기 위해 박테리아 컨소시엄(bacterial consortium)에서 상호작용할 수 있다12,13. 그것은 Azospirillum 및 Sphingomonas와 함께 담수에서 생물막의 일부일 수 있으며 49 °C14,15를 초과하는 온도를 성장시키고 견딜 수도 있습니다. 또한 물에 잠긴 토양에서 증식할 수 있으며 최대 379일(16) 동안 생존할 수 있으며, 1년 동안 질병을 유발할 수 있는 능력을 보존할 수 있습니다17,18. 그러나 수역 내의 생태와 수역 내에 어떻게 분포되어 있는지에 대해서는 알려진 바가 거의 없습니다.
발견 이후 Leptospira 속에 대한 연구는 혈청 학적 검사를 기반으로했습니다. 현재 세기가 되어서야 분자 기술이이 spirochaete의 연구에서 더 널리 퍼졌습니다. 점-블롯은 (1) 16S rRNA 및 ISSR(Inter-Simple Sequence Repeat)19,20을 기반으로 하는 동위원소 프로브를 사용하거나, (2) 소변21에 적용되는 인간 렙토스피라증에 대한 나노골드 기반 면역분석법으로, 또는 (3) 소 소변 샘플22에 대한 항체 기반 분석법으로 사용하는 식별에 거의 사용되지 않았습니다. 이 기술은 원래 동위원소 프로브를 기반으로 했기 때문에 사용되지 않았습니다. 그러나 PCR과 결합하여 향상된 결과를 얻을 수 있는 잘 알려진 기술이며 비동위원소 프로브를 사용하기 때문에 안전한 것으로 간주됩니다. PCR은 샘플의 미량에서 발견될 수 있는 특정 DNA 단편을 증폭하여 Leptospira DNA의 농축에 중요한 역할을 합니다. 각 PCR 주기 동안 표적 DNA 단편의 양은 반응에서 두 배가 됩니다. 반응이 끝날 때, 앰플리콘은 100만23 이상의 계수로 곱해졌다. PCR에 의해 증폭된 생성물은 아가로스 전기영동에서 종종 보이지 않지만, 도트 블롯24,25,26에서 DIG 표지 프로브와의 특정 혼성화를 통해 보이게 됩니다.
도트 블롯 기법은 간단하고 견고하며 수많은 샘플에 적합하므로 자원이 제한된 실험실에서 사용할 수 있습니다. 이는 (1) 구강 세균27, (2) 식품 및 배설물과 같은 다른 샘플 유형(28), (3) 배양 불가능한 세균29의 식별을 포함한 다양한 박테리아 연구에 사용되어 왔으며, 종종 다른 분자 기술과 일치합니다. 도트 블롯 기법이 제공하는 장점은 다음과 같습니다 : (1) 멤브레인은 200 μg / cm2 이상의 핵산 및 최대 400 μg / cm2 와 결합 할 수있는 높은 결합 능력을 가지고 있습니다. (2) 도트 블롯 결과는 특별한 장비 없이 시각적으로 해석할 수 있으며 (3) 실온(RT)에서 수년 동안 편리하게 보관할 수 있습니다.
렙토스피라(Leptospira) 속은 병원성, 중간성, 부생식물군(saprophytic clades)30,31로 분류되었다. 이러한 clade 간의 구별은 lipL41, lipL32 및 16S rRNA와 같은 특정 유전자를 기반으로 달성할 수 있습니다. LipL32는 병원성 clades에 존재하며 다양한 혈청학적 및 분자 도구에서 높은 민감도를 나타내는 반면, 부생식물 종21에는 없습니다. 하우스키핑 유전자 lipL41은 안정적인 발현으로 알려져 있으며 분자 기술(32)에 사용되는 반면, 16S rRNA 유전자는 분류에 활용됩니다.
이 방법론은 원심분리에 의해 농축된 많은 양의 물에 적용할 수 있습니다. 이를 통해 수역 내의 다양한 지점과 깊이를 평가하여 렙토스피럴 DNA의 존재와 그것이 속한 클래드를 감지할 수 있습니다. 이 도구는 생태학적 및 일반 스크리닝 목적 모두에 유용하며 물에 존재할 수 있는 다른 배양 불가능한 박테리아를 감지하는 데에도 사용할 수 있습니다.
또한 PCR 및 도트 블롯 분석은 정교하거나 고가의 장비가 없는 실험실을 포함하여 다양한 실험실에서 기술적으로나 경제적으로 저렴합니다. 이 연구는 자연 수역에서 수집된 물 샘플에서 3개의 렙토스피라 군을 식별하기 위해 디곡시게닌 기반 점-블롯을 적용하는 것을 목표로 합니다.
박테리아 균주
이 연구에는 12종의 렙토스피라 혈청형(Autumnalis, Bataviae, Bratislava, Canicola, Celledoni, Grippothyphosa, Hardjoprajitno, Icterohaemorrhagiae, Pomona, Pyrogenes, Tarassovi, Wolffi)이 포함되었습니다. 이 혈청형은 멕시코 국립 자치 대학교 수의학 및 동물원 학부의 미생물학 및 면역학과 컬렉션의 일부이며 현재 미세 응집 검사(MAT)에 사용되고 있습니다.
모든 렙토스피라 혈청형을 EMJH에서 배양하고, 이들의 DNA를 상업용 DNA 추출 키트를 사용하여 추출하였다( 재료 표 참조). 12개의 혈청형의 게놈 DNA 혼합물을 렙토스피라 병원성 군집에 대한 양성 대조군으로 사용했습니다. 렙토스피라 중간 군집의 양성 대조군으로, 렙토스피라 페이네이 혈청형 허스트브리지 균주 BUT6로부터의 게놈 DNA를 포함하였고, 렙토스피라 부생식물 군집에 대한 양성 대조군으로서, 렙토스피라 biflexa serovar Patoc 균주 Patoc I의 게놈 DNA도 포함하였다.
음성 대조군은 빈 플라스미드, 관련 없는 박테리아(Ureaplasma urealyticum, Staphylococcus aureus, Brucella abortus, Salmonella typhimurium, Shigella boydii, Klebsiella pneumoniae, Acinetobacter baumannii, Escherichia coli)의 DNA, 그리고 비템플릿 대조군으로 작용한 PCR 등급의 물로 구성되었습니다.
물 샘플
Cuemanco Biological and Aquaculture Research Center (CIBAC) (19° 16′ 54″ N 99° 6′ 11″ W)에서 층화 우연 샘플링 방법을 사용하여 12개의 시험 채취 샘플을 수집했습니다. 이 샘플은 표층, 10cm 및 30cm의 세 가지 깊이에서 획득되었습니다(그림 1A, B). 물 수집 절차는 멸종 위기에 처한 종이나 보호 종에 영향을 미치지 않았습니다. 각 샘플은 멸균된 15mL 미세 원심분리기 튜브에서 수집되었습니다. 샘플을 수집하기 위해 각 튜브를 물에 부드럽게 담그고 선택한 깊이로 채운 다음 밀봉했습니다. 샘플은 22°C에서 유지되었으며 처리를 위해 즉시 실험실로 운반되었습니다.
각 샘플은 실온에서 20분 동안 8000 x g 의 멸균 1.5mL 미세 원심분리기 튜브에서 원심분리하여 농축했습니다. 이 단계를 반복하여 모든 샘플이 하나의 튜브에 농축될 때까지 이 튜브를 DNA 추출에 사용했습니다(그림 1C).
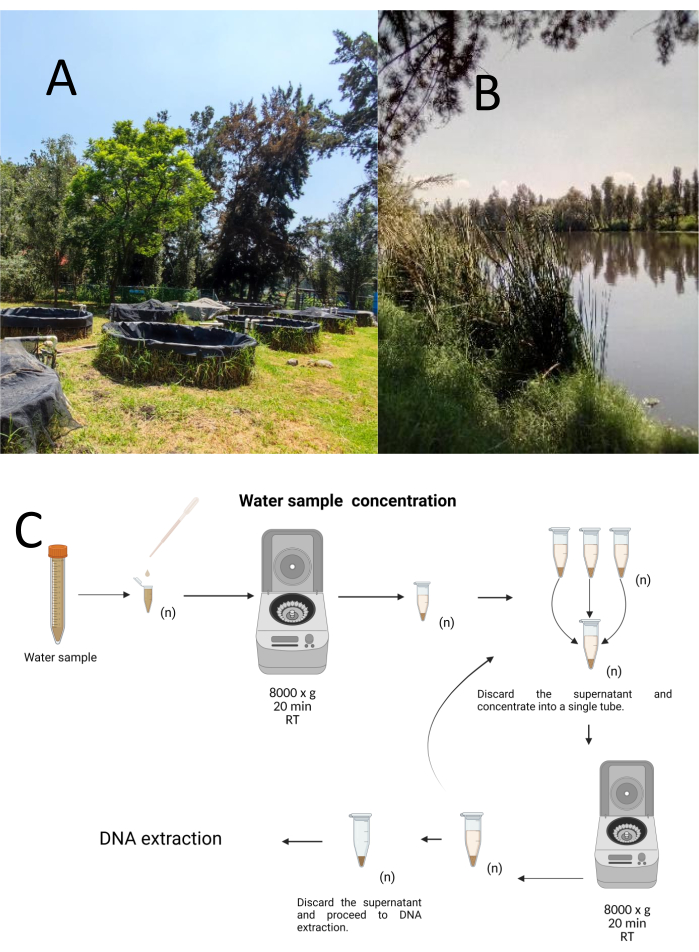
그림 1: 원심분리에 의한 물 샘플의 농도. (A) 물 샘플링 연못, (B) 자연 하천. (C) 원심분리 기반 물 샘플 처리는 필요한 만큼 반복적인 단계로 (n). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
DNA 추출
총 DNA는 제조업체의 지침에 따라 상용 Genomic DNA 키트를 사용하여 분리하였다( 재료 표 참조). DNA 추출은 20 μL의 용출 완충액에서 용출되고, DNA 농도는 260-280 nm에서 UV 분광 광도계로 측정하고, 사용할 때까지 4 °C에서 보관하였다.
PCR 증폭
PCR 표적은 16개의S rRNA, lipL41 및 lipL32 유전자로, Leptospira 속의 DNA를 식별하고 병원성, 부생성 및 중간성의 세 가지 분류를 구별할 수 있습니다. 프라이머와 프로브 설계는 모두 Ahmed et al., Azali et al., Bourhy et al., Weiss et al. 및 Branger et al.33,34,35,36,37의 이전 작업을 기반으로 했습니다. 각 프로브, 프라이머 및 증폭된 단편의 서열은 표 1에 기재되어 있으며, 참조 서열과의 정렬은 보충 파일 1, 보충 파일 2, 보충 파일 3, 보충 파일 4 및 보충 파일 5에 제공되어 있습니다. PCR 시약 및 열순환 조건은 프로토콜 섹션에 설명되어 있습니다.
증폭 산물은 보충 그림 1에 표시된 바와 같이 에티듐-브로마이드 검출과 함께 60V에서 45분 동안 TAE(40mM Tris 염기, 20mM 아세트산 및 1mM EDTA, pH 8.3)의 1% 아가로스 겔에서 전기영동 분리를 통해 시각화했습니다. 각 혈청형에서 수득한 게놈 DNA는 병원성 렙토스피라의 경우 L. interrogans (4, 691, 184 bp)38의 게놈 크기, 부생식물성 렙토스피라의 경우 L. biflexa(3, 956, 088 bp)39의 게놈 크기를 기준으로 각 PCR 반응에서 6 x 106 내지 1 x 104 게놈 등가 사본(GEq) 범위의 농도로 사용되었으며, 및 등록 번호 AKWZ00000000.2를 가진 L. fainei serovar Hurstbridge 균주 BUT6 (4, 267, 324 bp)의 게놈 크기.
프로브의 민감도는 각 실험에서 각 병원성 혈청형, L. biflexa serovar Patoc 균주 Patoc I 및 L. fainei 혈청형 Hurstbridge 균주 BUT6의 DNA로 평가했습니다. PCR 및 점-블롯 혼성화 분석의 특이성을 평가하기 위해 비관련 박테리아의 DNA를 포함했습니다.
표 1: Leptospira의 병원성, 부생식물 및 중간 clades를 식별하기 위한 제품을 증폭하기 위한 PCR 프라이머 및 프로브. 이 표를 다운로드하려면 여기를 클릭하십시오.
Dot-blot hybridization 어세이
이 기술은 DNA 샘플이 놓인 구멍이 점 모양을 가지고 있기 때문에 도트 블롯이라고 불리며, 진공 흡입에 의해 제자리에 고정되도록 흡입되면 이러한 모양을 얻습니다. 이 기술은 Kafatos et al.40에 의해 개발되었습니다. 이 기술을 사용하면 각 PCR 양성 샘플에서 Leptospira의 반 정량화가 가능합니다. 이 프로토콜은 실온에서 NaOH 0.4M를 사용한 변성으로 구성되며, 6 x 106 to 1 x 104 leptospires에 해당하는 30ng – 0.05ng의 Leptospira DNA가 있는 샘플은 96웰 도트 블롯 장치를 사용하여 나일론 멤브레인에 블롯됩니다. 고정화 후 DNA는 120mJ 자외선에 노출되어 멤브레인에 결합됩니다. 각 DNA 프로브는 3′ 말단에서 말단 전이효소 촉매 단계에 의해 digoxigenin-11 dUTP와 접합됩니다(Digoxigenin은 리포터41로 사용되는 Digitalis purpurea에서 얻은 식물 스테로이드입니다). 특정 온도에서 라벨링된 DNA 프로브(50 pmol)를 타겟 DNA에 엄격하게 혼성화한 후, DNA 하이브리드는 기질 CSPD와 공유 결합된 항-디고시게닌 알칼리 인산가수분해효소 항체와의 화학발광 반응에 의해 시각화됩니다. 발광은 X선 필름에 노출되어 캡처됩니다(그림 2).
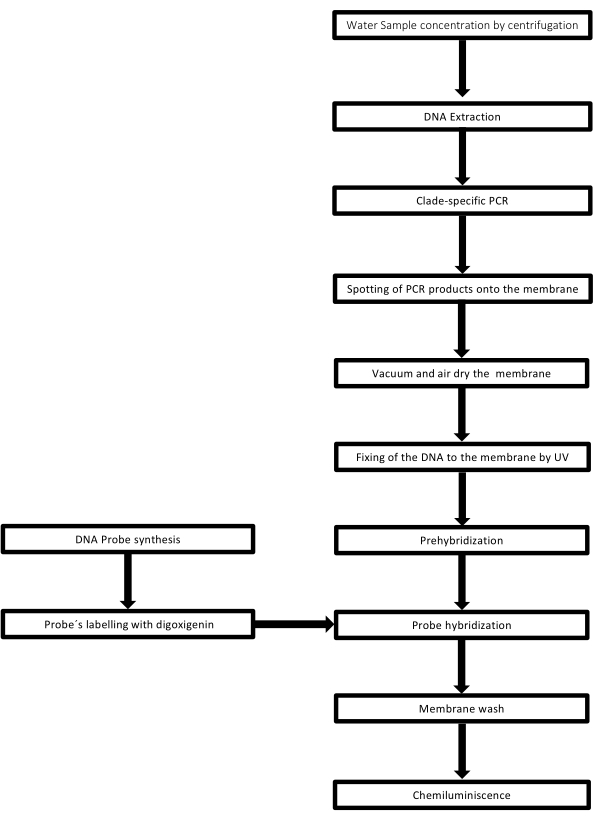
그림 2: PCR-dot-blot assay를 위한 절차 단계. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Protocol
Representative Results
Discussion
도트 블롯 기법의 중요한 단계에는 (1) DNA 고정화, (2) 비상동 DNA로 멤브레인의 자유 결합 부위 차단, (3) 어닐링 조건에서 프로브와 타겟 단편 사이의 상보성, (4) 비혼성화 프로브 제거, (5) 리포터 분자41의 검출이 포함됩니다.
PCR-도트-블롯은 특정 제한사항을 갖는데, 이를테면 이 기술은 혼성화된 단편(37)의 크기에 관한 정보를 제공하지 않고…
Disclosures
The authors have nothing to disclose.
Acknowledgements
우리는 멕시코 국립 자치 대학교 (National Autonomous University of Mexico)의 수의학 및 동물 공학 학부 미생물학 및 면역학과의 렙토스피라 컬렉션에 빚을지고 있습니다. 우리는 참조 Leptospira 균주의 관대 한 기부에 감사드립니다. Leptospira fainei serovar Hurstbridge 균주 BUT6 및 Leptospira biflexa serovar Patoc 균주 Patoc I에서 Dr. Alejandro de la Peña Moctezuma. CIBAC 코디네이터인 Dr. José Antonio Ocampo Cervantes와 직원들의 물류 지원에 감사드립니다. EDT는 Metropolitan Autonomous University-Campus Cuajimalpa의 학부생을 위한 터미널 프로젝트 프로그램에 있었습니다. 우리는 그림 1과 3에서 9까지의 생성을 위한 Biorender.com 소프트웨어를 인정합니다.
Materials
| REAGENTS | |||
| Purelink DNA extraction kit | Invitrogen | K182002 | |
| Gotaq Flexi DNA Polimerase (End-Point PCR Taq polymerase kit) | Promega | M3001 | |
| Whatman filter paper, grade 1, | Merk | WHA1001325 | |
| Nylon Membranes, positively charged Roll 30cm x 3 m | Roche | 11417240001 | |
| Anti-Digoxigenin-AP, Fab fragments Sheep Polyclonal Primary-antibody | Roche | 11093274910 | |
| Medium Base EMJH | Difco | S1368JAA | |
| Leptospira Enrichment EMJH | Difco | BD 279510 | |
| Blocking Reagent | Roche | 11096176001 | |
| CSPD ready to use Disodium 3-(4-methoxyspiro {1,2-dioxetane-3,2′-(5′-chloro) tricyclo [3.3.1.13,7] decan}8-4-yl) phenyl phosphate | Merk | 11755633001 | |
| Deoxyribonucleic acid from herring sperm | Sigma Aldrich | D3159 | |
| Developer Carestream | Carestream Health Inc | GBX5158621 | |
| Digoxigenin-11-ddUTP | Roche | 11363905910 | |
| EDTA, Disodium Salt (Dihydrate) | Promega | H5032 | |
| Ficoll 400 | Sigma Aldrich | F8016 | |
| Fixer Carestream | Carestream Health Inc | GBX 5158605 | |
| Lauryl sulfate Sodium Salt (Sodium dodecyl sulfate; SDS) C12H2504SNa | Sigma Aldrich | L5750 | |
| N- Lauroylsarcosine sodium salt CH3(CH2)10CON(CH3) CH2COONa | Sigma Aldrich | L-9150 | It is an anionic surfactant |
| Polivinylpyrrolidone (PVP-40) | Sigma Aldrich | PVP40 | |
| Polyethylene glycol Sorbitan monolaurate (Tween 20) | Sigma Aldrich | 9005-64-5 | |
| Sodium Chloride (NaCl) | Sigma Aldrich | 7647-14-5 | |
| Sodium dodecyl sulfate (SDS) | Sigma Aldrich | 151-21-3 | |
| Sodium hydroxide (NaOH) | Sigma Aldrich | 1310-73-2 | |
| Sodium phosphate dibasic (NaH2PO4) | Sigma-Aldrich | 7558-79-4 | |
| Terminal transferase, recombinant | Roche | 3289869103 | |
| Tris hydrochloride (Tris HCl) | Sigma-Aldrich | 1185-53-1 | |
| SSPE 20X | Sigma-Aldrich | S2015-1L | It can be Home-made following Supplementary File 6 |
| Primers | Sigma-Aldrich | On demand | Follow table 1 |
| Probes | Sigma-Aldrich | On demand | Follow table 1 |
| Equipment | |||
| Nanodrop™ One Spectrophotometer | Thermo-Scientific | ND-ONE-W | |
| Refrigerated microcentrifuge Sigma 1-14K, suitable for centrifugation of 1.5 ml microcentrifuge tubes at 14,000 rpm | Sigma-Aldrich | 1-14K | |
| Disinfected adjustable pipettes, range 2-20 µl, 20-200 µl | Gilson | SKU:F167360 | |
| Disposable 1.5 ml microcentrifuge tubes (autoclaved) | Axygen | MCT-150-SP | |
| Disposable 600 µl microcentrifuge tubes (autoclaved) | Axygen | 3208 | |
| Disposable Pipette tips 1-10 µl | Axygen | T-300 | |
| Disposable Pipette tips 1-200 µl | Axygen | TR-222-Y | |
| Dot-Blot apparatus Bio-Dot | BIORAD | 1706545 | |
| Portable Hergom Suction | Hergom | 7E-A | |
| Scientific Light Box (Visible-light PH90-115V) | Hoefer | PH90-115V | |
| UV Crosslinker | Hoefer | UVC-500 | |
| Thermo Hybaid PCR Express Thermocycler | Hybaid | HBPX110 | |
| Radiographic cassette with IP Plate14 X 17 | Fuji |
References
- Bierque, E., Thibeaux, R., Girault, D., Soupé-Gilbert, M. E., Goarant, C. A systematic review of Leptospira in water and soil environments. PLOS One. 15 (1), e0227055 (2020).
- Haake, D. A., Levett, P. N. Leptospirosis in humans. Current Topics in Microbiology and Immunology. 387, 65-97 (2015).
- Tripathy, D. N., Hanson, L. E. Leptospires from water sources at Dixon Springs Agricultural Center. Journal of Wildlife Diseases. 9 (3), 209-212 (1973).
- Smith, D. J., Self, H. R. Observations on the survival of Leptospira australis A in soil and water. The Journal of Hygiene. 53 (4), 436-444 (1955).
- Karpagam, K. B., Ganesh, B. Leptospirosis: a neglected tropical zoonotic infection of public health importance-an updated review. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 39 (5), 835-846 (2020).
- Casanovas-Massana, A., et al. Spatial and temporal dynamics of pathogenic Leptospira in surface waters from the urban slum environment. Water Research. 130, 176-184 (2018).
- Costa, F., et al. Global morbidity and mortality of Leptospirosis: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003898 (2015).
- Mwachui, M. A., Crump, L., Hartskeerl, R., Zinsstag, J., Hattendorf, J. Environmental and behavioural determinants of Leptospirosis transmission: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003843 (2015).
- Andre-Fontaine, G., Aviat, F., Thorin, C. Waterborne Leptospirosis: Survival and preservation of the virulence of pathogenic Leptospira spp. in fresh water. Current Microbiology. 71 (1), 136-142 (2015).
- Trueba, G., Zapata, S., Madrid, K., Cullen, P., Haake, D. Cell aggregation: A mechanism of pathogenic Leptospira to survive in freshwater. International Microbiology: the Official Journal of the Spanish Society for Microbiology. 7 (1), 35-40 (2004).
- Smith, C. E., Turner, L. H. The effect of pH on the survival of leptospires in water. Bulletin of the World Health Organization. 24 (1), 35-43 (1961).
- Barragan, V. A., et al. Interactions of Leptospira with environmental bacteria from surface water. Current Microbiology. 62 (6), 1802-1806 (2011).
- Abdoelrachman, R. Comparative investigations into the influence of the presence of bacteria on the life of pathogenic and apathogenic leptospirae. Antonie van Leeuwenhoek. 13 (1), 21-32 (1947).
- Singh, R., et al. Microbial diversity of biofilms in dental unit water systems. Applied and Environmental Microbiology. 69 (6), 3412-3420 (2003).
- Kumar, K. V., Lall, C., Raj, R. V., Vedhagiri, K., Vijayachari, P. Coexistence and survival of pathogenic leptospires by formation of biofilm with Azospirillum. FEMS Microbiology Ecology. 91 (6), 051 (2015).
- Yanagihara, Y., et al. Leptospira Is an environmental bacterium that grows in waterlogged soil. Microbiology Spectrum. 10 (2), 0215721 (2022).
- Gillespie, R. W., Ryno, J. Epidemiology of leptospirosis. American Journal of Public Health and Nation’s Health. 53 (6), 950-955 (1963).
- Bierque, E., et al. Leptospira interrogans retains direct virulence after long starvation in water. Current Microbiology. 77 (10), 3035-3043 (2020).
- Zhang, Y., Dai, B. Marking and detection of DNA of leptospires in the dot-blot and situ hybridization with digoxigenin-labeled probes. Journal of West China University of Medical Sciences. 23 (4), 353-435 (1992).
- Mérien, F., Amouriaux, P., Perolat, P., Baranton, G., Saint Girons, I. Polymerase chain reaction for detection of Leptospira spp. in clinical samples. Journal of Clinical Microbiology. 30 (9), 2219-2224 (1992).
- Veerapandian, R., et al. Silver enhanced nano-gold dot-blot immunoassay for leptospirosis. Journal of Microbiological Methods. 156, 20-22 (2019).
- Junpen, S., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Ishmael, F. T., Stellato, C. Principles and applications of polymerase chain reaction: basic science for the practicing physician. Annals of Allergy, Asthma & Immunology: Official Publication of the American College of Allergy, Asthma, & Immunology. 101 (4), 437-443 (2008).
- Boerner, B., Weigelt, W., Buhk, H. J., Castrucci, G., Ludwig, H. A sensitive and specific PCR/Southern blot assay for detection of bovine herpesvirus 4 in calves infected experimentally. Journal of Virological Methods. 83 (1-2), 169-180 (1999).
- Curry, E., Pratt, S. L., Kelley, D. E., Lapin, D. R., Gibbons, J. R. Use of a Combined duplex PCR/Dot-blot assay for more sensitive genetic characterization. Biochemistry Insights. 1, 35-39 (2008).
- Pilatti, M. M., Ferreira, S. d. e. A., de Melo, M. N., de Andrade, A. S. Comparison of PCR methods for diagnosis of canine visceral leishmaniasis in conjunctival swab samples. Research in Veterinary Science. 87 (2), 255-257 (2009).
- Conrads, G., et al. PCR reaction and dot-blot hybridization to monitor the distribution of oral pathogens within plaque samples of periodontally healthy individuals. Journal of Periodontology. 67 (10), 994-1003 (1996).
- Langa, S., et al. Differentiation of Enterococcus faecium from Lactobacillus delbrueckii subsp. bulgaricus and Streptococcus thermophilus strains by PCR and dot-blot hybridisation. International Journal of Food Microbiology. 88 (2-3), 197-200 (2003).
- Francesca, C., Lucilla, I., Marco, F., Giuseppe, C., Marisa, M. Identification of the unculturable bacteria Candidatus arthromitus in the intestinal content of trouts using dot-blot and Southern blot techniques. Veterinary Microbiology. 156 (3-4), 389-394 (2012).
- Arent, Z., Pardyak, L., Dubniewicz, K., Plachno, B. J., Kotula-Balak, M. Leptospira taxonomy: then and now. Medycyna Weterynaryjna. 78 (10), 489-496 (2022).
- Thibeaux, R., et al. Biodiversity of environmental Leptospira: Improving identification and revisiting the diagnosis. Frontiers in Microbiology. 9, 816 (2018).
- Carrillo-Casas, E. M., Hernández-Castro, R., Suárez-Güemes, F., de la Peña-Moctezuma, A. Selection of the internal control gene for real-time quantitative RT-PCR assays in temperature treated Leptospira. Current Microbiology. 56 (6), 539-546 (2008).
- Azali, M. A., Yean Yean, C., Harun, A., Aminuddin Baki A, N. N., Ismail, N. Molecular characterization of Leptospira spp. in environmental samples from North-Eastern Malaysia revealed a pathogenic strain, Leptospira alstonii. Journal of Tropical Medicine. 2016, 2060241 (2016).
- Ahmed, N., et al. Multilocus sequence typing method for identification and genotypic classification of pathogenic Leptospira species. Annals of Clinical Microbiology and Antimicrobials. 5, 28 (2006).
- Bourhy, P., Collet, L., Brisse, S., Picardeau, M. Leptospira mayottensis sp. nov., a pathogenic species of the genus Leptospira isolated from humans. International Journal of Systematic and Evolutionary Microbiology. 64, 4061-4067 (2014).
- Weiss, S., et al. An extended Multilocus Sequence Typing (MLST) scheme for rapid direct typing of Leptospira from clinical samples. PLOS Neglected Tropical Diseases. 10 (9), e0004996 (2016).
- Branger, C., et al. Polymerase chain reaction assay specific for pathogenic Leptospira based on the gene hap1 encoding the hemolysis-associated protein-1. FEMS Microbiology Letters. 243 (2), 437-445 (2005).
- Ren, S. X., et al. Unique physiological and pathogenic features of Leptospira interrogans revealed by whole-genome sequencing. Nature. 422 (6934), 888-893 (2003).
- Picardeau, M., et al. Genome sequence of the saprophyte Leptospira biflexa provides insights into the evolution of Leptospira and the pathogenesis of leptospirosis. PLOS One. 3 (2), e1607 (2008).
- Kafatos, F. C., Jones, C. W., Efstratiadis, A. Determination of nucleic acid sequence homologies and relative concentrations by a dot hybridization procedure. Nucleic Acids Research. 7 (6), 1541-1552 (1979).
- Bhat, A. I., Rao, G. P. Dot-blot hybridization technique. Characterization of Plant Viruses. , 303-321 (2020).
- Yadav, J. P., Batra, K., Singh, Y., Singh, M. Comparative evaluation of indirect-ELISA and Dot-blot assay for serodetection of Mycoplasma gallisepticum and Mycoplasma synoviae antibodies in poultry. Journal of Microbiological Methods. 189, 106317 (2021).
- Malinen, E., Kassinen, A., Rinttilä, T., Palva, A. Comparison of real-time PCR with SYBR Green I or 5′-nuclease assays and dot-blot hybridization with rDNA-targeted oligonucleotide probes in quantification of selected faecal bacteria. Microbiology. 149, 269-277 (2003).
- Wyss, C., et al. Treponema lecithinolyticum sp. nov., a small saccharolytic spirochaete with phospholipase A and C activities associated with periodontal diseases. International Journal of Systematic Bacteriology. 49, 1329-1339 (1999).
- Shah, J. S., I, D. C., Ward, S., Harris, N. S., Ramasamy, R. Development of a sensitive PCR-dot-blot assay to supplement serological tests for diagnosing Lyme disease. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 37 (4), 701-709 (2018).
- Niu, C., Wang, S., Lu, C. Development and evaluation of a dot-blot assay for rapid determination of invasion-associated gene ibeA directly in fresh bacteria cultures of E. coli. Folia microbiologica. 57 (6), 557-561 (2012).
- Wetherall, B. L., McDonald, P. J., Johnson, A. M. Detection of Campylobacter pylori DNA by hybridization with non-radioactive probes in comparison with a 32P-labeled probe. Journal of Medical Microbiology. 26 (4), 257-263 (1988).
- Kolk, A. H., et al. Detection of Mycobacterium tuberculosis in clinical samples by using polymerase chain reaction and a nonradioactive detection system. Journal of Clinical Microbiology. 30 (10), 2567-2575 (1992).
- Scherer, L. C., et al. PCR colorimetric dot-blot assay and clinical pretest probability for diagnosis of Pulmonary Tuberculosis in smear-negative patients. BMC Public Health. 7, 356 (2007).
- Armbruster, D. A., Pry, T. Limit of blank, limit of detection and limit of quantitation. The Clinical Biochemist Reviews. 29, S49-S52 (2008).
- Zhang, Y., Dai, B. Detection of Leptospira by dot-blot hybridization with photobiotin- and 32P-labeled DNA. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 130-132 (1992).
- Terpstra, W. J., Schoone, G. J., ter Schegget, J. Detection of leptospiral DNA by nucleic acid hybridization with 32P- and biotin-labeled probes. Journal of Medical Microbiology. 22 (1), 23-28 (1986).
- Shukla, J., Tuteja, U., Batra, H. V. DNA probes for identification of leptospires and disease diagnosis. The Southeast Asian Journal of Tropical Medicine and Public Health. 35 (2), 346-352 (2004).
- Jiang, N., Jin, B., Dai, B., Zhang, Y. Identification of pathogenic and nonpathogenic leptospires by recombinant probes. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 26 (1), 1-5 (1995).
- Fach, P., Trap, D., Guillou, J. P. Biotinylated probes to detect Leptospira interrogans on dot-blot hybridization or by in situ hybridization. Letters in Applied Microbiology. 12 (5), 171-176 (1991).
- Huang, N., Dai, B. Assay of genomic DNA homology among strains of different virulent leptospira by DNA hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 122-125 (1992).
- Dong, X., Dai, B., Chai, J. Homology study of leptospires by molecular hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (1), 1-4 (1992).
- Komminoth, P. Digoxigenin as an alternative probe labeling for in situ hybridization. Diagnostic Molecular Pathology: The American Journal of Surgical Pathology, part B. 1 (2), 142-150 (1992).
- Saengjaruk, P., et al. Diagnosis of human leptospirosis by monoclonal antibody-based antigen detection in urine. Journal of Clinical Microbiology. 40 (2), 480-489 (2002).
- Okuda, M., et al. Enzyme-linked immunosorbent assay for the detection of canine Leptospira antibodies using recombinant OmpL1 protein. The Journal of Veterinary Medical Science. 67 (3), 249-254 (2005).
- Suwimonteerabutr, J., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Kanagavel, M., et al. Peptide-specific monoclonal antibodies of Leptospiral LigA for acute diagnosis of leptospirosis. Scientific reports. 7 (1), 3250 (2017).
- Levett, P. N. Leptospirosis. Clinical Microbiology Reviews. 14 (2), 296-326 (2001).
- Monahan, A. M., Callanan, J. J., Nally, J. E. Proteomic analysis of Leptospira interrogans shed in urine of chronically infected hosts. Infection and Immunity. 76 (11), 4952-4958 (2008).
- Rojas, P., et al. Detection and quantification of leptospires in urine of dogs: a maintenance host for the zoonotic disease leptospirosis. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 29 (10), 1305-1309 (2010).

