Su Örneklerinde Leptospira Tespiti için Polimeraz Zincir Reaksiyonu ve Nokta-Blot Hibridizasyonu,
Summary
Bu çalışmada, su örneklerinde üç ana daldan Leptospira’yı tespit etmek için bir nokta-leke uygulaması tasarlanmıştır. Bu yöntem, bir anti-digoksigenin antikoru tarafından kolayca tespit edilen, digoksigenin etiketli bir prob tarafından spesifik olarak hedeflenen minimum DNA miktarlarının tanımlanmasına izin verir. Bu yaklaşım, tarama amaçları için değerli ve tatmin edici bir araçtır.
Abstract
Nokta lekesi, taşıyıcı DNA’nın varlığında prob hibridizasyonuyla özel olarak hedeflenen minimum miktarda DNA’nın tanımlanmasını sağlayan basit, hızlı, hassas ve çok yönlü bir tekniktir. Bilinen bir miktarda DNA’nın, nokta-leke aparatı kullanılarak ve elektroforetik ayırma olmaksızın naylon zar gibi inert bir katı destek üzerine aktarılmasına dayanır. Naylon membranlar, yüksek nükleik asit bağlama kapasitesi (400 μg/cm2), yüksek mukavemet avantajına sahiptir ve pozitif veya nötr yüklüdür. Kullanılan prob, digoksijenenin (DIG) ile uzun süre etiketlenmiş 18 ila 20 bazdan oluşan oldukça spesifik bir ssDNA fragmanıdır. Prob, Leptospira DNA’sı ile konjuge olacaktır. Prob, hedef DNA ile hibritlendikten sonra, bir anti-digoksigenin antikoru tarafından tespit edilir ve bir X-ışını filminde ortaya çıkan emisyonları sayesinde kolay tespit edilmesini sağlar. Emisyonlu noktalar, ilgilenilen DNA parçalarına karşılık gelecektir. Bu yöntem, çok uzun bir yarı ömre sahip olabilen probun izotopik olmayan etiketlemesini kullanır. Bu standart immüno-etiketin dezavantajı, izotopik problardan daha düşük bir hassasiyettir. Bununla birlikte, polimeraz zincir reaksiyonu (PCR) ve nokta-blot deneylerinin birleştirilmesiyle hafifletilir. Bu yaklaşım, hedef dizinin zenginleştirilmesini ve algılanmasını sağlar. Ek olarak, iyi bilinen bir standardın seri seyreltilmesiyle karşılaştırıldığında nicel bir uygulama olarak kullanılabilir. Su örneklerinde üç ana daldan Leptospira’yı tespit etmek için bir nokta-leke uygulaması burada sunulmaktadır. Bu metodoloji, Leptospiral DNA’nın varlığına dair kanıt sağlamak için santrifüjleme ile konsantre edildikten sonra büyük miktarlarda suya uygulanabilir. Bu, genel tarama amaçları için değerli ve tatmin edici bir araçtır ve suda bulunabilecek diğer kültürlenemeyen bakteriler için kullanılabilir, bu da ekosistemin anlaşılmasını geliştirir.
Introduction
İnsanlarda leptospirosis esas olarak çevresel kaynaklardan kaynaklanır 1,2. Göllerde, nehirlerde ve akarsularda Leptospira’nın varlığı, yaban hayatı ve sonunda bu su kütleleriyle temas edebilecek evcil ve üretim hayvanları arasında leptospirosis bulaşmasının bir göstergesidir 1,3,4. Ayrıca, Leptospira, kanalizasyon, durgun ve musluk suyu dahil olmak üzere doğal olmayan kaynaklarda tespit edilmiştir 5,6.
Leptospira, dünya çapında dağılmış bir bakteridir 7,8 ve çevrenin korunması ve iletilmesindeki rolü iyi bilinmektedir. Leptospira, değişken pH ve mineraller9 altında içme suyunda ve doğal su kütlelerinde1 hayatta kalabilir. Ayrıca damıtılmış suda10 uzun süre hayatta kalabilir ve sabit pH (7.8) altında 152 güne kadar hayatta kalabilir11. Ayrıca, Leptospira zorlu koşullarda hayatta kalmak için bakteri konsorsiyumlarında etkileşime girebilir12,13. Azospirillum ve Sphingomonas ile tatlı sudaki biyofilmlerin bir parçası olabilir ve hatta 49 ° C’yi aşan sıcaklıklarda büyüme ve dayanma yeteneğine sahiptir14,15. Ayrıca su ile tıkanmış toprakta çoğalabilir ve 379 güne kadarcanlı kalabilir 16 ve hastalığa neden olma yeteneğini bir yıl kadar uzun süre koruyabilir17,18. Bununla birlikte, su kütleleri içindeki ekoloji ve bunların içinde nasıl dağıldığı hakkında çok az şey bilinmektedir.
Keşfinden bu yana, Leptospira cinsinin çalışması serolojik testlere dayanıyordu. İçinde bulunduğumuz yüzyıla kadar, bu spiroketin çalışmasında moleküler teknikler daha yaygın hale gelmedi. Nokta-leke, (1) 16S rRNA’ya dayalı bir izotopik prob ve basitler arası dizi tekrarına (ISSR) 19,20, (2) idrara uygulanan insan leptospirozu için nanogold bazlı bir immünolojik test olarak21 veya (3) sığır idrar örnekleri için antikor bazlı bir test olarak22. Teknik, başlangıçta izotopik problara dayandığı için kullanım dışı kaldı. Bununla birlikte, PCR ile birleştiğinde gelişmiş sonuçlar veren iyi bilinen bir tekniktir ve izotopik olmayan probların kullanılması nedeniyle güvenli kabul edilir. PCR, bir numunede eser miktarlarda bulunabilen spesifik bir DNA fragmanını amplifiye ederek Leptospira DNA’sının zenginleştirilmesinde çok önemli bir rol oynar. Her PCR döngüsü sırasında, hedeflenen DNA fragmanının miktarı reaksiyonda iki katına çıkar. Reaksiyonun sonunda, amplikon bir milyondan fazla23 faktörü ile çarpılmıştır. PCR ile amplifiye edilen, genellikle agaroz elektroforezinde görülmeyen ürün, nokta lekesi 24,25,26’da DIG etiketli bir prob ile spesifik hibridizasyon yoluyla görünür hale gelir.
Nokta-leke tekniği basit, sağlam ve çok sayıda numune için uygundur, bu da onu sınırlı kaynaklara sahip laboratuvarlar için erişilebilir kılar. (1) oral bakteriler27, (2) gıda ve dışkı28 gibi diğer numune türleri ve (3) kültürlenemeyen bakterilerintanımlanması 29 dahil olmak üzere, genellikle diğer moleküler tekniklerle uyumlu olarak çeşitli bakteri çalışmalarında kullanılmıştır. Dot-blot tekniğinin sunduğu avantajlar arasında şunlar yer alır: (1) Membran, 200 μg/cm2’den fazla nükleik asit ve 400 μg/cm2’ye kadar bağlayabilen yüksek bir bağlama kapasitesine sahiptir; (2) Nokta lekesi sonuçları, özel ekipman gerektirmeden görsel olarak yorumlanabilir ve (3) oda sıcaklığında (RT) yıllarca rahatlıkla saklanabilir.
Leptospira cinsi patojenik, orta ve saprofit dallar olarak sınıflandırılmıştır30,31. Bu kladlar arasındaki ayrım, lipL41, lipL32 ve 16 S rRNA gibi spesifikgenlere dayalı olarak elde edilebilir. LipL32 patojenik dallarda bulunur ve çeşitli serolojik ve moleküler araçlarda yüksek hassasiyet gösterirken, saprofit türlerindeyoktur 21. Temizlik geni lipL41, kararlı ekspresyonu ile bilinir ve moleküler tekniklerde32 kullanılırken, 16S rRNA geni bunların sınıflandırılması için kullanılır.
Bu metodoloji, santrifüjleme ile konsantre edildikten sonra büyük hacimlerde suya uygulanabilir. Leptospiral DNA’nın varlığını ve ait olduğu kolu tespit etmek için bir su kütlesi içindeki çeşitli noktaların ve derinliklerin değerlendirilmesine izin verir. Bu araç hem ekolojik hem de genel tarama amaçları için değerlidir ve suda bulunabilecek diğer kültürlenemeyen bakterileri tespit etmek için de kullanılabilir.
Ek olarak, PCR ve nokta lekesi tahlilleri, karmaşık veya pahalı ekipmana sahip olmayanlar da dahil olmak üzere çok çeşitli laboratuvarlar için teknik ve ekonomik olarak uygun maliyetlidir. Bu çalışma, doğal su kütlelerinden toplanan su örneklerinde üç Leptospira dalının tanımlanması için digoksigenin bazlı nokta-lekeyi uygulamayı amaçlamaktadır.
Bakteri suşları
On iki Leptospira serovarı (Autumnalis, Bataviae, Bratislava, Canicola, Celledoni, Grippothyphosa, Hardjoprajitno, Icterohaemorrhagiae, Pomona, Pyrogenes, Tasma ve Wolffi) bu çalışmaya dahil edildi. Bu serovarlar, Meksika Ulusal Özerk Üniversitesi Veterinerlik ve Zooteknik Fakültesi, Mikrobiyoloji ve İmmünoloji Bölümü’ndeki koleksiyonun bir parçasıdır ve şu anda mikroaglütinasyon testinde (MAT) kullanılmaktadır.
Tüm Leptospira serovarları EMJH’de kültürlendi ve DNA’ları ticari bir DNA ekstraksiyon kiti kullanılarak ekstrakte edildi (bkz. On iki serovarın bir genomik DNA karışımı, Leptospira patojenik sınıfı için pozitif bir kontrol olarak kullanıldı. Leptospira ara sınıfının pozitif bir kontrolü olarak, Leptospira fainei serovar Hurstbridge suşu BUT6’dan genomik DNA dahil edildi ve Leptospira saprofit sınıfı için pozitif bir kontrol olarak, Leptospira biflexa serovar Patoc suşu Patoc I’in genomik DNA’sı da dahil edildi.
Negatif kontroller boş bir plazmit, akraba olmayan bakterilerden (Ureaplasma urealyticum, Staphylococcus aureus, Brucella abortus, Salmonella typhimurium, Shigella boydii, Klebsiella pneumoniae, Acinetobacter baumannii ve Escherichia coli) DNA’dan ve şablon olmayan kontrol olarak görev yapan PCR dereceli sudan oluşuyordu.
Su örnekleri
Cuemanco Biyolojik ve Su Ürünleri Araştırma Merkezi’nden (CIBAC) (19 ° 16′ 54″ N 99 ° 6 ’11” W) tabakalı-gelişigüzel örnekleme yöntemi kullanılarak on iki deneme yakalama örneği toplandı. Bu örnekler yüzeysel, 10 ve 30 cm olmak üzere üç derinlikte elde edilmiştir (Şekil 1A, B). Su toplama prosedürleri, nesli tükenmekte olan veya korunan herhangi bir türü etkilemedi. Her örnek steril 15 mL’lik bir mikrosantrifüj tüpünde toplandı. Numuneyi toplamak için, her tüp nazikçe suya batırıldı, seçilen derinlikte dolduruldu ve ardından kapatıldı. Numuneler 22 °C’de tutuldu ve işlenmek üzere derhal laboratuvara taşındı.
Her numune, oda sıcaklığında 20 dakika boyunca 8000 x g’da steril 1.5 mL mikrosantrifüj tüplerinde santrifüjleme yoluyla konsantre edildi. Bu adım, tüm numuneler daha sonra DNA ekstraksiyonu için kullanılan tek bir tüpe konsantre edilene kadar tekrarlandı (Şekil 1C).
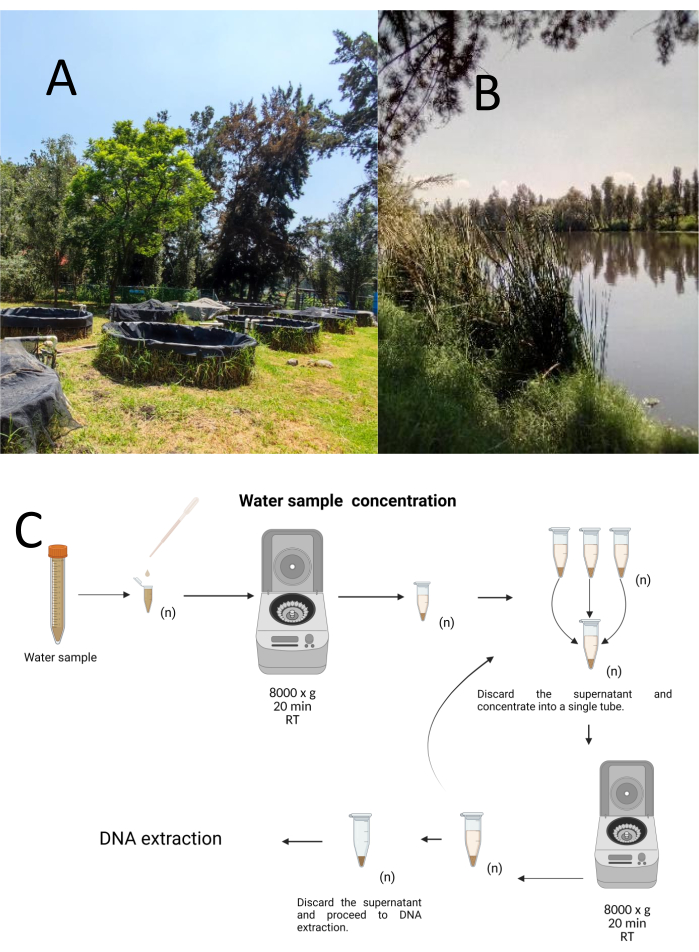
Şekil 1: Santrifüjleme ile su örneklerinin konsantrasyonu. (A) Su örnekleme havuzları ve (B) Doğal akarsular. (C) Santrifüjleme bazlı su numunesinin gerektiği kadar tekrarlanan adımlarla işlenmesi (n). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
DNA ekstraksiyonu
Toplam DNA, üreticinin talimatlarına göre ticari bir Genomik DNA kiti kullanılarak izole edildi (bkz. Malzeme Tablosu). DNA ekstraksiyonları 20 μL elüsyon tamponunda ayrıştırıldı ve DNA konsantrasyonu 260-280 nm’de bir UV Spektrofotometre ile belirlendi ve kullanıma kadar 4 ° C’de saklandı.
PCR amplifikasyonu
PCR hedefleri, Leptospira cinsinden DNA’yı tanımlayan ve patojenik, saprofit ve orta olmak üzere üç dal arasında ayrım yapılmasına izin veren 16S rRNA, lipL41 ve lipL32 genleriydi. Hem primerler hem de prob tasarımları, Ahmed ve ark., Azali ve ark., Bourhy ve ark., Weiss ve ark. ve Branger ve ark.33,34,35,36,37 tarafından yapılan önceki çalışmalara dayanıyordu. Her bir probun, primerin ve amplifiye edilmiş parçanın dizisi Tablo 1’de açıklanmıştır ve bunların referans dizileriyle hizalanması Ek Dosya 1, Ek Dosya 2, Ek Dosya 3, Ek Dosya 4 ve Ek Dosya 5’te verilmiştir. PCR reaktifleri ve termosiklasyon koşulları protokol bölümünde açıklanmıştır.
Amplifikasyon ürünleri, Ek Şekil 1’de gösterildiği gibi, etidyum-bromür tespiti ile 45 dakika boyunca 60 V’ta TAE’de (40 mM Tris bazı, 20 mM Asetik asit ve 1 mM EDTA; pH 8.3) %1’lik bir agaroz jel üzerinde elektroforetik ayırma ile görselleştirildi. Her bir serovardan elde edilen genomik DNA, her PCR reaksiyonunda 6 x 106 ila 1 x 104 genomik eşdeğer kopya (GEq) arasında değişen konsantrasyonlarda kullanıldı, L. interrogans’ın genom boyutuna dayalı olarak (4, 691, 184 bp)38 patojenik Leptospira için, saprofit Leptospira için L. biflexa’nın (3, 956, 088 bp)39 genom boyutu, ve L. fainei serovar Hurstbridge suşu BUT6’nın (4, 267, 324 bp) genom boyutu, erişim numarası AKWZ00000000.2.
Probların duyarlılığı, her deneyde her bir patojenik serovar, L. biflexa serovar Patoc suşu Patoc I ve L. fainei serovar Hurstbridge suşu BUT6’dan alınan DNA ile değerlendirildi. PCR ve nokta-blot hibridizasyon testinin özgüllüğünü değerlendirmek için, ilgili olmayan bakterilerden alınan DNA dahil edildi.
Tablo 1: Leptospira’nın patojenik, saprofit ve ara sınıflarını tanımlamak için ürünleri çoğaltmak için PCR primerleri ve probları. Bu Tabloyu indirmek için lütfen buraya tıklayın.
Nokta-blot hibridizasyon deneyi
Bu tekniğe nokta-leke adı verilir çünkü DNA örneğinin yerleştirildiği delikler nokta şeklindedir ve vakumla emme ile yerlerine sabitlenmek üzere emildiklerinde bu şekli alırlar. Bu teknik Kafatos ve ark.40 tarafından geliştirilmiştir. Teknik, her PCR pozitif numunede Leptospira’nın yarı miktar tayinine izin verir. Protokol, oda sıcaklığında NaOH 0.4 M ile bir denatürasyondan, 6 x 106 ila 1 x 104 leptospire karşılık gelen 30 ng ila 0.05 ng Leptospira DNA’lı numunelerden oluşur, 96 oyuklu bir nokta lekesi aparatı ile bir naylon membran üzerine lekelenir. İmmobilizasyondan sonra DNA, 120 mJ UV ışığına maruz bırakılarak zara bağlanır. Her DNA probu, 3′ ucunda bir terminal transferaz kataliz adımı ile digoksigenin-11 dUTP ile konjuge edilir (Digoksigenin, raportör olarak kullanılan Digitalis purpurea’dan elde edilen bir bitki steroididir41). Etiketli DNA probunun (50 pmol) spesifik sıcaklıkta hedef DNA üzerine sıkı bir şekilde hibridizasyonunu takiben, DNA hibritleri, substratı CSPD ile kovalent olarak konjuge edilmiş anti-digoksigenin alkalin fosfataz antikoru ile kemilüminesans reaksiyonu ile görselleştirilir. Lüminesans, bir X-ışını filmine maruz bırakılarak yakalanır (Şekil 2).
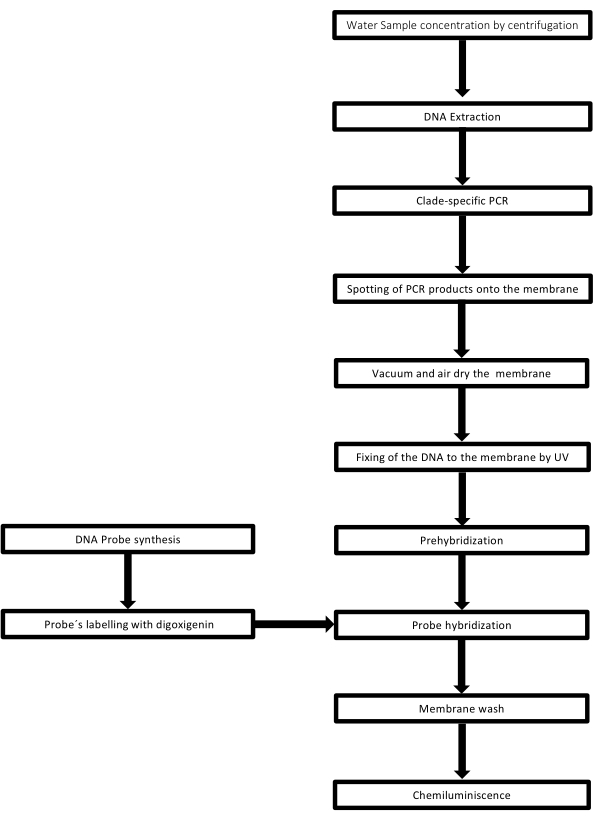
Şekil 2: PCR-nokta-leke testi için prosedür adımları. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Protocol
Representative Results
Discussion
Nokta-blot tekniğinin kritik adımları şunları içerir: (1) DNA immobilizasyonu, (2) membran üzerindeki serbest bağlanma bölgelerinin homolog olmayan DNA ile bloke edilmesi, (3) tavlama koşulları altında prob ve hedef fragman arasındaki tamamlayıcılık, (4) hibritlenmemiş probun çıkarılması ve (5) raportör molekülün41 tespiti.
PCR-Dot-blot’un, tekniğin hibritleştirilmiş parçanın37 boyutu hakkında bilgi sağlamaması, ik…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Meksika Ulusal Özerk Üniversitesi, Veterinerlik ve Zootekni Fakültesi, Mikrobiyoloji ve İmmünoloji Bölümü’nün Leptospira koleksiyonuna borçluyuz. Referans Leptospira suşlarının cömert bağışı için minnettarız; Leptospira fainei serovar Hurstbridge suşu BUT6 ve Leptospira biflexa serovar Patoc suşu Patoc I’den Dr. Alejandro de la Peña Moctezuma’ya. CIBAC Koordinatörü Dr. José Antonio Ocampo Cervantes’e ve personele lojistik destekleri için teşekkür ederiz. EDT, Metropolitan Özerk Üniversitesi-Kampüsü Cuajimalpa’nın lisans öğrencileri için Terminal Projesi programı kapsamındaydı. Şekil 1 ve 3’ten 9’a kadar olan şekillerin oluşturulması için Biorender.com yazılımı kabul ediyoruz.
Materials
| REAGENTS | |||
| Purelink DNA extraction kit | Invitrogen | K182002 | |
| Gotaq Flexi DNA Polimerase (End-Point PCR Taq polymerase kit) | Promega | M3001 | |
| Whatman filter paper, grade 1, | Merk | WHA1001325 | |
| Nylon Membranes, positively charged Roll 30cm x 3 m | Roche | 11417240001 | |
| Anti-Digoxigenin-AP, Fab fragments Sheep Polyclonal Primary-antibody | Roche | 11093274910 | |
| Medium Base EMJH | Difco | S1368JAA | |
| Leptospira Enrichment EMJH | Difco | BD 279510 | |
| Blocking Reagent | Roche | 11096176001 | |
| CSPD ready to use Disodium 3-(4-methoxyspiro {1,2-dioxetane-3,2′-(5′-chloro) tricyclo [3.3.1.13,7] decan}8-4-yl) phenyl phosphate | Merk | 11755633001 | |
| Deoxyribonucleic acid from herring sperm | Sigma Aldrich | D3159 | |
| Developer Carestream | Carestream Health Inc | GBX5158621 | |
| Digoxigenin-11-ddUTP | Roche | 11363905910 | |
| EDTA, Disodium Salt (Dihydrate) | Promega | H5032 | |
| Ficoll 400 | Sigma Aldrich | F8016 | |
| Fixer Carestream | Carestream Health Inc | GBX 5158605 | |
| Lauryl sulfate Sodium Salt (Sodium dodecyl sulfate; SDS) C12H2504SNa | Sigma Aldrich | L5750 | |
| N- Lauroylsarcosine sodium salt CH3(CH2)10CON(CH3) CH2COONa | Sigma Aldrich | L-9150 | It is an anionic surfactant |
| Polivinylpyrrolidone (PVP-40) | Sigma Aldrich | PVP40 | |
| Polyethylene glycol Sorbitan monolaurate (Tween 20) | Sigma Aldrich | 9005-64-5 | |
| Sodium Chloride (NaCl) | Sigma Aldrich | 7647-14-5 | |
| Sodium dodecyl sulfate (SDS) | Sigma Aldrich | 151-21-3 | |
| Sodium hydroxide (NaOH) | Sigma Aldrich | 1310-73-2 | |
| Sodium phosphate dibasic (NaH2PO4) | Sigma-Aldrich | 7558-79-4 | |
| Terminal transferase, recombinant | Roche | 3289869103 | |
| Tris hydrochloride (Tris HCl) | Sigma-Aldrich | 1185-53-1 | |
| SSPE 20X | Sigma-Aldrich | S2015-1L | It can be Home-made following Supplementary File 6 |
| Primers | Sigma-Aldrich | On demand | Follow table 1 |
| Probes | Sigma-Aldrich | On demand | Follow table 1 |
| Equipment | |||
| Nanodrop™ One Spectrophotometer | Thermo-Scientific | ND-ONE-W | |
| Refrigerated microcentrifuge Sigma 1-14K, suitable for centrifugation of 1.5 ml microcentrifuge tubes at 14,000 rpm | Sigma-Aldrich | 1-14K | |
| Disinfected adjustable pipettes, range 2-20 µl, 20-200 µl | Gilson | SKU:F167360 | |
| Disposable 1.5 ml microcentrifuge tubes (autoclaved) | Axygen | MCT-150-SP | |
| Disposable 600 µl microcentrifuge tubes (autoclaved) | Axygen | 3208 | |
| Disposable Pipette tips 1-10 µl | Axygen | T-300 | |
| Disposable Pipette tips 1-200 µl | Axygen | TR-222-Y | |
| Dot-Blot apparatus Bio-Dot | BIORAD | 1706545 | |
| Portable Hergom Suction | Hergom | 7E-A | |
| Scientific Light Box (Visible-light PH90-115V) | Hoefer | PH90-115V | |
| UV Crosslinker | Hoefer | UVC-500 | |
| Thermo Hybaid PCR Express Thermocycler | Hybaid | HBPX110 | |
| Radiographic cassette with IP Plate14 X 17 | Fuji |
References
- Bierque, E., Thibeaux, R., Girault, D., Soupé-Gilbert, M. E., Goarant, C. A systematic review of Leptospira in water and soil environments. PLOS One. 15 (1), e0227055 (2020).
- Haake, D. A., Levett, P. N. Leptospirosis in humans. Current Topics in Microbiology and Immunology. 387, 65-97 (2015).
- Tripathy, D. N., Hanson, L. E. Leptospires from water sources at Dixon Springs Agricultural Center. Journal of Wildlife Diseases. 9 (3), 209-212 (1973).
- Smith, D. J., Self, H. R. Observations on the survival of Leptospira australis A in soil and water. The Journal of Hygiene. 53 (4), 436-444 (1955).
- Karpagam, K. B., Ganesh, B. Leptospirosis: a neglected tropical zoonotic infection of public health importance-an updated review. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 39 (5), 835-846 (2020).
- Casanovas-Massana, A., et al. Spatial and temporal dynamics of pathogenic Leptospira in surface waters from the urban slum environment. Water Research. 130, 176-184 (2018).
- Costa, F., et al. Global morbidity and mortality of Leptospirosis: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003898 (2015).
- Mwachui, M. A., Crump, L., Hartskeerl, R., Zinsstag, J., Hattendorf, J. Environmental and behavioural determinants of Leptospirosis transmission: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003843 (2015).
- Andre-Fontaine, G., Aviat, F., Thorin, C. Waterborne Leptospirosis: Survival and preservation of the virulence of pathogenic Leptospira spp. in fresh water. Current Microbiology. 71 (1), 136-142 (2015).
- Trueba, G., Zapata, S., Madrid, K., Cullen, P., Haake, D. Cell aggregation: A mechanism of pathogenic Leptospira to survive in freshwater. International Microbiology: the Official Journal of the Spanish Society for Microbiology. 7 (1), 35-40 (2004).
- Smith, C. E., Turner, L. H. The effect of pH on the survival of leptospires in water. Bulletin of the World Health Organization. 24 (1), 35-43 (1961).
- Barragan, V. A., et al. Interactions of Leptospira with environmental bacteria from surface water. Current Microbiology. 62 (6), 1802-1806 (2011).
- Abdoelrachman, R. Comparative investigations into the influence of the presence of bacteria on the life of pathogenic and apathogenic leptospirae. Antonie van Leeuwenhoek. 13 (1), 21-32 (1947).
- Singh, R., et al. Microbial diversity of biofilms in dental unit water systems. Applied and Environmental Microbiology. 69 (6), 3412-3420 (2003).
- Kumar, K. V., Lall, C., Raj, R. V., Vedhagiri, K., Vijayachari, P. Coexistence and survival of pathogenic leptospires by formation of biofilm with Azospirillum. FEMS Microbiology Ecology. 91 (6), 051 (2015).
- Yanagihara, Y., et al. Leptospira Is an environmental bacterium that grows in waterlogged soil. Microbiology Spectrum. 10 (2), 0215721 (2022).
- Gillespie, R. W., Ryno, J. Epidemiology of leptospirosis. American Journal of Public Health and Nation’s Health. 53 (6), 950-955 (1963).
- Bierque, E., et al. Leptospira interrogans retains direct virulence after long starvation in water. Current Microbiology. 77 (10), 3035-3043 (2020).
- Zhang, Y., Dai, B. Marking and detection of DNA of leptospires in the dot-blot and situ hybridization with digoxigenin-labeled probes. Journal of West China University of Medical Sciences. 23 (4), 353-435 (1992).
- Mérien, F., Amouriaux, P., Perolat, P., Baranton, G., Saint Girons, I. Polymerase chain reaction for detection of Leptospira spp. in clinical samples. Journal of Clinical Microbiology. 30 (9), 2219-2224 (1992).
- Veerapandian, R., et al. Silver enhanced nano-gold dot-blot immunoassay for leptospirosis. Journal of Microbiological Methods. 156, 20-22 (2019).
- Junpen, S., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Ishmael, F. T., Stellato, C. Principles and applications of polymerase chain reaction: basic science for the practicing physician. Annals of Allergy, Asthma & Immunology: Official Publication of the American College of Allergy, Asthma, & Immunology. 101 (4), 437-443 (2008).
- Boerner, B., Weigelt, W., Buhk, H. J., Castrucci, G., Ludwig, H. A sensitive and specific PCR/Southern blot assay for detection of bovine herpesvirus 4 in calves infected experimentally. Journal of Virological Methods. 83 (1-2), 169-180 (1999).
- Curry, E., Pratt, S. L., Kelley, D. E., Lapin, D. R., Gibbons, J. R. Use of a Combined duplex PCR/Dot-blot assay for more sensitive genetic characterization. Biochemistry Insights. 1, 35-39 (2008).
- Pilatti, M. M., Ferreira, S. d. e. A., de Melo, M. N., de Andrade, A. S. Comparison of PCR methods for diagnosis of canine visceral leishmaniasis in conjunctival swab samples. Research in Veterinary Science. 87 (2), 255-257 (2009).
- Conrads, G., et al. PCR reaction and dot-blot hybridization to monitor the distribution of oral pathogens within plaque samples of periodontally healthy individuals. Journal of Periodontology. 67 (10), 994-1003 (1996).
- Langa, S., et al. Differentiation of Enterococcus faecium from Lactobacillus delbrueckii subsp. bulgaricus and Streptococcus thermophilus strains by PCR and dot-blot hybridisation. International Journal of Food Microbiology. 88 (2-3), 197-200 (2003).
- Francesca, C., Lucilla, I., Marco, F., Giuseppe, C., Marisa, M. Identification of the unculturable bacteria Candidatus arthromitus in the intestinal content of trouts using dot-blot and Southern blot techniques. Veterinary Microbiology. 156 (3-4), 389-394 (2012).
- Arent, Z., Pardyak, L., Dubniewicz, K., Plachno, B. J., Kotula-Balak, M. Leptospira taxonomy: then and now. Medycyna Weterynaryjna. 78 (10), 489-496 (2022).
- Thibeaux, R., et al. Biodiversity of environmental Leptospira: Improving identification and revisiting the diagnosis. Frontiers in Microbiology. 9, 816 (2018).
- Carrillo-Casas, E. M., Hernández-Castro, R., Suárez-Güemes, F., de la Peña-Moctezuma, A. Selection of the internal control gene for real-time quantitative RT-PCR assays in temperature treated Leptospira. Current Microbiology. 56 (6), 539-546 (2008).
- Azali, M. A., Yean Yean, C., Harun, A., Aminuddin Baki A, N. N., Ismail, N. Molecular characterization of Leptospira spp. in environmental samples from North-Eastern Malaysia revealed a pathogenic strain, Leptospira alstonii. Journal of Tropical Medicine. 2016, 2060241 (2016).
- Ahmed, N., et al. Multilocus sequence typing method for identification and genotypic classification of pathogenic Leptospira species. Annals of Clinical Microbiology and Antimicrobials. 5, 28 (2006).
- Bourhy, P., Collet, L., Brisse, S., Picardeau, M. Leptospira mayottensis sp. nov., a pathogenic species of the genus Leptospira isolated from humans. International Journal of Systematic and Evolutionary Microbiology. 64, 4061-4067 (2014).
- Weiss, S., et al. An extended Multilocus Sequence Typing (MLST) scheme for rapid direct typing of Leptospira from clinical samples. PLOS Neglected Tropical Diseases. 10 (9), e0004996 (2016).
- Branger, C., et al. Polymerase chain reaction assay specific for pathogenic Leptospira based on the gene hap1 encoding the hemolysis-associated protein-1. FEMS Microbiology Letters. 243 (2), 437-445 (2005).
- Ren, S. X., et al. Unique physiological and pathogenic features of Leptospira interrogans revealed by whole-genome sequencing. Nature. 422 (6934), 888-893 (2003).
- Picardeau, M., et al. Genome sequence of the saprophyte Leptospira biflexa provides insights into the evolution of Leptospira and the pathogenesis of leptospirosis. PLOS One. 3 (2), e1607 (2008).
- Kafatos, F. C., Jones, C. W., Efstratiadis, A. Determination of nucleic acid sequence homologies and relative concentrations by a dot hybridization procedure. Nucleic Acids Research. 7 (6), 1541-1552 (1979).
- Bhat, A. I., Rao, G. P. Dot-blot hybridization technique. Characterization of Plant Viruses. , 303-321 (2020).
- Yadav, J. P., Batra, K., Singh, Y., Singh, M. Comparative evaluation of indirect-ELISA and Dot-blot assay for serodetection of Mycoplasma gallisepticum and Mycoplasma synoviae antibodies in poultry. Journal of Microbiological Methods. 189, 106317 (2021).
- Malinen, E., Kassinen, A., Rinttilä, T., Palva, A. Comparison of real-time PCR with SYBR Green I or 5′-nuclease assays and dot-blot hybridization with rDNA-targeted oligonucleotide probes in quantification of selected faecal bacteria. Microbiology. 149, 269-277 (2003).
- Wyss, C., et al. Treponema lecithinolyticum sp. nov., a small saccharolytic spirochaete with phospholipase A and C activities associated with periodontal diseases. International Journal of Systematic Bacteriology. 49, 1329-1339 (1999).
- Shah, J. S., I, D. C., Ward, S., Harris, N. S., Ramasamy, R. Development of a sensitive PCR-dot-blot assay to supplement serological tests for diagnosing Lyme disease. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 37 (4), 701-709 (2018).
- Niu, C., Wang, S., Lu, C. Development and evaluation of a dot-blot assay for rapid determination of invasion-associated gene ibeA directly in fresh bacteria cultures of E. coli. Folia microbiologica. 57 (6), 557-561 (2012).
- Wetherall, B. L., McDonald, P. J., Johnson, A. M. Detection of Campylobacter pylori DNA by hybridization with non-radioactive probes in comparison with a 32P-labeled probe. Journal of Medical Microbiology. 26 (4), 257-263 (1988).
- Kolk, A. H., et al. Detection of Mycobacterium tuberculosis in clinical samples by using polymerase chain reaction and a nonradioactive detection system. Journal of Clinical Microbiology. 30 (10), 2567-2575 (1992).
- Scherer, L. C., et al. PCR colorimetric dot-blot assay and clinical pretest probability for diagnosis of Pulmonary Tuberculosis in smear-negative patients. BMC Public Health. 7, 356 (2007).
- Armbruster, D. A., Pry, T. Limit of blank, limit of detection and limit of quantitation. The Clinical Biochemist Reviews. 29, S49-S52 (2008).
- Zhang, Y., Dai, B. Detection of Leptospira by dot-blot hybridization with photobiotin- and 32P-labeled DNA. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 130-132 (1992).
- Terpstra, W. J., Schoone, G. J., ter Schegget, J. Detection of leptospiral DNA by nucleic acid hybridization with 32P- and biotin-labeled probes. Journal of Medical Microbiology. 22 (1), 23-28 (1986).
- Shukla, J., Tuteja, U., Batra, H. V. DNA probes for identification of leptospires and disease diagnosis. The Southeast Asian Journal of Tropical Medicine and Public Health. 35 (2), 346-352 (2004).
- Jiang, N., Jin, B., Dai, B., Zhang, Y. Identification of pathogenic and nonpathogenic leptospires by recombinant probes. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 26 (1), 1-5 (1995).
- Fach, P., Trap, D., Guillou, J. P. Biotinylated probes to detect Leptospira interrogans on dot-blot hybridization or by in situ hybridization. Letters in Applied Microbiology. 12 (5), 171-176 (1991).
- Huang, N., Dai, B. Assay of genomic DNA homology among strains of different virulent leptospira by DNA hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 122-125 (1992).
- Dong, X., Dai, B., Chai, J. Homology study of leptospires by molecular hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (1), 1-4 (1992).
- Komminoth, P. Digoxigenin as an alternative probe labeling for in situ hybridization. Diagnostic Molecular Pathology: The American Journal of Surgical Pathology, part B. 1 (2), 142-150 (1992).
- Saengjaruk, P., et al. Diagnosis of human leptospirosis by monoclonal antibody-based antigen detection in urine. Journal of Clinical Microbiology. 40 (2), 480-489 (2002).
- Okuda, M., et al. Enzyme-linked immunosorbent assay for the detection of canine Leptospira antibodies using recombinant OmpL1 protein. The Journal of Veterinary Medical Science. 67 (3), 249-254 (2005).
- Suwimonteerabutr, J., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Kanagavel, M., et al. Peptide-specific monoclonal antibodies of Leptospiral LigA for acute diagnosis of leptospirosis. Scientific reports. 7 (1), 3250 (2017).
- Levett, P. N. Leptospirosis. Clinical Microbiology Reviews. 14 (2), 296-326 (2001).
- Monahan, A. M., Callanan, J. J., Nally, J. E. Proteomic analysis of Leptospira interrogans shed in urine of chronically infected hosts. Infection and Immunity. 76 (11), 4952-4958 (2008).
- Rojas, P., et al. Detection and quantification of leptospires in urine of dogs: a maintenance host for the zoonotic disease leptospirosis. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 29 (10), 1305-1309 (2010).

