הגנום עריכה בשורות תאים בתרבית של שימוש CRISPR-Cas
Summary
CRISPR-Cas היא טכנולוגיה רבת עוצמה להנדס הגנום מורכבים של צמחים ובעלי חיים. כאן, אנחנו פירוט נוהל לעריכת ביעילות הגנום האנושי באמצעות endonucleases רשויות אישורים שונים. אנחנו מדגישים שיקולים חשובים ופרמטרים עיצוב כדי למטב את יעילות העריכה.
Abstract
המערכת חזרה palindromic קצר באופן קבוע interspaced (CRISPR) באשכולות מתפקד באופן טבעי של חיידקי החסינות אדפטיבית, אך יש כבר בהצלחה לשנות את ייעודו הגנום הנדסה רבים אורגניזמים שונים. בדרך כלל, wildtype CRISPR הקשורים 9 (Cas9) או Cas12a endonuclease משמשת כדי לדבוק אתרים ספציפיים בגנום, אחרי אשר יתוקן מעבר כפול גדילי הדנ א באמצעות הסוף שאינם הומולוגיים הצטרפות לשביל (NHEJ) או את (תיקון מכוון הומולוגיה HDR) מסלול בהתאם תבנית התורם נעדרים או להציג בהתאמה. עד כה, CRISPR מערכות ממינים שונים חיידקי הוכחו להיות מסוגל לבצע עריכה הגנום בתאים בתרבית. עם זאת, למרות הפשטות לכאורה של הטכנולוגיה, מספר פרמטרים צריכים לקחת בחשבון, אשר לעיתים קרובות להשאיר משתמשים מבולבלים אודות הטוב ביותר לבצע את הגנום שלהם עריכת ניסויים. כאן, אנו מתארים שזרימת עבודה מלאה של הנבחנים: זיהוי של שיבוטים תא שנושאים שינויים DNA הרצוי, במטרה להקל על ביצוע מוצלח של הגנום עריכת ניסויים בקווים בתרבית של תאים. אנחנו האר שיקולים מרכזיים עבור משתמשים כדי לשים לב, כולל הבחירה של מערכת CRISPR, אורך מרווח, את העיצוב של תבנית התורם חד גדילי oligodeoxynucleotide (ssODN). נוכל לדמות כי זרימת עבודה זו תהיה שימושית ללימודים נוקאאוט גנטי, מחלת מידול המאמצים, או הדור של הכתב תא קווים.
Introduction
היכולת מהנדס הגנום של כל אורגניזם חי יש לא מעט אפליקציות ביו, הביו-טכנולוגיה, כגון התיקון של גורמי מחלות מוטציות, בניית דגמי הסלולר מדויק לחקר המחלה, או דור של החקלאי יבולים עם תכונות רצויות. מאז המאה ה, טכנולוגיות שונות פותחו עבור גנום הנדסה בתאי יונקים, כולל meganucleases1,2,3, אבץ אצבע nucleases4,5, או תמלול אפקטור activator דמוי nucleases (TALENs)6,7,8,9. עם זאת, טכנולוגיות קודמות אלה הם קשה לתוכנית או מייגע להרכיב, ובכך פוגעות שלהם אימוץ נרחב מחקר ובתעשייה.
בשנים האחרונות, באופן קבוע באשכולות interspaced חזרה palindromic קצר (CRISPR) – מערכת CRISPR-הקשורים (Ca) התפתחה גנום חדש חזק הנדסה טכנולוגיה10,11. במקור מערכת חיסונית אדפטיבית בחיידקים, עבר בהצלחה לפריסה הגנום שינוי בצמחים ובעלי חיים, כולל האדם. הסיבות העיקריות מדוע CRISPR-Cas צברה פופולריות רבה בזמן כל כך קצר זה הרכיב המביאה את endonuclease Cas מפתח, כגון Cas9 או Cas12a (ידוע גם בשם Cpf1), על המיקום הנכון הגנום הוא פשוט חתיכת קצר מדריך בודד chimeric RN (SgRNA), אשר היא פעולה פשוטה עיצוב וזול לסנתז. לאחר לאתר היעד האנזים Cas מתפקד בתור זוג מספריים מולקולרית ודבק ה-DNA מאוגד עם שלו RuvC, HNH או Nuc תחומים12,13,14. מעבר נטושים כפול וכתוצאה מכך (DSB) לאחר מכן תיקון על ידי התאים באמצעות קצה שאינם הומולוגיים להצטרף (NHEJ) או מסלול תיקון מכוון הומולוגיה (HDR). בהיעדרו של תבנית תיקון, DSB יתוקן על ידי השביל NHEJ לשגיאות, אשר יכולים להצמיח אקראיים מדומים ההוספה או המחיקה של נוקלאוטידים (indels) באתר לחתוך, ובכך לגרום frameshift מוטציות בגנים חלבונים. עם זאת, בנוכחות התורם תבנית המכילה את השינויים הרצויים של הדנ א, DSB יתוקן על ידי השביל HDR דיוק גבוה. סוגים נפוצים של תבניות התורם כוללים חד גדילי oligonucleotides (ssODNs), פלסמידים. לשעבר משמש בדרך כלל אם השינויים DNA המיועד קטנות (לדוגמה, שינוי של בסיס יחיד זוג רשימות), בזמן האחרון זה משמש בדרך כלל אם אנחנו רוצים להוסיף רצף ארוך יחסית (לדוגמה, רצף קידוד של חלבון פלואורסצנטי ירוק או GFP) לתוך מיקומה היעד.
הפעילות endonuclease של החלבון Cas דורש הנוכחות של protospacer הסמוך מוטיב (פאם) ב אתר היעד15. פאם של Cas9 נמצא בקצה 3′ של protospacer, פאם Cas12a (הנקראת גם Cpf1) נמצאת בקצה 5′ במקום16. Cas-המדריך RNA מורכב אין אפשרות להציג DSB אם פאם הוא נעדר17. לפיכך, פאם המקומות אילוץ על המיקומים גנומית איפה מסוגל לבקע נוקלאז Cas מסוים. למרבה המזל, nucleases Cas של מינים שונים של חיידקים בדרך כלל התערוכה פאם דרישות שונות. לפיכך, על ידי שילוב של המערכות CRISPR-Cas השונות שלנו ארגז כלים הנדסיים, אפשר להרחיב את מגוון אתרים אשר עשוי להיות ממוקד הגנום. יתר על כן, אנזים Cas טבעי שניתן הנדסה לאחור או התפתחו כדי לזהות רצפים פאם חלופיים, שמפרידה את היקף נגיש מניפולציה18,19,20גנומית מטרות.
למרות מספר מערכות CRISPR-Cas זמינים למטרות הנדסה הגנום, רוב המשתמשים של הטכנולוגיה יש הסתמך בעיקר על נוקלאז Cas9 מ הפישחה ינפל תומימת סטרפטוקוקוס (SpCas9) מסיבות רבות. קודם כל, זה דורש יחסית פשוט הגולה פאם, בניגוד רבים לחלבונים אחרים-Cas זה יכול לבקע רק בנוכחות PAMs מורכבים יותר. שנית, זה endonuclease Cas הראשון כדי לפרוס בהצלחה תאים אנושיים21,22,23,24. שלישית, SpCas9 הוא האנזים מאופיין בצורה הטובה ביותר עד כה. אם חוקר מבקש להשתמש נוקלאז Cas אחר, הוא או היא לעתים קרובות יהיה ברור על הדרך הטובה ביותר לעצב את הניסוי ותבצע כמה אנזימים אחרים בהקשרים ביולוגיים שונים, בהשוואה ל- SpCas9.
כדי להוסיף הבהרות לגבי את הביצועים היחסיים של מערכות CRISPR-Cas שונות, לאחרונה ערכנו השוואה שיטתית של חמש רשויות אישורים endonucleases – SpCas9, האנזים Cas9 מ Staphylococcus aureus (SaCas9), האנזים Cas9 מ- מנינגוקוקוס (NmCas9), האנזים Cas12a מן Acidaminococcus sp. BV3L6 (AsCas12a), האנזים Cas12a של החיידק Lachnospiraceae ND2006 (LbCas12a)25. להשוואה הוגן, הערכנו את nucleases רשויות אישורים שונים באמצעות אותה קבוצה של אתרי יעד ותנאים אחרים ניסיוני. המחקר גם מאפשרת עיצוב הפרמטרים עבור כל מערכת CRISPR-Cas, אשר ישמש כהפניה שימושי עבור משתמשים של הטכנולוגיה. . הנה, טוב יותר לאפשר לחוקרים לנצל CRISPR-Cas מערכת, אנו מספקים פרוטוקול צעד אחר צעד עבור הנדסה הגנום אופטימלית עם אנזימים שונים Cas9 ו- Cas12a (ראה איור 1). הפרוטוקול כוללת לא רק פרטים ניסיוני אבל שיקולי התכנון חשוב גם כדי להגדיל את הסבירות של תוצאה הנדסה מוצלחת הגנום בתאים בתרבית.
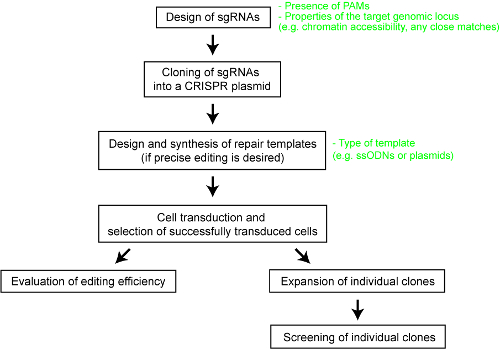
איור 1 : סקירה כללית של זרימת העבודה כדי ליצור הגנום נערך שורות תאים אנושיים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
Protocol
Representative Results
Discussion
מערכת CRISPR-Cas היא טכנולוגיה חזקה, מהפכני להנדס את הגנום ואת transcriptomes של צמחים ובעלי חיים. זנים חיידקיים רבים נמצאו יכילו מערכות CRISPR-Cas, אשר פוטנציאל עשוי להיות מותאם הגנום, transcriptome הנדסה למטרות44. למרות Cas9 endonuclease של סטרפטוקוקוס הפישחה ינפל תומימת (SpCas9) היה האנזים הראשון כדי לפ?…
Offenlegungen
The authors have nothing to disclose.
Acknowledgements
M.H.T. נתמך על ידי סוכנות מענק משרד המועצה המשותפת של מדע טכנולוגיה ומחקר (1431AFG103), המועצה הלאומית למחקר רפואי גרנט (OFIRG 0017 2016), קרן מחקר לאומי מעניק (NRF2013-THE001-046, NRF2013-THE001-093), משרד החינוך Tier 1 גרנט (RG50/17 (S)), סטארט-אפ הענק Nanyang הטכנולוגי מהאוניברסיטה, ו כספים עבור התחרות הבינלאומית גנטית הנדסת מכונות (iGEM) מאוניברסיטת Nanyang הטכנולוגי.
Materials
| T4 Polynucleotide Kinase (PNK) | NEB | M0201 | |
| Shrimp Alkaline Phosphatase (rSAP) | NEB | M0371 | |
| Tris-Acetate-EDTA (TAE) Buffer, 50X | 1st Base | BUF-3000-50X4L | Dilute to 1X before use. The 1X solution contains 40 mM Tris, 20 mM acetic acid, and 1 mM EDTA. |
| Tris-EDTA (TE) Buffer, 10X | 1st Base | BUF-3020-10X4L | Dilute to 1X before use. The 1X solution contains 10 mM Tris (pH 8.0) and 1 mM EDTA. |
| BbsI | NEB | R0539 | |
| BsmBI | NEB | R0580 | |
| T4 DNA Ligase | NEB | M0202 | 400,000 units/ml |
| Quick Ligation Kit | NEB | M2200 | An alternative to T4 DNA Ligase. |
| Rapid DNA Ligation Kit | Thermo Scientific | K1423 | An alternative to T4 DNA Ligase. |
| Zero Blunt TOPO PCR Cloning Kit | Thermo Scientific | 451245 | The salt solution comes with the TOPO vector in the kit. |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Kit for Gibson assembly. |
| One Shot Stbl3 Chemically Competent E.Coli | Thermo Scientific | C737303 | |
| LB Broth (Lennox), powder | Sigma Aldrich | L3022 | Reconstitute in ddH20, and autoclave before use |
| LB Broth with Agar (Lennox), powder | Sigma Aldrich | L2897 | Reconstitute in ddH20, and autoclave before use |
| SOC media | – | – | 2.5 mM KCl, 10 mM MgCl2, 20 mM glucose in 1 L of LB Broth |
| Ampicillin (Sodium), USP Grade | Gold Biotechnology | A-301 | |
| REDiant 2X PCR Mastermix | 1st Base | BIO-5185 | |
| Agarose | 1st Base | BIO-1000 | |
| T7 Endonuclease I | NEB | M0302 | |
| Plasmid DNA Extraction Miniprep Kit | Favorgen | FAPDE 300 | |
| Dulbecco's Modified Eagle Medium (DMEM), High Glucose | Hyclone | SH30081.01 | 4.5 g/L Glucose, no L-glutamine, HEPES and Sodium Pyruvate |
| L-Glutamine, 200mM | Gibco | 25030 | |
| Penicillin-Streptomycin, 10, 000U/mL | Gibco | 15140 | |
| 0.25% Trypsin-EDTA, 1X | Gibco | 25200 | |
| Fetal Bovine Serum | Hyclone | SV30160 | FBS is heat inactivated before use at 56 oC for 30 min |
| Phosphate Buffered Saline, 1X | Gibco | 20012 | |
| jetPRIME transfection reagent | Polyplus Transfection | 114-75 | |
| QuickExtract DNA Extraction Solution, 1.0 | Epicentre | LUCG-QE09050 | |
| ISOLATE II Genomic DNA Kit | Bioline | BIO-52067 | An alternative to QuickExtract |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491 | |
| Deoxynucleotide (dNTP) Solution Mix | NEB | N0447 | |
| 6X DNA Loading Dye | Thermo Scientific | R0611 | 10 mM Tris-HCl (pH 7.6) 0.03% bromophenol blue, 0.03% xylene cyanol FF, 60% glycerol, 60 mM EDTA |
| Protease Inhibitor Cocktail, Set3 | Merck | 539134 | |
| Nitrocellulose membrane, 0.2µm | Bio-Rad | 1620112 | |
| Tris-glycine-SDS buffer, 10X | Bio-Rad | 1610772 | Dilute to 1X before use. The 1x solution contains 25 mM Tris, 192 mM glycine, and 0.1% SDS. |
| Tris-glycine buffer, 10X | 1st base | BUF-2020 | Dilute to 1X before use. The 1x solution contains 25 mM Tris and 192 mM glycine. |
| Ponceau S solution | Sigma Aldrich | P7170 | |
| TBS, 20X | 1st base | BUF-3030 | Dilute to 1X before use. The 1x solution contains 25 mM Tris-HCl (pH 7.5) and 150 mM NaCl. |
| Tween 20 | Sigma Aldrich | P9416 | |
| Skim Milk for immunoassay | Nacalai Tesque | 31149-75 | |
| WesternBright Sirius-femtogram HRP | Advansta | K12043 | |
| Antibody for β-actin (C4) | Santa Cruz Biotechnology | sc-47778 | Lot number: C0916 |
| MiSeq system | Illumina | SY-410-1003 | |
| NanoDrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit fluorometer | Thermo Scientific | Q33226 | |
| EVOS FL Cell Imaging System | Thermo Scientific | AMF4300 | |
| CRISPR plasmid: pSpCas9(BB)-2A-GFP (PX458) | Addgene | 48138 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: pX601-AAV-CMV::NLS-SaCas9-NLS-3xHA-bGHpA | Addgene | 61591 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: xCas9 3.7 | Addgene | 108379 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: pX330-U6-Chimeric_BB-CBh-hSpCas9 | Addgene | 42230 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: hCas9 | Addgene | 41815 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: eSpCas9(1.1) | Addgene | 71814 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: VP12 (SpCas9-HF1) | Addgene | 72247 | Dual vector system: The gRNA is expressed from a different plasmid. |
Referenzen
- Epinat, J. C., et al. A novel engineered meganuclease induces homologous recombination in yeast and mammalian cells. Nucleic Acids Research. 31 (11), 2952-2962 (2003).
- Arnould, S., et al. Engineered I-CreI derivatives cleaving sequences from the human XPC gene can induce highly efficient gene correction in mammalian cells. Journal of Molecular Biology. 371 (1), 49-65 (2007).
- Chapdelaine, P., Pichavant, C., Rousseau, J., Paques, F., Tremblay, J. P. Meganucleases can restore the reading frame of a mutated dystrophin. Gene Therapy. 17 (7), 846-858 (2010).
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetik. 188 (4), 773-782 (2011).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nature Biotechnology. 29 (2), 143-148 (2011).
- Zhang, F., et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription. Nature Biotechnology. 29 (2), 149-153 (2011).
- Boch, J., et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science. 326 (5959), 1509-1512 (2009).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Nishimasu, H., et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell. 156 (5), 935-949 (2014).
- Yamano, T., et al. Crystal Structure of Cpf1 in Complex with Guide RNA and Target DNA. Cell. 165 (4), 949-962 (2016).
- Swarts, D. C., Mosterd, C., van Passel, M. W., Brouns, S. J. CRISPR interference directs strand specific spacer acquisition. PLoS One. 7 (4), e35888 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Kleinstiver, B. P., et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition. Nature Biotechnology. 33 (12), 1293-1298 (2015).
- Kleinstiver, B. P., et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature. 523 (7561), 481-485 (2015).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Jinek, M., et al. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Cho, S. W., Kim, S., Kim, J. M., Kim, J. S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nature Biotechnology. 31 (3), 230-232 (2013).
- Wang, Y., et al. Systematic evaluation of CRISPR-Cas systems reveals design principles for genome editing in human cells. Genome Biology. 19 (1), 62 (2018).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences U S A. 110 (39), 15644-15649 (2013).
- Kim, E., et al. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni. Nature Communications. 8, 14500 (2017).
- Edraki, A., et al. A Compact, High-Accuracy Cas9 with a Dinucleotide PAM for In Vivo Genome Editing. Molecular Cell. , (2018).
- Chatterjee, P., Jakimo, N., Jacobson, J. M. Minimal PAM specificity of a highly similar SpCas9 ortholog. Science Advances. 4 (10), (2018).
- Muller, M., et al. Streptococcus thermophilus CRISPR-Cas9 Systems Enable Specific Editing of the Human Genome. Mol Therapy. 24 (3), 636-644 (2016).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Boratyn, G. M., et al. BLAST: a more efficient report with usability improvements. Nucleic Acids Research. 41 (Web Server issue), W29-W33 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Montague, T. G., Cruz, J. M., Gagnon, J. A., Church, G. M., Valen, E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research. 42 (Web Server issue), W401-W407 (2014).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 122-123 (2014).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30 (10), 1473-1475 (2014).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Richardson, C. D., Ray, G. J., Bray, N. L., Corn, J. E. Non-homologous DNA increases gene disruption efficiency by altering DNA repair outcomes. Nature Communications. 7, 12463 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biology. 18 (1), 35 (2017).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shmakov, S., et al. Diversity and evolution of class 2 CRISPR-Cas systems. Nature Reviews Microbiology. 15 (3), 169-182 (2017).
- Moreno-Mateos, M. A., et al. CRISPR-Cpf1 mediates efficient homology-directed repair and temperature-controlled genome editing. Nature Communications. 8 (1), 2024 (2017).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Yang, L., et al. Optimization of scarless human stem cell genome editing. Nucleic Acids Research. 41 (19), 9049-9061 (2013).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).

