Edición del genoma en las líneas celulares de mamíferos utilizando CRISPR-Cas
Summary
CRISPR-Cas es una tecnología de gran alcance para los complejos genomas de plantas y animales. Aquí detallamos un protocolo eficiente editar el genoma humano utilizando diferentes endonucleasas de Cas. Se destacan importantes consideraciones y parámetros de diseño para optimizar la eficiencia de la edición.
Abstract
El sistema regularmente otro corto repite palindrómico (CRISPR) Cluster funciona naturalmente en bacteria inmunidad adaptativa, pero ha sido repurposed con éxito para la ingeniería del genoma de muchos organismos vivos diferentes. Comúnmente, el wildtype CRISPR asociados 9 (Cas9) o endonucleasa de Cas12a se utiliza para unirse a sitios específicos en el genoma, después de que la rotura de doble hebra de ADN se repara mediante la no homóloga se terminan uniendo (NHEJ) camino o la reparación dirigida de homología ( Camino HDR) dependiendo de si es una plantilla de donantes ausente o presente respectivamente. Hasta la fecha, sistemas CRISPR de diferentes especies bacterianas han demostrado ser capaces de realizar la edición del genoma en células de mamíferos. Sin embargo, a pesar de la aparente simplicidad de la tecnología, varios parámetros de diseño necesitan ser considerados, que a menudo dejan perplejos sobre la forma mejor para llevar a cabo su genoma experimentos de edición de usuarios. Aquí, describimos un completo flujo de trabajo de diseño experimental para identificación de clones de células que llevan a modificaciones de ADN deseados, con el objetivo de facilitar la ejecución exitosa del genoma edición experimentos en líneas celulares mamíferos. Destacamos consideraciones clave para los usuarios a tomar nota, incluyendo la elección del sistema CRISPR, la longitud del espaciador y el diseño de una plantilla de donantes oligodeoxynucleotide monocatenario (ssODN). Imaginamos que este flujo de trabajo será útil para estudios gene knockout, modelado de esfuerzos, la enfermedad o líneas celulares de la generación del reportero.
Introduction
La capacidad para diseñar el genoma de cualquier organismo vivo tiene muchas aplicaciones biomédicas y biotecnológicas, como la corrección de enfermedad-causar mutaciones, construcción de modelos celulares precisos estudios de la enfermedad, o generación de agrícola cultivos con características deseables. Puesto que la vuelta del siglo, se han desarrollado diversas tecnologías para la ingeniería del genoma en células de mamíferos, incluyendo meganucleases1,2,3, zinc finger nucleasas4,5, o transcripción efector activador como nucleasas (TALENs)6,7,8,9. Sin embargo, estas tecnologías anteriores son programa difícil o tedioso de montar, tal modo obstaculizando su adopción generalizada en la investigación y la industria.
En los últimos años, el cluster regularmente otro corto repite palindrómico (CRISPR) – sistema CRISPR-asociado (Cas) se ha convertido en un genoma nuevo potente ingeniería tecnología10,11. Originalmente un sistema inmune adaptante en las bacterias, ha sido exitosamente implementado para la modificación del genoma en plantas y animales, incluyendo seres humanos. La razón principal por qué CRISPR-Cas ha ganado tanta popularidad en tan corto tiempo es el elemento que trae la clave endonucleasa de Cas, como Cas9 o Cas12a (también conocido como Cpf1), en la ubicación correcta en el genoma es simplemente una pequeña pieza de guía solo quimérica RN A (sgRNA), que es de diseño sencillo y barato sintetizar. Después de ser reclutado al sitio de destino, la enzima Cas funciona como un par de tijeras moleculares y hiende la DNA dependiente con su RuvC, HNH o Nuc dominios12,13,14. Posteriormente se repara la rotura trenzada doble resultante (de diferencias OSD) por las células a través de la vía de reparación dirigido por homología (HDR) o no homóloga se terminan uniendo (NHEJ). En la ausencia de una plantilla de reparación, el OSD es reparado por la vía NHEJ propenso, que puede dar lugar a pseudo-random inserción o deleción de nucleótidos (indels) en el sitio de corte, potencialmente causando mutágeno ‘ frameshift ‘ mutaciones en genes de la proteína-codificación. Sin embargo, en presencia de una plantilla de donante que contiene los cambios de ADN deseados, el OSD es reparado por la vía HDR de alta fidelidad. Tipos comunes de donantes plantillas son oligonucleótidos de cadena simple (ssODNs) y plásmidos. El primero se utiliza normalmente si los cambios previstos de DNA son pequeños (por ejemplo, alteración de un solo par de base), mientras que este último se suele utilizar si se desea insertar una secuencia relativamente larga (por ejemplo, la secuencia de codificación de una proteína verde fluorescente o GFP) en el lugar de destino.
La actividad endonucleasa de la proteína Cas requiere la presencia de un motivo adyacente de protospacer (PAM) en el sitio de destino15. PAM de Cas9 es el extremo 3′ de la protospacer, mientras que el PAM de Cas12a (también llamado Cpf1) está en el extremo 5′ en lugar16. La guía de Cas RNA complejo es incapaz de introducir un OSD si el PAM es ausente17. Por lo tanto, el PAM coloca una restricción en las localizaciones genómicas donde una nucleasa Cas particular es capaz de unirse. Afortunadamente, las nucleasas de la Cas de diferentes especies bacterianas típicamente exhiben diversos requisitos de PAM. Por lo tanto, al integrar varios sistemas de CRISPR-Cas en nuestra caja de herramientas de ingeniería, podemos ampliar la gama de sitios que pueden ser objeto de un genoma. Además, una enzima natural de la Cas puede ser diseñado o evolucionado para reconocer secuencias de PAM alternativas, ampliando aún más el alcance de objetivos genómicas accesibles a la manipulación18,19,20.
Aunque varios sistemas CRISPR-Cas están disponibles para propósitos de ingeniería del genoma, la mayoría de usuarios de la tecnología ha dependido principalmente la nucleasa Cas9 de Streptococcus pyogenes (SpCas9) por múltiples razones. En primer lugar, requiere un PAM relativamente simplemente NGG, a diferencia de muchas otras proteínas de Cas que pueden unirse sólo en presencia de PAM más compleja. En segundo lugar, es la primera endonucleasa de Cas a ser implementado con éxito en células humanas21,22,23,24. En tercer lugar, SpCas9 es la enzima mejor caracterizada hasta la fecha. Si un investigador desea utilizar otro nucleasa de Cas, él o ella a menudo sería confusa acerca de la mejor manera de diseñar el experimento y así otras enzimas llevará a cabo en diferentes contextos biológicos comparados con SpCas9.
Para dar claridad al desempeño relativo de los diferentes sistemas de CRISPR-Cas, recientemente hemos realizado una comparación sistemática de cinco endonucleases de Cas-SpCas9, la enzima Cas9 de Staphylococcus aureus (SaCas9), la enzima Cas9 de Meningitidis de la Neisseria (NmCas9), la enzima Cas12a de Acidaminococcus SP. BV3L6 (AsCas12a) y la enzima Cas12a de Lachnospiraceae bacteria ND2006 (LbCas12a)25. Para una comparación justa, se evaluaron las diversas nucleasas Cas mediante el mismo conjunto de sitios de destino y otras condiciones experimentales. Los parámetros de diseño del estudio también delineado para cada sistema CRISPR-Cas, que serviría como una referencia útil para los usuarios de la tecnología. Aquí, mejor permiten a los investigadores a hacer uso de la Cas CRISPR sistema, proporcionamos un protocolo paso a paso para la ingeniería del genoma óptima con los distintos enzimas Cas9 y Cas12a (ver figura 1). El Protocolo no sólo incluye detalles experimentales pero también importantes consideraciones para maximizar la probabilidad de un resultado de ingeniería exitosa del genoma en células de mamíferos.
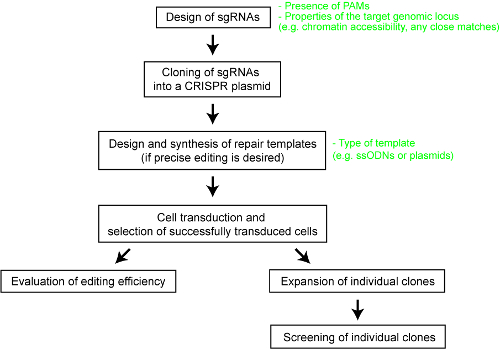
Figura 1 : Una visión general del flujo de trabajo para generar el genoma editado líneas celulares humanas. Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
El sistema CRISPR-Cas es una potente y revolucionaria tecnología diseñar genomas los transcriptomas de plantas y animales. Numerosas especies bacterianas se han encontrado para contener sistemas CRISPR-Cas, que potencialmente pueden ser adaptados para el genoma y transcriptoma ingeniería propósitos44. Aunque la endonucleasa Cas9 de Streptococcus pyogenes (SpCas9) fue la primera enzima a implementarse con éxito en células humanas21,<sup class="xref"…
Offenlegungen
The authors have nothing to disclose.
Acknowledgements
M.H.T. es apoyada por una agencia para la concesión de oficina conjunta del Consejo de ciencia, tecnología y de investigación (1431AFG103), un Consejo de investigación médica nacional conceder (OFIRG/0017/2016), otorga la Fundación Nacional de investigación (NRF2013-THE001-046 y NRF2013-THE001-093), un Subvención del Ministerio de educación nivel 1 (RG50/17 (S)), una startup becado por Universidad Tecnológica de Nanyang y fondos para la competencia internacional genéticamente máquina de la ingeniería (iGEM) de la Universidad Tecnológica de Nanyang.
Materials
| T4 Polynucleotide Kinase (PNK) | NEB | M0201 | |
| Shrimp Alkaline Phosphatase (rSAP) | NEB | M0371 | |
| Tris-Acetate-EDTA (TAE) Buffer, 50X | 1st Base | BUF-3000-50X4L | Dilute to 1X before use. The 1X solution contains 40 mM Tris, 20 mM acetic acid, and 1 mM EDTA. |
| Tris-EDTA (TE) Buffer, 10X | 1st Base | BUF-3020-10X4L | Dilute to 1X before use. The 1X solution contains 10 mM Tris (pH 8.0) and 1 mM EDTA. |
| BbsI | NEB | R0539 | |
| BsmBI | NEB | R0580 | |
| T4 DNA Ligase | NEB | M0202 | 400,000 units/ml |
| Quick Ligation Kit | NEB | M2200 | An alternative to T4 DNA Ligase. |
| Rapid DNA Ligation Kit | Thermo Scientific | K1423 | An alternative to T4 DNA Ligase. |
| Zero Blunt TOPO PCR Cloning Kit | Thermo Scientific | 451245 | The salt solution comes with the TOPO vector in the kit. |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Kit for Gibson assembly. |
| One Shot Stbl3 Chemically Competent E.Coli | Thermo Scientific | C737303 | |
| LB Broth (Lennox), powder | Sigma Aldrich | L3022 | Reconstitute in ddH20, and autoclave before use |
| LB Broth with Agar (Lennox), powder | Sigma Aldrich | L2897 | Reconstitute in ddH20, and autoclave before use |
| SOC media | – | – | 2.5 mM KCl, 10 mM MgCl2, 20 mM glucose in 1 L of LB Broth |
| Ampicillin (Sodium), USP Grade | Gold Biotechnology | A-301 | |
| REDiant 2X PCR Mastermix | 1st Base | BIO-5185 | |
| Agarose | 1st Base | BIO-1000 | |
| T7 Endonuclease I | NEB | M0302 | |
| Plasmid DNA Extraction Miniprep Kit | Favorgen | FAPDE 300 | |
| Dulbecco's Modified Eagle Medium (DMEM), High Glucose | Hyclone | SH30081.01 | 4.5 g/L Glucose, no L-glutamine, HEPES and Sodium Pyruvate |
| L-Glutamine, 200mM | Gibco | 25030 | |
| Penicillin-Streptomycin, 10, 000U/mL | Gibco | 15140 | |
| 0.25% Trypsin-EDTA, 1X | Gibco | 25200 | |
| Fetal Bovine Serum | Hyclone | SV30160 | FBS is heat inactivated before use at 56 oC for 30 min |
| Phosphate Buffered Saline, 1X | Gibco | 20012 | |
| jetPRIME transfection reagent | Polyplus Transfection | 114-75 | |
| QuickExtract DNA Extraction Solution, 1.0 | Epicentre | LUCG-QE09050 | |
| ISOLATE II Genomic DNA Kit | Bioline | BIO-52067 | An alternative to QuickExtract |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491 | |
| Deoxynucleotide (dNTP) Solution Mix | NEB | N0447 | |
| 6X DNA Loading Dye | Thermo Scientific | R0611 | 10 mM Tris-HCl (pH 7.6) 0.03% bromophenol blue, 0.03% xylene cyanol FF, 60% glycerol, 60 mM EDTA |
| Protease Inhibitor Cocktail, Set3 | Merck | 539134 | |
| Nitrocellulose membrane, 0.2µm | Bio-Rad | 1620112 | |
| Tris-glycine-SDS buffer, 10X | Bio-Rad | 1610772 | Dilute to 1X before use. The 1x solution contains 25 mM Tris, 192 mM glycine, and 0.1% SDS. |
| Tris-glycine buffer, 10X | 1st base | BUF-2020 | Dilute to 1X before use. The 1x solution contains 25 mM Tris and 192 mM glycine. |
| Ponceau S solution | Sigma Aldrich | P7170 | |
| TBS, 20X | 1st base | BUF-3030 | Dilute to 1X before use. The 1x solution contains 25 mM Tris-HCl (pH 7.5) and 150 mM NaCl. |
| Tween 20 | Sigma Aldrich | P9416 | |
| Skim Milk for immunoassay | Nacalai Tesque | 31149-75 | |
| WesternBright Sirius-femtogram HRP | Advansta | K12043 | |
| Antibody for β-actin (C4) | Santa Cruz Biotechnology | sc-47778 | Lot number: C0916 |
| MiSeq system | Illumina | SY-410-1003 | |
| NanoDrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit fluorometer | Thermo Scientific | Q33226 | |
| EVOS FL Cell Imaging System | Thermo Scientific | AMF4300 | |
| CRISPR plasmid: pSpCas9(BB)-2A-GFP (PX458) | Addgene | 48138 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: pX601-AAV-CMV::NLS-SaCas9-NLS-3xHA-bGHpA | Addgene | 61591 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: xCas9 3.7 | Addgene | 108379 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: pX330-U6-Chimeric_BB-CBh-hSpCas9 | Addgene | 42230 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: hCas9 | Addgene | 41815 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: eSpCas9(1.1) | Addgene | 71814 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: VP12 (SpCas9-HF1) | Addgene | 72247 | Dual vector system: The gRNA is expressed from a different plasmid. |
Referenzen
- Epinat, J. C., et al. A novel engineered meganuclease induces homologous recombination in yeast and mammalian cells. Nucleic Acids Research. 31 (11), 2952-2962 (2003).
- Arnould, S., et al. Engineered I-CreI derivatives cleaving sequences from the human XPC gene can induce highly efficient gene correction in mammalian cells. Journal of Molecular Biology. 371 (1), 49-65 (2007).
- Chapdelaine, P., Pichavant, C., Rousseau, J., Paques, F., Tremblay, J. P. Meganucleases can restore the reading frame of a mutated dystrophin. Gene Therapy. 17 (7), 846-858 (2010).
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetik. 188 (4), 773-782 (2011).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nature Biotechnology. 29 (2), 143-148 (2011).
- Zhang, F., et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription. Nature Biotechnology. 29 (2), 149-153 (2011).
- Boch, J., et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science. 326 (5959), 1509-1512 (2009).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Nishimasu, H., et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell. 156 (5), 935-949 (2014).
- Yamano, T., et al. Crystal Structure of Cpf1 in Complex with Guide RNA and Target DNA. Cell. 165 (4), 949-962 (2016).
- Swarts, D. C., Mosterd, C., van Passel, M. W., Brouns, S. J. CRISPR interference directs strand specific spacer acquisition. PLoS One. 7 (4), e35888 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Kleinstiver, B. P., et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition. Nature Biotechnology. 33 (12), 1293-1298 (2015).
- Kleinstiver, B. P., et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature. 523 (7561), 481-485 (2015).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Jinek, M., et al. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Cho, S. W., Kim, S., Kim, J. M., Kim, J. S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nature Biotechnology. 31 (3), 230-232 (2013).
- Wang, Y., et al. Systematic evaluation of CRISPR-Cas systems reveals design principles for genome editing in human cells. Genome Biology. 19 (1), 62 (2018).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences U S A. 110 (39), 15644-15649 (2013).
- Kim, E., et al. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni. Nature Communications. 8, 14500 (2017).
- Edraki, A., et al. A Compact, High-Accuracy Cas9 with a Dinucleotide PAM for In Vivo Genome Editing. Molecular Cell. , (2018).
- Chatterjee, P., Jakimo, N., Jacobson, J. M. Minimal PAM specificity of a highly similar SpCas9 ortholog. Science Advances. 4 (10), (2018).
- Muller, M., et al. Streptococcus thermophilus CRISPR-Cas9 Systems Enable Specific Editing of the Human Genome. Mol Therapy. 24 (3), 636-644 (2016).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Boratyn, G. M., et al. BLAST: a more efficient report with usability improvements. Nucleic Acids Research. 41 (Web Server issue), W29-W33 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Montague, T. G., Cruz, J. M., Gagnon, J. A., Church, G. M., Valen, E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research. 42 (Web Server issue), W401-W407 (2014).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 122-123 (2014).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30 (10), 1473-1475 (2014).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Richardson, C. D., Ray, G. J., Bray, N. L., Corn, J. E. Non-homologous DNA increases gene disruption efficiency by altering DNA repair outcomes. Nature Communications. 7, 12463 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biology. 18 (1), 35 (2017).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shmakov, S., et al. Diversity and evolution of class 2 CRISPR-Cas systems. Nature Reviews Microbiology. 15 (3), 169-182 (2017).
- Moreno-Mateos, M. A., et al. CRISPR-Cpf1 mediates efficient homology-directed repair and temperature-controlled genome editing. Nature Communications. 8 (1), 2024 (2017).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Yang, L., et al. Optimization of scarless human stem cell genome editing. Nucleic Acids Research. 41 (19), 9049-9061 (2013).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).

