Modifica di genoma in linee cellulari di mammifero usando CRISPR-Cas
Summary
CRISPR-Cas è una potente tecnologia per progettare i complessi genomi delle piante e degli animali. Qui, dettagliamo un protocollo efficiente modificare il genoma umano usando diverse endonucleasi di Cas. Evidenziamo considerazioni importanti e parametri di progettazione per ottimizzare l’efficienza di editing.
Abstract
Il sistema regolarmente interspaziati breve palindromi ripetizioni (CRISPR) cluster funziona naturalmente nell’immunità adattiva batterica, ma è stato reimpiegato con successo per l’ingegneria del genoma in molti organismi differenti. Più comunemente, il wildtype CRISPR associato 9 (Cas9) o dell’endonucleasi Cas12a sono usato per fendere siti specifici nel genoma, dopo che la rottura del doppio filamento di DNA è riparata tramite l’estremità non-omologo adesione via (NHEJ) o la riparazione di omologia-diretto ( Via di HDR) a seconda che sia un modello di donatore assenti o presenti rispettivamente. Ad oggi, sistemi CRISPR da diverse specie batteriche sono stati indicati per essere in grado di eseguire l’editing genomico in cellule di mammifero. Tuttavia, nonostante l’apparente semplicità della tecnologia, più parametri di progettazione devono essere considerati, che spesso lasciano gli utenti perplessi su come meglio per svolgere il loro genoma esperimenti di editing. Qui, descriviamo un flusso di lavoro completo dal disegno sperimentale identificazione di cloni di cellule che trasportano desiderati modificazioni del DNA, con l’obiettivo di facilitare la corretta esecuzione di esperimenti in linee cellulari di mammifero di editing genomico. Evidenziamo le considerazioni chiave per gli utenti di prendere nota, tra cui la scelta del sistema CRISPR, la lunghezza del distanziale e la progettazione di un modello di donatore singolo filamento oligodeoxynucleotide (ssODN). Immaginiamo che questo flusso di lavoro sarà utile per gli studi di gene knockout, modellazione di sforzi, la malattia o la generazione di reporter linee cellulari.
Introduction
La capacità di progettare il genoma di ogni organismo vivente ha molte applicazioni biomediche e biotecnologiche, come la correzione di malattia-causare mutazioni, costruzione di accurati modelli cellulari per lo studio di malattia, o la generazione di agricolo colture con caratteristiche desiderabili. Poiché la girata del secolo, diverse tecnologie sono state sviluppate per l’ingegneria del genoma in cellule di mammiferi, tra cui meganucleases1,2,3, zinco dito nucleasi4,5, o trascrizione dell’effettore attivatore-come nucleasi (TALENs)6,7,8,9. Tuttavia, queste tecnologie precedenti sono difficili da programma o noioso da montare, quindi che ostacolano l’adozione diffusa nella ricerca e nell’industria.
Negli ultimi anni, il cluster regolarmente intervallate brevi ripetizioni palindromi (CRISPR) – sistema di CRISPR-collegata (Cas) è emerso come un potente nuovo genoma ingegneria tecnologia10,11. Originariamente un sistema immunitario adattativo nei batteri, è stato correttamente distribuito per la modificazione del genoma in piante e animali, compreso gli esseri umani. Un motivo principale perché CRISPR-Cas ha guadagnato popolarità così tanto in così poco tempo che è l’elemento che porta la chiave dell’endonucleasi di Cas, ad esempio Cas9 o Cas12a (noto anche come Cpf1), nella posizione corretta nel genoma è semplicemente un breve pezzo di guida singola chimerico RN A (sgRNA), che è al design semplice ed economico per sintetizzare. Dopo essere reclutato nel sito di destinazione, l’enzima Cas funziona come un paio di forbici molecolari e si unirà il DNA associato con relativi RuvC, HNH o Nuc domini12,13,14. La risultante doppia interruzione incagliato (DSB) è più riparato dalle cellule via via regia di omologia repair (HDR) o non-omologo fine unirsi (NHEJ). In assenza di un modello di riparazione, il DSB è riparato dalla via NHEJ errori, che possa dar luogo a pseudo-caso inserimento o l’eliminazione di nucleotidi (indels) presso il sito di taglio, causando potenzialmente frameshift mutazioni in geni di proteina-codificazione. Tuttavia, in presenza di un modello di donatore che contiene le modifiche desiderate di DNA, il DSB è riparato dal pathway HDR ad alta fedeltà. Comuni tipi di modelli di donatore includono oligonucleotidi a singolo filamento (ssODNs) e plasmidi. Il primo viene in genere utilizzato se i previsti cambiamenti del DNA sono piccoli (per esempio, alterazione di un singolo paio di base), mentre quest’ultimo viene in genere utilizzato se si vuole inserire una sequenza relativamente lunga (ad esempio, la sequenza di codificazione di una proteina fluorescente verde o GFP) in luogo del bersaglio.
L’attività endonucleasica della proteina Cas richiede la presenza di un motivo di protospacer adiacenti (PAM) presso il sito di destinazione15. Il PAM di Cas9 è all’estremità 3′ del protospacer, mentre il PAM di Cas12a (chiamato anche Cpf1) è invece all’estremità 5′16. La Cas-guida RNA complesso è in grado di introdurre un DSB se il PAM è assente17. Da qui, il PAM luoghi un vincolo sulle posizioni genomiche dove una particolare nucleasi di Cas è in grado di fendere. Fortunatamente, nucleasi Cas da diverse specie batteriche in genere presentano diversi requisiti di PAM. Quindi, integrando i vari sistemi di CRISPR-Cas nel nostro toolbox di ingegneria, possiamo espandere la gamma di siti che possono essere mirati in un genoma. Inoltre, un enzima naturale Cas possa essere progettato o si è evoluto per riconoscere sequenze alternative di PAM, ampliando ulteriormente l’ambito di target genomici accessibile a manipolazione18,19,20.
Anche se più sistemi CRISPR-Cas sono disponibili per scopi di ingegneria del genoma, maggior parte degli utenti della tecnologia si affidano principalmente le nucleasi Cas9 da Streptococcus pyogenes (SpCas9) per molteplici ragioni. Innanzitutto, richiede un PAM relativamente semplicemente NGG, a differenza di molte altre proteine di Cas possono fendere solo in presenza di più complesse PAMs. In secondo luogo, è il primo endonucleasi di Cas per essere impiegati con successo in cellule umane21,22,23,24. In terzo luogo, la SpCas9 è di gran lunga l’enzima meglio caratterizzato fino ad oggi. Se un ricercatore vuole utilizzare un altro nucleasi di Cas, è possibile che lui o lei spesso sarebbe poco chiara sul modo migliore per progettare l’esperimento e come ben altri enzimi si esibiranno in diversi contesti biologici rispetto ai SpCas9.
Per fornire chiarezza per le prestazioni relative dei differenti sistemi di CRISPR-Cas, recentemente abbiamo effettuato un confronto sistematico delle cinque endonucleasi Cas – SpCas9, l’enzima Cas9 da Staphylococcus aureus (SaCas9), l’enzima di Cas9 da Neisseria meningitidis (NmCas9), l’enzima di Cas12a da Thermoanaerobacter SP. BV3L6 (AsCas12a) e l’enzima Cas12a dal batterio Lachnospiraceae ND2006 (LbCas12a)25. Per un confronto equo, abbiamo valutato le varie nucleasi Cas utilizzando lo stesso set di siti di destinazione e le altre condizioni sperimentali. I parametri di progettazione di studio anche delineato per ciascun sistema CRISPR-Cas, che dovrebbe servire come un utile riferimento per gli utenti della tecnologia. Qui, per meglio consentire ai ricercatori di fare uso della CRISPR-Cas sistema, forniamo un protocollo dettagliato per l’ingegneria di genoma ottimale con differenti enzimi Cas9 e Cas12a (Vedi Figura 1). Il protocollo comprende non solo i dettagli sperimentali ma anche importanti considerazioni di progettazione per massimizzare la probabilità di un risultato di successo genoma Ingegneria in cellule di mammifero.
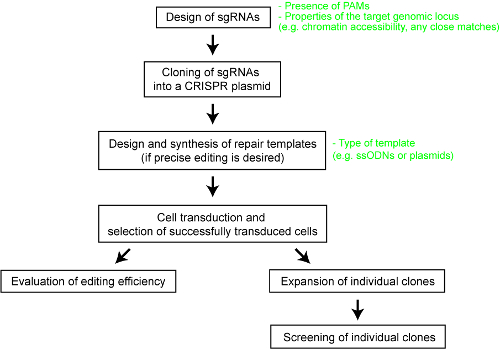
Figura 1 : Una panoramica del flusso di lavoro per generare genoma modificato linee cellulari umane. Clicca qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
Il sistema CRISPR-Cas è una potente, rivoluzionaria tecnologia per progettare i genomi e trascrittomi delle piante e degli animali. Numerose specie batteriche sono state trovate per contenere sistemi CRISPR-Cas, che potenzialmente possono essere adattati per genoma e del trascrittoma fini44di ingegneria. Anche se l’endonucleasi Cas9 da Streptococcus pyogenes (SpCas9) era il primo enzima per essere distribuito con successo in cellule umane21,<sup class="…
Offenlegungen
The authors have nothing to disclose.
Acknowledgements
M.H.T. è supportato da un’agenzia per concessione congiunta Consiglio ufficio di ricerca e di scienza e tecnologia (1431AFG103), un Consiglio nazionale delle ricerche mediche concedere (OFIRG/0017/2016), National Research Foundation concede (NRF2013-THE001-046 e NRF2013-THE001-093), un Concessione del Ministero di educazione Tier 1 (RG50/17 (S)), una startup grant, Nanyang Technological University e fondi per il concorso International Genetically Engineering Machine (iGEM) da Nanyang Technological University.
Materials
| T4 Polynucleotide Kinase (PNK) | NEB | M0201 | |
| Shrimp Alkaline Phosphatase (rSAP) | NEB | M0371 | |
| Tris-Acetate-EDTA (TAE) Buffer, 50X | 1st Base | BUF-3000-50X4L | Dilute to 1X before use. The 1X solution contains 40 mM Tris, 20 mM acetic acid, and 1 mM EDTA. |
| Tris-EDTA (TE) Buffer, 10X | 1st Base | BUF-3020-10X4L | Dilute to 1X before use. The 1X solution contains 10 mM Tris (pH 8.0) and 1 mM EDTA. |
| BbsI | NEB | R0539 | |
| BsmBI | NEB | R0580 | |
| T4 DNA Ligase | NEB | M0202 | 400,000 units/ml |
| Quick Ligation Kit | NEB | M2200 | An alternative to T4 DNA Ligase. |
| Rapid DNA Ligation Kit | Thermo Scientific | K1423 | An alternative to T4 DNA Ligase. |
| Zero Blunt TOPO PCR Cloning Kit | Thermo Scientific | 451245 | The salt solution comes with the TOPO vector in the kit. |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Kit for Gibson assembly. |
| One Shot Stbl3 Chemically Competent E.Coli | Thermo Scientific | C737303 | |
| LB Broth (Lennox), powder | Sigma Aldrich | L3022 | Reconstitute in ddH20, and autoclave before use |
| LB Broth with Agar (Lennox), powder | Sigma Aldrich | L2897 | Reconstitute in ddH20, and autoclave before use |
| SOC media | – | – | 2.5 mM KCl, 10 mM MgCl2, 20 mM glucose in 1 L of LB Broth |
| Ampicillin (Sodium), USP Grade | Gold Biotechnology | A-301 | |
| REDiant 2X PCR Mastermix | 1st Base | BIO-5185 | |
| Agarose | 1st Base | BIO-1000 | |
| T7 Endonuclease I | NEB | M0302 | |
| Plasmid DNA Extraction Miniprep Kit | Favorgen | FAPDE 300 | |
| Dulbecco's Modified Eagle Medium (DMEM), High Glucose | Hyclone | SH30081.01 | 4.5 g/L Glucose, no L-glutamine, HEPES and Sodium Pyruvate |
| L-Glutamine, 200mM | Gibco | 25030 | |
| Penicillin-Streptomycin, 10, 000U/mL | Gibco | 15140 | |
| 0.25% Trypsin-EDTA, 1X | Gibco | 25200 | |
| Fetal Bovine Serum | Hyclone | SV30160 | FBS is heat inactivated before use at 56 oC for 30 min |
| Phosphate Buffered Saline, 1X | Gibco | 20012 | |
| jetPRIME transfection reagent | Polyplus Transfection | 114-75 | |
| QuickExtract DNA Extraction Solution, 1.0 | Epicentre | LUCG-QE09050 | |
| ISOLATE II Genomic DNA Kit | Bioline | BIO-52067 | An alternative to QuickExtract |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491 | |
| Deoxynucleotide (dNTP) Solution Mix | NEB | N0447 | |
| 6X DNA Loading Dye | Thermo Scientific | R0611 | 10 mM Tris-HCl (pH 7.6) 0.03% bromophenol blue, 0.03% xylene cyanol FF, 60% glycerol, 60 mM EDTA |
| Protease Inhibitor Cocktail, Set3 | Merck | 539134 | |
| Nitrocellulose membrane, 0.2µm | Bio-Rad | 1620112 | |
| Tris-glycine-SDS buffer, 10X | Bio-Rad | 1610772 | Dilute to 1X before use. The 1x solution contains 25 mM Tris, 192 mM glycine, and 0.1% SDS. |
| Tris-glycine buffer, 10X | 1st base | BUF-2020 | Dilute to 1X before use. The 1x solution contains 25 mM Tris and 192 mM glycine. |
| Ponceau S solution | Sigma Aldrich | P7170 | |
| TBS, 20X | 1st base | BUF-3030 | Dilute to 1X before use. The 1x solution contains 25 mM Tris-HCl (pH 7.5) and 150 mM NaCl. |
| Tween 20 | Sigma Aldrich | P9416 | |
| Skim Milk for immunoassay | Nacalai Tesque | 31149-75 | |
| WesternBright Sirius-femtogram HRP | Advansta | K12043 | |
| Antibody for β-actin (C4) | Santa Cruz Biotechnology | sc-47778 | Lot number: C0916 |
| MiSeq system | Illumina | SY-410-1003 | |
| NanoDrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit fluorometer | Thermo Scientific | Q33226 | |
| EVOS FL Cell Imaging System | Thermo Scientific | AMF4300 | |
| CRISPR plasmid: pSpCas9(BB)-2A-GFP (PX458) | Addgene | 48138 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: pX601-AAV-CMV::NLS-SaCas9-NLS-3xHA-bGHpA | Addgene | 61591 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: xCas9 3.7 | Addgene | 108379 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: pX330-U6-Chimeric_BB-CBh-hSpCas9 | Addgene | 42230 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: hCas9 | Addgene | 41815 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: eSpCas9(1.1) | Addgene | 71814 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: VP12 (SpCas9-HF1) | Addgene | 72247 | Dual vector system: The gRNA is expressed from a different plasmid. |
Referenzen
- Epinat, J. C., et al. A novel engineered meganuclease induces homologous recombination in yeast and mammalian cells. Nucleic Acids Research. 31 (11), 2952-2962 (2003).
- Arnould, S., et al. Engineered I-CreI derivatives cleaving sequences from the human XPC gene can induce highly efficient gene correction in mammalian cells. Journal of Molecular Biology. 371 (1), 49-65 (2007).
- Chapdelaine, P., Pichavant, C., Rousseau, J., Paques, F., Tremblay, J. P. Meganucleases can restore the reading frame of a mutated dystrophin. Gene Therapy. 17 (7), 846-858 (2010).
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetik. 188 (4), 773-782 (2011).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nature Biotechnology. 29 (2), 143-148 (2011).
- Zhang, F., et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription. Nature Biotechnology. 29 (2), 149-153 (2011).
- Boch, J., et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science. 326 (5959), 1509-1512 (2009).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Nishimasu, H., et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell. 156 (5), 935-949 (2014).
- Yamano, T., et al. Crystal Structure of Cpf1 in Complex with Guide RNA and Target DNA. Cell. 165 (4), 949-962 (2016).
- Swarts, D. C., Mosterd, C., van Passel, M. W., Brouns, S. J. CRISPR interference directs strand specific spacer acquisition. PLoS One. 7 (4), e35888 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Kleinstiver, B. P., et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition. Nature Biotechnology. 33 (12), 1293-1298 (2015).
- Kleinstiver, B. P., et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature. 523 (7561), 481-485 (2015).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Jinek, M., et al. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Cho, S. W., Kim, S., Kim, J. M., Kim, J. S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nature Biotechnology. 31 (3), 230-232 (2013).
- Wang, Y., et al. Systematic evaluation of CRISPR-Cas systems reveals design principles for genome editing in human cells. Genome Biology. 19 (1), 62 (2018).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences U S A. 110 (39), 15644-15649 (2013).
- Kim, E., et al. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni. Nature Communications. 8, 14500 (2017).
- Edraki, A., et al. A Compact, High-Accuracy Cas9 with a Dinucleotide PAM for In Vivo Genome Editing. Molecular Cell. , (2018).
- Chatterjee, P., Jakimo, N., Jacobson, J. M. Minimal PAM specificity of a highly similar SpCas9 ortholog. Science Advances. 4 (10), (2018).
- Muller, M., et al. Streptococcus thermophilus CRISPR-Cas9 Systems Enable Specific Editing of the Human Genome. Mol Therapy. 24 (3), 636-644 (2016).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Boratyn, G. M., et al. BLAST: a more efficient report with usability improvements. Nucleic Acids Research. 41 (Web Server issue), W29-W33 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Montague, T. G., Cruz, J. M., Gagnon, J. A., Church, G. M., Valen, E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research. 42 (Web Server issue), W401-W407 (2014).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 122-123 (2014).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30 (10), 1473-1475 (2014).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Richardson, C. D., Ray, G. J., Bray, N. L., Corn, J. E. Non-homologous DNA increases gene disruption efficiency by altering DNA repair outcomes. Nature Communications. 7, 12463 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biology. 18 (1), 35 (2017).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shmakov, S., et al. Diversity and evolution of class 2 CRISPR-Cas systems. Nature Reviews Microbiology. 15 (3), 169-182 (2017).
- Moreno-Mateos, M. A., et al. CRISPR-Cpf1 mediates efficient homology-directed repair and temperature-controlled genome editing. Nature Communications. 8 (1), 2024 (2017).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Yang, L., et al. Optimization of scarless human stem cell genome editing. Nucleic Acids Research. 41 (19), 9049-9061 (2013).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).

