Analyser la dynamique des protéines en utilisant l'hydrogène change spectrométrie de masse
Summary
conformation des protéines et la dynamique sont essentielles pour comprendre la relation entre la structure et la fonction des protéines. l'échange d'hydrogène couplé à haute résolution spectrométrie de masse est une méthode polyvalente pour étudier la dynamique conformationnelle des protéines ainsi que la caractérisation de la protéine-ligand et des interactions protéine-protéine, y compris les interfaces de contact et des effets allostériques.
Abstract
Tous les processus cellulaires dépendent de la fonctionnalité des protéines. Bien que la fonctionnalité d'une protéine donnée est la conséquence directe de la séquence d'acides aminés unique, il ne se matérialise que par le repliement de la chaîne polypeptidique en une seule disposition tridimensionnelle définie ou plus couramment en un ensemble de conformations interconversion. Enquête sur le lien entre la conformation de la protéine et sa fonction est donc essentiel pour une compréhension complète de la façon dont les protéines sont en mesure de remplir leur grande variété de tâches. Une possibilité d'étudier des changements de conformation d'une protéine subit tout en progressant au travers de son cycle de fonctionnement est de l'hydrogène H-1/2 H-échange, en combinaison avec la spectrométrie de masse à haute résolution (HX-MS). HX-MS est une méthode souple et robuste qui ajoute une nouvelle dimension à l'information structurale obtenue par cristallographie par exemple. Il est utilisé pour étudier le repliement des protéines et le déploiement, la liaison de petit molecule ligands, les interactions protéine-protéine, des changements conformationnels liés à la catalyse enzymatique, et allostery. En outre, HX-MS est souvent utilisé lorsque la quantité de protéine est très limitée ou la cristallisation de la protéine n'est pas réalisable. Ici, nous fournissons un protocole général pour étudier la dynamique des protéines avec HX-MS et décrivons comme un exemple pour révéler l'interface d'interaction des deux protéines dans un complexe.
Introduction
Le nombre de structures cristallines des protéines et complexes de protéines a augmenté rapidement ces dernières années. Ils présentent des instantanés précieux de l'organisation structurelle de ces protéines et fournissent une base pour l'analyse structure-fonction. Cependant, la dynamique des protéines et les changements de conformation, qui sont essentielles pour leurs fonctions, sont rarement révélées par cristallographie aux rayons X. Cryo-microscopie électronique, d'autre part, est capable de capturer des complexes de protéines et des protéines dans des conformations différentes, mais en général ne peut pas résoudre les changements conformationnels jusqu'au niveau de la structure secondaire 1. Dynamique conformationnelle des protéines en solution à des détails atomiques ne peuvent être résolus par RMN, mais cette méthode reste limitée aux protéines de taille relativement petite (généralement ≤ 30 kDa) et les besoins des concentrations élevées de protéines (≥ 100 de pM), qui entrave expériences avec oligomérisation ou d'agrégation des protéines sujettes 2. Une méthode quiest capable de combler entre haute résolution cristallographie aux rayons X et cryo-microscopie électronique, et qui n'est pas limitée par la taille de la protéine ou de la concentration de l'hydrogène est l'amide-1 H / 2 H-échange (HX) en combinaison avec la spectrométrie de masse (MS). Ces dernières années, ce procédé a mis au point un outil d'analyse utile pour l'analyse de la dynamique des protéines, le repliement des protéines, la stabilité des protéines et des changements de conformation 3-5. La base moléculaire de cette méthode est la nature labile du squelette hydrogènes d'amide dans les protéines, qui vont échanger avec des atomes de deutérium lorsque la protéine est placé dans une solution de D 2 O. L'augmentation subséquente de la masse de la protéine au cours du temps est mesurée avec MS à haute résolution.
En bref peptides non structurés ne HX dépend de la température, la concentration en catalyseur (OH -, H 3 O + ie pH, voir la figure 3) et des chaînes latérales d'acides aminés des résidus adjacents dues à inductive, chateffets alytic et stériques. Ces effets sur la valeur intrinsèque du taux de change chimique k ch ont été élégamment quantifiée par Bai et al. 6 et un programme est disponible (avec la permission Z. Zhang), qui calcule k ch pour chaque acide aminé dans un polypeptide dépend du pH et de la température. A pH neutre, et des températures ambiantes ch k est de l'ordre de 10 1 à 10 3 s -1. Dans les protéines pliées HX peut être 2-9 ordres de grandeur plus lentes principalement due à la liaison hydrogène dans la structure secondaire et à un degré moindre en raison de l'accès restreint de hydratés ions OH – à l'intérieur d'une protéine étroitement replié. HX en protéines natives implique donc déroulement, l'échange chimique partielle ou globale et repliement à l'état natif selon l'équation (1) et les taux de change observés k obs dépendent du taux d'ouverture k op, les taux de clôture k cl et l'échange chimique intrinsèque rate ch k selon l'équation (2).
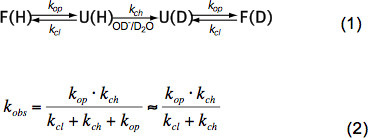
Dans des conditions natives de l'Etat k op est beaucoup plus petit que k ch et peut être négligée dans le dénominateur. Il existe deux régimes de change extrêmes appelés EX1 et EX2. Si la cl k est beaucoup plus petit que k ch (EX1) du taux observé est pratiquement égale à la vitesse d'ouverture et de HX permet l'observation immédiate du déploiement d'un élément de structure. Un tel régime de change, où tout protons amide échange à la fois lors de l'ouverture de l'élément structurel, est facilement observable dans MS par une distribution bimodale des pics isotopiques 7. Si k cl est beaucoup plus grand que k ch (EX2) du taux observé est proportionnel à k ch lequel la constante de proportionnalité est égal aux équilibres de pliage-dépliage constante K = k u op </sub> / K cl. Dans ces conditions, beaucoup d'ouverture et de clôture des événements sont nécessaires avant tout échange de protons amide pour deutons, conduisant à une augmentation graduelle de la masse moyenne, tandis que la distribution isotopique reste à peu près le même. Le régime de EX2 permet la détermination de l'énergie libre de dépliement Ag u et donc la stabilité d'un élément de structure. Sous la condition native de l'Etat du régime de EX2 est la plus courante. Augmentation du pH et l'ajout d'agents chaotropiques peut changer le mécanisme de change pour EX1. Par conséquent, HX-MS peut être utilisée pour explorer thermodynamique ainsi que des paramètres cinétiques de repliement des protéines et des changements conformationnels.
Comme mentionné ci-dessus HX est intrinsèquement dépendante du pH et de la température et de l'échange de demi-vie d'un proton complètement exposé au solvant du groupe amide du squelette est comprise entre 5 à 400 msec à pH physiologique (pH 7,6) et 30 ° C, mais à 10 min> 15 h avec une moyenne de> 2 h à pH 2,9 et à 0 °C (sauf pour le proton de la première épine dorsale liaison amide d'un polypeptide, qui échange avec une demi-vie de ca. 1-2 min). Dans ces conditions, l'échange lent, il est possible de digérer l'échantillon en utilisant des protéases (par exemple la pepsine) qui sont actives dans ces conditions, avec les perdre toutes les informations contenues dans les deutons incorporés. Depuis l'introduction de la digestion gastro-duodénal dans des conditions échanger lents, non seulement la cinétique de HX ensemble de protéines de pleine longueur peuvent être analysés mais HX peuvent être localisés à des régions spécifiques 8,9. La résolution spatiale est limitée à la taille des fragments peptiques générés, ce qui est en général comprise entre 10-30 résidus. Cependant, les fragments chevauchants créés en raison de la nature non spécifique de clivage par la pepsine pourraient conduire à une augmentation de la résolution spatiale. En outre, plusieurs autres proteases se sont révélés être actifs dans des conditions de trempe, cependant, beaucoup moins efficaces que la pepsine 10. En outre augsoi de la résolution spatiale peut être atteint par la fragmentation de peptides en phase de gaz par des méthodes qui ont préservé le modèle de deutération comme la capture d'électrons dissociation (DPE), transfert d'électrons dissociation (ETD) et infrarouge dissociation multiphotonique (IRMPD) 11-13. Ces techniques empêchent la perte de résolution spatiale due à la migration intramoléculaire de proton ("brouillage"), qui est observée par dissociation induite par collision (CID) de la technique de fragmentation le plus couramment utilisé. Cependant, ces méthodes nécessitent l'optimisation pour chaque peptide individuel et est donc encore assez difficile.
HX-MS a été utilisé pour analyser les interactions protéine-ligand et la protéine-protéine, y compris l'assemblage de la capside virale 14-17. Protéines de repliage et dépliage ainsi que la température des changements conformationnels induits ont été étudiés 7,18,19. Phosphorylation et unique de conformation liée à la mutation d'acides aminés et modifie 16,20 nucleotchangements ide-induits ont été analysés 21,22. Par conséquent, cette méthode semble parfaitement adapté pour analyser l'assemblage et la dynamique des machines moléculaires. Un candidat intéressant, dont le mécanisme est d'un grand intérêt général, est le complexe chaperon Hsp90.
Protocol
Representative Results
Discussion
Liaison d'un partenaire d'interaction d'une protéine entraîne inévitablement des changements dans l'accessibilité au solvant sur le site de liaison. En outre, de nombreuses protéines subissent des changements conformationnels dynamiques lors de la liaison, qui touchent d'autres régions que l'interface de liaison réelle. HX-MS est une méthode robuste pour surveiller ces changements et est même capable de révéler des changements conformationnels de protéines sur des échelles de temps q…
Divulgaciones
The authors have nothing to disclose.
Acknowledgements
Nous remercions M. Boysen des commentaires sur le manuscrit. Ce projet a été financé par la Deutsche Forschungsgemeinschaft (SFB638 et MA 1278/4-1 à MPM, et Cluster d'excellence: CellNetworks EXC 81/1). MPM est enquêteur du Pôle d'excellence: CellNetworks.
Materials
| Reagent/Equipment | Company | Catalogue number | Comments (optional) |
| maXis QTOF | Bruker | ||
| nanoAcquity UPLC | Waters Corp. | ||
| Shimadzu 10AD-VP | Shimadzu | ||
| 6-port Valve EPC6W with microelectric actuator | Valco | #EPC6W | |
| Injection valve (manual) | Rheodyne | #7725 | |
| Poros AL20 media | Applied Biosystems | #1-6029-06 | |
| Poros R2 | Applied Biosystems | #1-1118-02 | |
| Pepsin | Sigma | #P6887 | use fresh pepsin |
| Microbore (1 mm) | IDEX | #C-128 | |
| Microbore (2 mm) | IDEX | #C-130B | |
| Acquity UPLC BEH C8 Column | Waters Corp. | #186002876 | |
| Thermomixer | Eppendorf | #5355000.011 | |
| Tubing (various diameters) | IDEX | ||
| Fittings | IDEX | #PK-110 with PK-100 |
Referencias
- Saibil, H. R. Conformational changes studied by cryo-electron microscopy. Nat. Struct. Biol. 7 (9), 711-714 (2000).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Hoofnagle, A. N., Resing, K. A., Ahn, N. G. Protein analysis by hydrogen exchange mass spectrometry. Annu. Rev. Biophys. Biomol. Struct. 32, 1-25 (2003).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass. Spectrom. Rev. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. -. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chem Soc. Rev. 40 (3), 1224-1210 (2011).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Rist, W., Jørgensen, T. J. D., Roepstorff, P., Bukau, B., Mayer, M. P. Mapping temperature-induced conformational changes in the Escherichia coli heat shock transcription factor sigma 32 by amide hydrogen exchange. The Journal of biological chemistry. 278 (51), 51415-51421 (1074).
- Englander, J. J., Rogero, J. R., Englander, S. W. Protein hydrogen exchange studied by the fragment separation method. Anal Biochem. 147 (1), 234-244 (1985).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Cravello, L., Lascoux, D., Forest, E. Use of different proteases working in acidic conditions to improve sequence coverage and resolution in hydrogen/deuterium exchange of large proteins. Rapid Commun. Mass Spectrom. : RCM. 17 (21), 2387-2393 (2003).
- Rand, K. D., Zehl, M., Jensen, O. N., Jørgensen, T. J. D. Protein hydrogen exchange measured at single-residue resolution by electron transfer dissociation mass spectrometry. Anal chem. 81 (14), 5577-5584 (2009).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Electron capture dissociation of electrosprayed protein ions for spatially resolved hydrogen exchange measurements. J. Am. Chem. Soc. 130 (35), 11574-11575 (2008).
- Yamada, N., Suzuki, E. -. I., Hirayama, K. Identification of the interface of a large protein-protein complex using H/D exchange and Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 16 (4), 293-299 (2002).
- Lee, T., Hoofnagle, A. N., et al. Docking motif interactions in MAP kinases revealed by hydrogen exchange mass spectrometry. Mol. Cell. 14 (1), 43-55 (2004).
- Hasan, A., Smith, D. L., Smith, J. B. Alpha-crystallin regions affected by adenosine 5′-triphosphate identified by hydrogen-deuterium exchange. Biochem. 41 (52), 15876-15882 (2002).
- Lanman, J., Lam, T. T., Emmett, M. R., Marshall, A. G., Sakalian, M., Prevelige, P. E. Key interactions in HIV-1 maturation identified by hydrogen-deuterium exchange. Nat. Struct. Mol. Biol. 11 (7), 676-677 (2004).
- Wang, L., Lane, L. C., Smith, D. L. Detecting structural changes in viral capsids by hydrogen exchange and mass spectrometry. Protein Sci. 10 (6), 1234-1243 (2001).
- Pan, H., Raza, A. S., Smith, D. L. Equilibrium and kinetic folding of rabbit muscle triosephosphate isomerase by hydrogen exchange mass spectrometry. J. Mol. Biol. 336 (5), 1251-1263 (2004).
- Mazon, H., Marcillat, O., Forest, E., Smith, D. L., Vial, C. Conformational dynamics of the GdmHCl-induced molten globule state of creatine kinase monitored by hydrogen exchange and mass spectrometry. Biochem. 43 (17), 5045-5054 (2004).
- Lanman, J., Lam, T. T., et al. Identification of novel interactions in HIV-1 capsid protein assembly by high-resolution mass spectrometry. J. Mol. Biol. 325 (4), 759-772 (2003).
- Rist, W., Graf, C., Bukau, B., Mayer, M. P. Amide hydrogen exchange reveals conformational changes in hsp70 chaperones important for allosteric regulation. J. Biol. chem. 281 (24), 16493-16501 (2006).
- Graf, C., Stankiewicz, M., Kramer, G., Mayer, M. P. Spatially and kinetically resolved changes in the conformational dynamics of the Hsp90 chaperone machine. EMBO J. 28 (5), 602-613 (2009).
- Lee, C. -. T., Graf, C., Mayer, F. J., Richter, S. M., Mayer, M. P. Dynamics of the regulation of Hsp90 by the co-chaperone Sti1. EMBO J. 31 (6), 1518-1528 (2012).
- Lou, X., Kirchner, M., et al. Deuteration distribution estimation with improved sequence coverage for HX/MS experiments. Bioinformatics. 26 (12), 1535-1541 (2010).
- Kreshuk, A., Stankiewicz, M., Lou, X., Kirchner, M., Hamprecht, F. A., Mayer, M. P. Automated detection and analysis of bimodal isotope peak distributions in H/D exchange mass spectrometry using HeXicon. Intl. J. mass spectrom. 302 (1-3), 125-131 (2011).
- Walters, B. T., Ricciuti, A., Mayne, L., Englander, S. W. Minimizing back exchange in the hydrogen exchange-mass spectrometry experiment. J. Am. Soc. Mass Spectrom. 23 (12), 2132-2139 (2012).
- Weis, D. D., Wales, T. E., Engen, J. R., Hotchko, M., Ten Eyck, L. F. Identification and characterization of EX1 kinetics in H/D exchange mass spectrometry by peak width analysis. J. Am. Soc. Mass Spectrom. 17 (11), 1498-1509 (2006).
- Fang, J., Rand, K. D., Beuning, P. J., Engen, J. R. False EX1 signatures caused by sample carryover during HX MS analyses. Intl. J. Mass Spectrom. 302 (1-3), 19-25 (2011).
- Deng, Y., Smith, D. L. Identification of unfolding domains in large proteins by their unfolding rates. Biochem. 37 (18), 6256-6262 (1998).
- Wales, T. E., Engen, J. R. Partial unfolding of diverse SH3 domains on a wide timescale. J. Mol. Biol. 357 (5), 1592-1604 (2006).
- Pan, Y., Piyadasa, H., O’Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by h/d exchange and covalent labeling: the glycerol facilitator. J. Mol. Biol. 416 (3), 400-413 (2012).
- Hebling, C. M., Morgan, C. R., Stafford, D. W., Jorgenson, J. W., Rand, K. D., Engen, J. R. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Anal chem. 82 (13), 5415-5419 (2010).
- Abzalimov, R. R., Kaplan, D. A., Easterling, M. L., Kaltashov, I. A. Protein conformations can be probed in top-down HDX MS experiments utilizing electron transfer dissociation of protein ions without hydrogen scrambling. J. Am. Soc. Mass Spectrom. 20 (8), 1514-1517 (2009).

