Analisando Dynamics proteína utilizando hidrogênio câmbio Espectrometria de Massa
Summary
Conformação da proteína e dinâmicas são a chave para a compreensão da relação entre estrutura e função de proteínas. Troca de hidrogênio acoplada a espectrometria de massa de alta resolução é um método versátil para estudar a dinâmica de conformação das proteínas, bem como a caracterização da proteína-ligante e interações proteína-proteína, incluindo interfaces de contato e efeitos alostéricos.
Abstract
Todos os processos celulares dependem da funcionalidade das proteínas. Embora a funcionalidade de uma dada proteína é a consequência directa da sua sequência de aminoácidos única, apenas é realizado pela dobragem da cadeia polipeptídica em um único arranjo tridimensional definido ou mais vulgarmente em um conjunto de conformações interconversão. Investigando a ligação entre a conformação da proteína e sua função é, portanto, essencial para um entendimento completo de como as proteínas são capazes de cumprir a sua grande variedade de tarefas. Uma possibilidade para estudar mudanças de conformação de uma proteína sofre, progredindo através do seu ciclo funcional é hidrogénio-1 H / 2 H-troca em combinação com espectrometria de massa de alta resolução (HX-MS). HX-MS é um método versátil e robusto que acrescenta uma nova dimensão às informações estruturais obtidas por cristalografia por exemplo. Ele é usado para estudar a proteína dobrando e desdobrando, a ligação da pequena molligantes ecule, interações proteína-proteína, mudanças conformacionais ligadas à catálise enzimática e allostery. Além disso, HX-MS é muitas vezes utilizado quando a quantidade de proteína é muito limitada, ou a cristalização da proteína não é viável. Aqui proporciona-se um protocolo geral para o estudo da dinâmica das proteínas com HX-MS e descrevem como um exemplo de como a interface para revelar a interacção de duas proteínas de um complexo.
Introduction
O número de estruturas de cristal de proteínas e complexos de proteína aumentou rapidamente nos últimos anos. Apresentam instantâneos inestimáveis da organização estrutural das proteínas e fornecer uma base para a análise da estrutura-função. No entanto, a dinâmica das proteínas e as mudanças conformacionais, que são essenciais para as suas funções, são raramente revelado por cristalografia de raios-X. Cryo-electronmicroscopy, por outro lado, é capaz de capturar os complexos de proteínas e de proteínas em diferentes conformações, mas geralmente não é possível resolver as alterações conformacionais para baixo para o nível de uma estrutura secundária. Dinâmica de conformação de proteínas em solução em detalhes atómicas só pode ser resolvido por RMN, mas este método é ainda limitado a proteínas de tamanhos relativamente pequenos (geralmente ≤ 30 kDa) e necessita de altas concentrações de proteínas (≥ 100 pM), o que dificulta as experiências com oligomerização ou agregação de proteínas propensas 2. Um método queé capaz de ponte entre a alta resolução de cristalografia de raios-X e crio-electronmicroscopy e que não é limitado pelo tamanho ou a concentração de proteína é de hidrogénio amida-1 H / 2 H-troca (HX) em combinação com espectrometria de massa (MS). Nos últimos anos, este método tem desenvolvido para um instrumento analítico valioso para a análise da dinâmica das proteínas, o dobramento de proteínas, a estabilidade da proteína e alterações conformacionais 3-5. A base molecular deste método é a natureza lábil do backbone hidrogénios de amida em proteínas, que irá trocar com átomos de deutério, quando a proteína é colocada em um D 2 O solução. O consequente aumento da massa de proteína ao longo do tempo é medido com alta resolução MS.
Em péptidos não estruturados curtas só HX depende da temperatura, da concentração do catalisador (OH -, H 3 O + por exemplo pH, ver Figura 3) e as cadeias laterais de resíduos de aminoácidos adjacentes, devido à indutivo, gatoefeitos alytic e estéricos. Estes efeitos sobre o ch intrínseca taxa de permuta química k foram elegantemente quantificada por Bai et al. 6 e um programa está disponível (por cortesia Z. Zhang), que calcula k ch para cada aminoácido num polipéptido dependente do pH e da temperatura. Em pH neutro e temperatura ambiente k ch é da ordem de 10 -10 1 3 seg -1. Em proteínas dobradas HX pode ser 2-9 ordens de grandeza mais lento, principalmente devido a pontes de hidrogénio na estrutura secundária e a um grau menor devido ao acesso limitado de hidratadas OH – iões para o interior de uma proteína fortemente dobrado. HX em proteínas nativas, portanto, implica desdobramento, por permuta química parcial ou global e redobrando para o estado nativo de acordo com a equação (1) e as taxas de câmbio observadas k obs dependem da taxa de abertura op k, os cl k taxa de fechamento ea troca química intrínseca rate k ch de acordo com a equação (2).
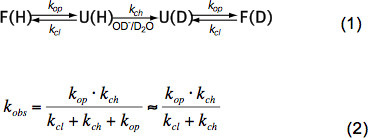
Sob condições de estado nativo k op é muito menor do k ch e pode ser negligenciada no denominador. Existem dois regimes cambiais extremos chamados EX1 e EX2. Se o cl k é muito menor do k ch (EX1) a taxa observada é praticamente igual à taxa de abertura e HX permite a observação imediata do desdobramento de um elemento estrutural. Tal regime cambial, onde todos os prótons amida troca de uma só vez aquando da abertura do elemento estrutural, é facilmente observáveis em MS por uma distribuição bimodal dos picos de isótopos 7. Se k cl é muito maior do que k ch (EX2) a taxa observada é proporcional à k ch em que a constante de proporcionalidade é igual aos equilíbrios-desdobramento dobráveis constante op K u = k </sub> K / cl. Sob estas condições, muitos eventos de abertura e fecho são necessárias antes de todos os protões amida troca de deutério, que conduz a um aumento gradual da massa média, enquanto a distribuição isotópica permanece praticamente a mesma. O regime EX2 permite a determinação da energia livre de desdobramento ÄG u e, por conseguinte, a estabilidade de um elemento estrutural. Sob condição de estado nativo do regime EX2 é mais comum. Aumento do pH e da adição de agentes caotrópicos pode mudar o mecanismo de troca de EX1. Portanto, HX-MS pode ser utilizada para explorar termodinâmico, bem como os parâmetros cinéticos de dobragem de proteína e alterações conformacionais.
Como mencionado acima HX é intrinsecamente pH e dependente da temperatura e meia-vida a troca de um protão exposta completamente solvente do grupo amida espinha dorsal é entre 5-400 ms a um pH fisiológico (pH 7,6) e 30 ° C, mas 10 min a> 15 horas, com uma média de> 2 horas a pH 2,9 e 0 °C (excepto para o protão da primeira estrutura de ligação amida de um polipeptídeo, que troca com uma meia-vida de aprox. 1-2 min). Sob tais condições lenta troca é possível para digerir a amostra usando proteases (por exemplo pepsina) que atuam nessas condições, sem perder todas as informações contidas nos dêuterons incorporadas. Desde a introdução de digestão péptica sob condições troca lenta, não apenas da cinética geral HX de proteínas de comprimento completo pode ser analisado, mas HX pode ser localizado em regiões específicas 8,9. A resolução espacial está limitado ao tamanho dos fragmentos gerados pépticas, que é, em geral, entre 10-30 resíduos. No entanto, fragmentos sobrepostos criados devido à natureza não específica da clivagem por pepsina pode levar a um aumento da resolução espacial. Além disso, várias outras proteases foram encontrados para ser activo em condições de têmpera, contudo, muito menos eficiente do que a pepsina 10. Além disso aumense de resolução espacial pode ser alcançado pela fragmentação de peptídeos em fase gasosa por meio de métodos que preservaram o padrão deuteração tais como a captura eletrônica de dissociação (ECD), transferência eletrônica de dissociação (ETD) e dissociação multiphoton infravermelho (IRMPD) 11-13. Estas técnicas de evitar a perda de resolução espacial devido à migração intramolecular de protões ("scrambling"), que é observada por dissociação induzida por colisão (CID) a técnica de fragmentação mais utilizada. No entanto, estes métodos requerem otimização para cada peptídeo individual e é, portanto, ainda bastante desafiador.
HX-MS foi utilizada para analisar interacções proteína-ligando e de proteína-proteína, incluindo a montagem da cápside viral 14-17. Proteína desdobramento e redobrando assim como temperatura provocadas mudanças conformacionais foram investigados 7,18,19. Fosforilação e conformacional relacionada com mutação único aminoácido muda 16,20 e nucleotmudanças ide-induzidas foram analisados 21,22. Portanto, esse método parece idealmente adequado para analisar a montagem e dinâmica de máquinas moleculares. Um candidato atraente, cujo mecanismo é de grande interesse geral, é o complexo chaperone Hsp90.
Protocol
Representative Results
Discussion
A ligação de um parceiro de interacção com uma proteína, inevitavelmente, provoca alterações na acessibilidade do solvente sobre o local de ligação. Além disso, muitas proteínas sofrem alterações conformacionais dinâmicas sobre a ligação, que afetam outras regiões do que a interface de união real. HX-MS é um método robusto para monitorar essas mudanças e ainda é capaz de revelar mudanças conformacionais em proteínas em escalas de tempo que outros métodos não pode cobrir.
<p class="jove_cont…Divulgaciones
The authors have nothing to disclose.
Acknowledgements
Agradecemos M. Boysen para comentários sobre o manuscrito. Este projecto foi financiado pelo Deutsche Forschungsgemeinschaft (SFB638 e MA 1278/4-1 a MPM, e Cluster de Excelência: CellNetworks EXC 81/1). MPM é investigador do Cluster de Excelência: CellNetworks.
Materials
| Reagent/Equipment | Company | Catalogue number | Comments (optional) |
| maXis QTOF | Bruker | ||
| nanoAcquity UPLC | Waters Corp. | ||
| Shimadzu 10AD-VP | Shimadzu | ||
| 6-port Valve EPC6W with microelectric actuator | Valco | #EPC6W | |
| Injection valve (manual) | Rheodyne | #7725 | |
| Poros AL20 media | Applied Biosystems | #1-6029-06 | |
| Poros R2 | Applied Biosystems | #1-1118-02 | |
| Pepsin | Sigma | #P6887 | use fresh pepsin |
| Microbore (1 mm) | IDEX | #C-128 | |
| Microbore (2 mm) | IDEX | #C-130B | |
| Acquity UPLC BEH C8 Column | Waters Corp. | #186002876 | |
| Thermomixer | Eppendorf | #5355000.011 | |
| Tubing (various diameters) | IDEX | ||
| Fittings | IDEX | #PK-110 with PK-100 |
Referencias
- Saibil, H. R. Conformational changes studied by cryo-electron microscopy. Nat. Struct. Biol. 7 (9), 711-714 (2000).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Hoofnagle, A. N., Resing, K. A., Ahn, N. G. Protein analysis by hydrogen exchange mass spectrometry. Annu. Rev. Biophys. Biomol. Struct. 32, 1-25 (2003).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass. Spectrom. Rev. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. -. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chem Soc. Rev. 40 (3), 1224-1210 (2011).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Rist, W., Jørgensen, T. J. D., Roepstorff, P., Bukau, B., Mayer, M. P. Mapping temperature-induced conformational changes in the Escherichia coli heat shock transcription factor sigma 32 by amide hydrogen exchange. The Journal of biological chemistry. 278 (51), 51415-51421 (1074).
- Englander, J. J., Rogero, J. R., Englander, S. W. Protein hydrogen exchange studied by the fragment separation method. Anal Biochem. 147 (1), 234-244 (1985).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Cravello, L., Lascoux, D., Forest, E. Use of different proteases working in acidic conditions to improve sequence coverage and resolution in hydrogen/deuterium exchange of large proteins. Rapid Commun. Mass Spectrom. : RCM. 17 (21), 2387-2393 (2003).
- Rand, K. D., Zehl, M., Jensen, O. N., Jørgensen, T. J. D. Protein hydrogen exchange measured at single-residue resolution by electron transfer dissociation mass spectrometry. Anal chem. 81 (14), 5577-5584 (2009).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Electron capture dissociation of electrosprayed protein ions for spatially resolved hydrogen exchange measurements. J. Am. Chem. Soc. 130 (35), 11574-11575 (2008).
- Yamada, N., Suzuki, E. -. I., Hirayama, K. Identification of the interface of a large protein-protein complex using H/D exchange and Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 16 (4), 293-299 (2002).
- Lee, T., Hoofnagle, A. N., et al. Docking motif interactions in MAP kinases revealed by hydrogen exchange mass spectrometry. Mol. Cell. 14 (1), 43-55 (2004).
- Hasan, A., Smith, D. L., Smith, J. B. Alpha-crystallin regions affected by adenosine 5′-triphosphate identified by hydrogen-deuterium exchange. Biochem. 41 (52), 15876-15882 (2002).
- Lanman, J., Lam, T. T., Emmett, M. R., Marshall, A. G., Sakalian, M., Prevelige, P. E. Key interactions in HIV-1 maturation identified by hydrogen-deuterium exchange. Nat. Struct. Mol. Biol. 11 (7), 676-677 (2004).
- Wang, L., Lane, L. C., Smith, D. L. Detecting structural changes in viral capsids by hydrogen exchange and mass spectrometry. Protein Sci. 10 (6), 1234-1243 (2001).
- Pan, H., Raza, A. S., Smith, D. L. Equilibrium and kinetic folding of rabbit muscle triosephosphate isomerase by hydrogen exchange mass spectrometry. J. Mol. Biol. 336 (5), 1251-1263 (2004).
- Mazon, H., Marcillat, O., Forest, E., Smith, D. L., Vial, C. Conformational dynamics of the GdmHCl-induced molten globule state of creatine kinase monitored by hydrogen exchange and mass spectrometry. Biochem. 43 (17), 5045-5054 (2004).
- Lanman, J., Lam, T. T., et al. Identification of novel interactions in HIV-1 capsid protein assembly by high-resolution mass spectrometry. J. Mol. Biol. 325 (4), 759-772 (2003).
- Rist, W., Graf, C., Bukau, B., Mayer, M. P. Amide hydrogen exchange reveals conformational changes in hsp70 chaperones important for allosteric regulation. J. Biol. chem. 281 (24), 16493-16501 (2006).
- Graf, C., Stankiewicz, M., Kramer, G., Mayer, M. P. Spatially and kinetically resolved changes in the conformational dynamics of the Hsp90 chaperone machine. EMBO J. 28 (5), 602-613 (2009).
- Lee, C. -. T., Graf, C., Mayer, F. J., Richter, S. M., Mayer, M. P. Dynamics of the regulation of Hsp90 by the co-chaperone Sti1. EMBO J. 31 (6), 1518-1528 (2012).
- Lou, X., Kirchner, M., et al. Deuteration distribution estimation with improved sequence coverage for HX/MS experiments. Bioinformatics. 26 (12), 1535-1541 (2010).
- Kreshuk, A., Stankiewicz, M., Lou, X., Kirchner, M., Hamprecht, F. A., Mayer, M. P. Automated detection and analysis of bimodal isotope peak distributions in H/D exchange mass spectrometry using HeXicon. Intl. J. mass spectrom. 302 (1-3), 125-131 (2011).
- Walters, B. T., Ricciuti, A., Mayne, L., Englander, S. W. Minimizing back exchange in the hydrogen exchange-mass spectrometry experiment. J. Am. Soc. Mass Spectrom. 23 (12), 2132-2139 (2012).
- Weis, D. D., Wales, T. E., Engen, J. R., Hotchko, M., Ten Eyck, L. F. Identification and characterization of EX1 kinetics in H/D exchange mass spectrometry by peak width analysis. J. Am. Soc. Mass Spectrom. 17 (11), 1498-1509 (2006).
- Fang, J., Rand, K. D., Beuning, P. J., Engen, J. R. False EX1 signatures caused by sample carryover during HX MS analyses. Intl. J. Mass Spectrom. 302 (1-3), 19-25 (2011).
- Deng, Y., Smith, D. L. Identification of unfolding domains in large proteins by their unfolding rates. Biochem. 37 (18), 6256-6262 (1998).
- Wales, T. E., Engen, J. R. Partial unfolding of diverse SH3 domains on a wide timescale. J. Mol. Biol. 357 (5), 1592-1604 (2006).
- Pan, Y., Piyadasa, H., O’Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by h/d exchange and covalent labeling: the glycerol facilitator. J. Mol. Biol. 416 (3), 400-413 (2012).
- Hebling, C. M., Morgan, C. R., Stafford, D. W., Jorgenson, J. W., Rand, K. D., Engen, J. R. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Anal chem. 82 (13), 5415-5419 (2010).
- Abzalimov, R. R., Kaplan, D. A., Easterling, M. L., Kaltashov, I. A. Protein conformations can be probed in top-down HDX MS experiments utilizing electron transfer dissociation of protein ions without hydrogen scrambling. J. Am. Soc. Mass Spectrom. 20 (8), 1514-1517 (2009).

