Hidrojen Değişim Kütle Spektrometre Kullanarak Protein Dinamiği Analizi
Summary
Protein yapı ve dinamikleri protein yapı ve fonksiyon arasındaki ilişkiyi anlamak için anahtardır. Yüksek çözünürlüklü kütle spektrometrisi ile birlikte hidrojen alış veriş proteinlerin konformasyonel dinamiklerini incelemek ve temas arayüzler ve allosterik etkileri de dahil olmak üzere protein-ligand ve protein-protein etkileşimleri, karakterize için çok yönlü bir yöntemdir.
Abstract
Bütün hücresel süreçleri proteinlerin işlevselliğine bağlıdır. Verilen bir proteinin işlevselliğini eşsiz amino asit dizisinin doğrudan bir sonucu olmasına rağmen, bu sadece bir tek tanımlanmış üç boyutlu bir düzenleme ya da daha yaygın da birbirine konformasyonlarının bir topluluk olarak içine polipeptid zincirinin katlanması ile gerçekleştirilir. Protein yapısında ve fonksiyonunda arasındaki bağlantıyı araştıran proteinlerin görevleri onların büyük çeşitliliği yerine edebiliyoruz nasıl anlaşılması için elzemdir. Fonksiyonel döngüsü boyunca ilerlerken, bir protein maruz yapısal değişiklikleri incelemek için bir olanak, hidrojen-1 yüksek çözünürlüklü kütle spektrometrisi (HX-MS) ile kombinasyon halinde H / 2, H-değişimidir. HX-MS örneğin kristalografisiyle elde edilen yapısal bilgilere yeni bir boyut ekler çok yönlü ve sağlam bir yöntemdir. Bu küçük mol bağlayıcı, katlama ve açılımı proteini incelemek için kullanılırecule ligandlar, protein-protein etkileşimleri, enzim katalizi ile bağlantılı konformasyonel değişiklikleri ve allostery. Protein miktarı çok sınırlıdır ya da proteinin kristalizasyon mümkün değildir Buna ek olarak, HX-MS sıklıkla kullanılmaktadır. Burada HX-MS ile protein dinamiğini çalışmak için genel bir protokol sağlar ve nasıl bir kompleks içinde iki proteinin etkileşim arayüz ortaya çıkarmak için bir örnek olarak tarif eder.
Introduction
Proteinler ve protein komplekslerinin kristal yapılarının sayısı son yıllarda hızla artmıştır. Bunlar, bu proteinlerin yapısal organizasyon çok değerli anlık mevcut ve yapı-fonksiyon analizi için bir temel sağlar. Ancak, proteinlerin ve bunların fonksiyonları için gerekli olan yapısal değişiklikler, dinamikleri, nadiren X-ışını kristalografisi ile ortaya çıkar. Cryo-elektronmikroskop, diğer taraftan, farklı yapıda protein ve protein kompleksleri yakalamak mümkün ama genellikle ikincil yapı seviye 1 şekilsel değişiklikleri aşağı çözemez. Atomik ayrıntıları, çözelti içinde proteinlerin konformasyonel dinamiği sadece NMR ile çözülebilir, ancak bu yöntem halen göreceli olarak küçük boyutlarda (genellikle ≤ 30 kDa) proteinlere sınırlı ile deneyler engellemektedir proteinlerin yüksek konsantrasyonlarda (≥ 100 uM), ihtiyacı var oligomerizasyon ya da toplama eğilimli proteinler 2. Bir yöntem, kiyüksek çözünürlüklü X-ışını kristalografisi ve kriyo-elektronmikroskop arasında olan ve köprü edebilmektedir protein boyut veya konsantrasyonu ile sınırlı değildir amid hidrojen-1 kütle spektrometrisi (MS) ile bir arada H / 2 H-değişim (HX) 'dir. Son yıllarda bu yöntem, protein dinamiği, protein katlanmasına, protein stabilitesi ve bu şekilsel değişikliklere 3-5 analizi için değerli bir analitik araç geliştirmiştir. Bu yöntemin moleküler temeli protein D 2 O çözeltisi yerleştirildiği zaman, döteryum atomları ile yer değiştirmeye doyurmaya proteinlerde omurga amid hidrojenlerin arasında kararsız doğasıdır. , Zaman içinde protein kütlesindeki artış, daha sonra yüksek çözünürlüklü MS ile ölçülür.
Kısa peptitler yapısal olmayan sadece sıcaklık, katalizör konsantrasyonuna bağlı HX (OH – Şekil 3, 3 H + O, yani pH) ve endüktif, kedi nedeniyle komşu artıkların amino asit yan zincirlerialytic ve sterik etkiler. Iç kimyasal kuru k kanal üzerindeki bu etkiler zarif Bai et al. 6 ile nicelleştirilmiştir edilmiş ve bir program pH ve sıcaklığa bağlı olarak bir polipeptid içinde her bir amino asit için k kanal hesaplar (izniyle Z. Zhang) mevcuttur. Nötr pH ve ortam sıcaklıklarında k ch 10 1 -10 saniye 3 -1 sırasına göre olan. Sıkı bir şekilde katlanmış proteinin iç iyonları kapandı – proteinlerinde HX temel olarak ikincil yapı içinde hidrojen bağı ve OH sulu sınırlı erişimi nedeniyle düşük derecede daha düşük büyüklükte 2-9 emir olabilir. Doğal protein HX bu nedenle kısmen ya da küresel açılımı, kimyasal değişimi ve denkleme göre yerel durumuna yeniden katlama etkisi altına (1) ve k obs açılış hızı k op, kapatma hızı k cl ve içsel kimyasal değişimi bağlıdır gözlenen kur rate k ch denkleme göre (2).
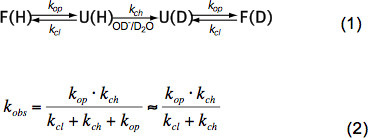
Yerli hal koşullarında k op k ch çok daha küçüktür ve paydada ihmal edilebilir. EX1 ve EX2 denilen iki uç döviz rejimleri vardır. K cl k kanal (EX1) daha küçük ise, gözlenen oranı açma hızına hemen hemen eşit olduğunu ve HX, bir yapı elemanının açılımı hemen gözlem sağlar. , Yapısal elemanın açılması üzerine bir kerede amid proton değişimi, izotop tepe 7 bir bimodal dağılım ile MS kolayca gözlemlenebilir böyle bir değişim rejimi,. K cl k ch çok daha büyük olduğu takdirde orantı sabit katlama açılımı dengesinin eşittir burada, (EX2) gözlenen oranı k ch ile orantılıdır sabiti K u = k op </sub> / K cl. Izotopik dağılımı yaklaşık olarak aynı kalırken, bu koşullar altında, bir çok açılış ve kapanış etkinlik ortalama kütlesi tedrici bir artışa yol açan, döteronların seyahati amid proton değişim önce gereklidir. EX2 rejimi ΔG u açılımı serbest enerji belirlenmesini ve bir yapı elemanının bu nedenle stabilitesini sağlar. Yerli devlet koşul altında EX2 rejimi en yaygın olanıdır. PH ve kaotropik ajanların eklenmesi artış EX1 için değişim mekanizmasını değiştirebilir. Bu nedenle, HX-MS termodinamiği keşfetmek hem de protein katlanma ve yapısal değişikliklerin kinetik parametreler için kullanılabilir.
HX yukarıda belirtildiği gibi doğal olarak pH ve sıcaklığa bağlıdır ve omurga amid grubu bir solvent tamamen açık proton değişim yarılanma ömrü> fizyolojik pH'da 5-400 ms (pH 7.6) ve 30 ° C, fakat 10 dakika arasında olduğu bir pH 2,9> 2 saat ortalama ve 0 ° ile 15 saat(Ca bir yarı ömür. 1-2 dakika ile alışverişi bir polipeptidin, ilk omurga amid bağının proton hariç) C. Bu tür değiş tokuş koşulları altında, yavaş dışarı dahil döteronların içerdiği tüm bilgileri kaybetme ile, bu koşullar altında aktif olan proteazlar (örneğin, pepsin) kullanılarak örnek sindirimi mümkündür. Yavaş alışverişi koşullar altında peptik sindirim tanıtılmasından bu yana, tam uzunlukta proteinlerin genel HX kinetik değil, sadece analiz edilebilir ama HX belirli bölgelerde 8,9 lokalize olabilir. Uzamsal çözünme anda 10-30 kalıntıları arasındaki genel olarak üretilen peptik parçalarının, büyüklüğü ile sınırlıdır. Bununla birlikte, pepsin ile bölünmesi nedeniyle spesifik olmayan doğası oluşturulan üst üste binen parçaları, uzamsal çözünürlük bir artışa neden olabilir. Buna ek olarak, çeşitli başka proteazlar, söndürme koşullar altında, ancak, daha az verimli 10 pepsin daha aktif olduğu bulunmuştur. Dahası increauzamsal çözünürlük se gibi elektron yakalama ayrışma (ECD), elektron transfer ayrılma (ETD) ve kızıl ötesi multiphoton ayrılma (IRMPD) 11-13 gibi döteryumlanma desen korunmuş yöntemler ile, gaz fazında peptitlerin parçalanması ile ulaşılabilir. Bu teknikler nedeniyle çarpışma kaynaklı ayrılma (CID) tarafından görülmektedir intramoleküler proton göçü ("karıştırma"), en yaygın olarak kullanılan parçalama tekniği bir uzamsal çözünürlük kaybını önler. Ancak, bu yöntemler her bir peptit için optimizasyon ve yine bu nedenle oldukça zordur.
HX-MS viral kapsid montaj 14-17 dahil olmak üzere protein-ligand ve protein-protein etkileşimlerini analiz etmek için kullanılmıştır. Protein açılımı ve sıcaklık kaynaklı konformasyonel değişiklikler 7,18,19 incelenmiştir hem de yeniden katlama. Fosforilasyonu ve tek bir amino asit mutasyon-ilişkili şekilsel değişiklikleri 16,20 ve nükleotidlerinide kaynaklı değişimler 21,22 analiz edilmiştir. Bu nedenle, bu yöntem derleme ve moleküler makineleri dinamiklerini analiz etmek için ideal uygun görünüyor. Kimin mekanizması büyük genel ilgi çekici biri aday, Hsp90 şaperon karmaşıktır.
Protocol
Representative Results
Discussion
Bir protein için bir bağlanma ortağının etkileşim kaçınılmaz olarak bağlanma sitesinde solvent erişilebilirlik değişikliklere neden olur. Buna ek olarak, pek çok protein, gerçek bağlama arayüzü başka bölgeleri etkileyen bağlanması üzerine dinamik bir konformasyonal değişiklik geçirmektedir. HX-MS bu değişiklikleri izlemek için sağlam bir yöntem ve diğer yöntemler örtemez o zaman ölçekleri proteinlerin yapısal değişimleri ortaya bile yeteneğine sahiptir.
<p class="jove_content"…Divulgaciones
The authors have nothing to disclose.
Acknowledgements
Biz yazının yorumları için M. Boysen teşekkür ederim. Bu proje (: CellNetworks EXC 81/1 Mükemmellik SFB638 ve MA MPM 1278/4-1 ve Kümede) Deutsche Forschungsgemeinschaft tarafından finanse edildi. MPM Mükemmeliyet Kümesi araştırmacı: CellNetworks.
Materials
| Reagent/Equipment | Company | Catalogue number | Comments (optional) |
| maXis QTOF | Bruker | ||
| nanoAcquity UPLC | Waters Corp. | ||
| Shimadzu 10AD-VP | Shimadzu | ||
| 6-port Valve EPC6W with microelectric actuator | Valco | #EPC6W | |
| Injection valve (manual) | Rheodyne | #7725 | |
| Poros AL20 media | Applied Biosystems | #1-6029-06 | |
| Poros R2 | Applied Biosystems | #1-1118-02 | |
| Pepsin | Sigma | #P6887 | use fresh pepsin |
| Microbore (1 mm) | IDEX | #C-128 | |
| Microbore (2 mm) | IDEX | #C-130B | |
| Acquity UPLC BEH C8 Column | Waters Corp. | #186002876 | |
| Thermomixer | Eppendorf | #5355000.011 | |
| Tubing (various diameters) | IDEX | ||
| Fittings | IDEX | #PK-110 with PK-100 |
Referencias
- Saibil, H. R. Conformational changes studied by cryo-electron microscopy. Nat. Struct. Biol. 7 (9), 711-714 (2000).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Hoofnagle, A. N., Resing, K. A., Ahn, N. G. Protein analysis by hydrogen exchange mass spectrometry. Annu. Rev. Biophys. Biomol. Struct. 32, 1-25 (2003).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass. Spectrom. Rev. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. -. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chem Soc. Rev. 40 (3), 1224-1210 (2011).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Rist, W., Jørgensen, T. J. D., Roepstorff, P., Bukau, B., Mayer, M. P. Mapping temperature-induced conformational changes in the Escherichia coli heat shock transcription factor sigma 32 by amide hydrogen exchange. The Journal of biological chemistry. 278 (51), 51415-51421 (1074).
- Englander, J. J., Rogero, J. R., Englander, S. W. Protein hydrogen exchange studied by the fragment separation method. Anal Biochem. 147 (1), 234-244 (1985).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Cravello, L., Lascoux, D., Forest, E. Use of different proteases working in acidic conditions to improve sequence coverage and resolution in hydrogen/deuterium exchange of large proteins. Rapid Commun. Mass Spectrom. : RCM. 17 (21), 2387-2393 (2003).
- Rand, K. D., Zehl, M., Jensen, O. N., Jørgensen, T. J. D. Protein hydrogen exchange measured at single-residue resolution by electron transfer dissociation mass spectrometry. Anal chem. 81 (14), 5577-5584 (2009).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Electron capture dissociation of electrosprayed protein ions for spatially resolved hydrogen exchange measurements. J. Am. Chem. Soc. 130 (35), 11574-11575 (2008).
- Yamada, N., Suzuki, E. -. I., Hirayama, K. Identification of the interface of a large protein-protein complex using H/D exchange and Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 16 (4), 293-299 (2002).
- Lee, T., Hoofnagle, A. N., et al. Docking motif interactions in MAP kinases revealed by hydrogen exchange mass spectrometry. Mol. Cell. 14 (1), 43-55 (2004).
- Hasan, A., Smith, D. L., Smith, J. B. Alpha-crystallin regions affected by adenosine 5′-triphosphate identified by hydrogen-deuterium exchange. Biochem. 41 (52), 15876-15882 (2002).
- Lanman, J., Lam, T. T., Emmett, M. R., Marshall, A. G., Sakalian, M., Prevelige, P. E. Key interactions in HIV-1 maturation identified by hydrogen-deuterium exchange. Nat. Struct. Mol. Biol. 11 (7), 676-677 (2004).
- Wang, L., Lane, L. C., Smith, D. L. Detecting structural changes in viral capsids by hydrogen exchange and mass spectrometry. Protein Sci. 10 (6), 1234-1243 (2001).
- Pan, H., Raza, A. S., Smith, D. L. Equilibrium and kinetic folding of rabbit muscle triosephosphate isomerase by hydrogen exchange mass spectrometry. J. Mol. Biol. 336 (5), 1251-1263 (2004).
- Mazon, H., Marcillat, O., Forest, E., Smith, D. L., Vial, C. Conformational dynamics of the GdmHCl-induced molten globule state of creatine kinase monitored by hydrogen exchange and mass spectrometry. Biochem. 43 (17), 5045-5054 (2004).
- Lanman, J., Lam, T. T., et al. Identification of novel interactions in HIV-1 capsid protein assembly by high-resolution mass spectrometry. J. Mol. Biol. 325 (4), 759-772 (2003).
- Rist, W., Graf, C., Bukau, B., Mayer, M. P. Amide hydrogen exchange reveals conformational changes in hsp70 chaperones important for allosteric regulation. J. Biol. chem. 281 (24), 16493-16501 (2006).
- Graf, C., Stankiewicz, M., Kramer, G., Mayer, M. P. Spatially and kinetically resolved changes in the conformational dynamics of the Hsp90 chaperone machine. EMBO J. 28 (5), 602-613 (2009).
- Lee, C. -. T., Graf, C., Mayer, F. J., Richter, S. M., Mayer, M. P. Dynamics of the regulation of Hsp90 by the co-chaperone Sti1. EMBO J. 31 (6), 1518-1528 (2012).
- Lou, X., Kirchner, M., et al. Deuteration distribution estimation with improved sequence coverage for HX/MS experiments. Bioinformatics. 26 (12), 1535-1541 (2010).
- Kreshuk, A., Stankiewicz, M., Lou, X., Kirchner, M., Hamprecht, F. A., Mayer, M. P. Automated detection and analysis of bimodal isotope peak distributions in H/D exchange mass spectrometry using HeXicon. Intl. J. mass spectrom. 302 (1-3), 125-131 (2011).
- Walters, B. T., Ricciuti, A., Mayne, L., Englander, S. W. Minimizing back exchange in the hydrogen exchange-mass spectrometry experiment. J. Am. Soc. Mass Spectrom. 23 (12), 2132-2139 (2012).
- Weis, D. D., Wales, T. E., Engen, J. R., Hotchko, M., Ten Eyck, L. F. Identification and characterization of EX1 kinetics in H/D exchange mass spectrometry by peak width analysis. J. Am. Soc. Mass Spectrom. 17 (11), 1498-1509 (2006).
- Fang, J., Rand, K. D., Beuning, P. J., Engen, J. R. False EX1 signatures caused by sample carryover during HX MS analyses. Intl. J. Mass Spectrom. 302 (1-3), 19-25 (2011).
- Deng, Y., Smith, D. L. Identification of unfolding domains in large proteins by their unfolding rates. Biochem. 37 (18), 6256-6262 (1998).
- Wales, T. E., Engen, J. R. Partial unfolding of diverse SH3 domains on a wide timescale. J. Mol. Biol. 357 (5), 1592-1604 (2006).
- Pan, Y., Piyadasa, H., O’Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by h/d exchange and covalent labeling: the glycerol facilitator. J. Mol. Biol. 416 (3), 400-413 (2012).
- Hebling, C. M., Morgan, C. R., Stafford, D. W., Jorgenson, J. W., Rand, K. D., Engen, J. R. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Anal chem. 82 (13), 5415-5419 (2010).
- Abzalimov, R. R., Kaplan, D. A., Easterling, M. L., Kaltashov, I. A. Protein conformations can be probed in top-down HDX MS experiments utilizing electron transfer dissociation of protein ions without hydrogen scrambling. J. Am. Soc. Mass Spectrom. 20 (8), 1514-1517 (2009).

