Induzierbares T7-RNA-Polymerase-vermitteltes Multigen-Expressionssystem, pMGX
Summary
Diese Studie beschreibt Methoden für die T7-vermittelte Co-Expression von mehreren Genen aus einem einzigen Plasmid in Escherichia coli unter Verwendung des pMGX-Plasmidsystems.
Abstract
Die Co-Expression mehrerer Proteine ist für die synthetische Biologie immer wichtiger, studiert Protein-Protein-Komplexe und charakterisiert und nutzt Biosynthesewege. In diesem Manuskript wird die Verwendung eines hochwirksamen Systems für die Konstruktion von multigenen synthetischen Operons unter der Kontrolle einer induzierbaren T7-RNA-Polymerase beschrieben. Dieses System erlaubt es, dass viele Gene gleichzeitig aus einem Plasmid exprimiert werden. Hier ist ein Satz von vier verwandten Vektoren, pMGX-A, pMGX-hisA, pMGX-K und pMGX-hisK, entweder mit dem Ampicillin- oder Kanamycin-resistente Marker (A und K) und entweder mit einem N-terminalen Hexahistidin oder einem N-terminalen Hexahistidin Tag (seine) werden offenbart. Detaillierte Protokolle für die Konstruktion von synthetischen Operen, die dieses Vektorsystem verwenden, werden zusammen mit den entsprechenden Daten bereitgestellt, was zeigt, dass ein pMGX-basiertes System, das fünf Gene enthält, leicht konstruiert und verwendet werden kann, um alle fünf codierten Proteine in Escherichia coli zu produzieren . Dieses systEm und Protokoll ermöglicht es Forschern, routinemäßig komplexe Mehrkomponentenmodule und Wege in E. coli auszudrücken.
Introduction
Die Koexpression mehrerer Proteine ist zunehmend von wesentlicher Bedeutung, vor allem in synthetischen Biologieanwendungen, bei denen mehrere Funktionsmodule ausgedrückt werden müssen 1 ; Bei der Untersuchung von Protein-Protein-Komplexen, wo Expression und Funktion oft Co-Expression 2 , 3 erfordern; Und bei der Charakterisierung und Nutzung von Biosynthesewegen, wo jedes Gen im Weg ausgedrückt werden muss 4 , 5 , 6 , 7 , 8 . Eine Reihe von Systemen wurde für die Co-Expression entwickelt, insbesondere im Wirtsorganismus Escherichia coli , dem Arbeitspferd für die rekombinante Protein-Expression des Labors 9 . Zum Beispiel können mehrere Plasmide mit unterschiedlichen selektierbaren Markern verwendet werden, um einzelne Proteine unter Verwendung einer Fülle von verschiedenen ex zu exprimieren Druckvektoren 10 , 11 . Einzelne Plasmidsysteme für die multiple Proteinexpression haben entweder mehrere Promotoren verwendet, um die Expression jedes Gens 10 , 12 zu kontrollieren; Synthetische Operons, wo mehrere Gene auf einem einzigen Transkript 2 , 13 codiert sind; Oder in einigen Fällen ein einzelnes Gen, das für ein Polypeptid kodiert, das letztlich proteolytisch verarbeitet wird, wobei die gewünschten interessierenden Proteine 14 erhalten werden .
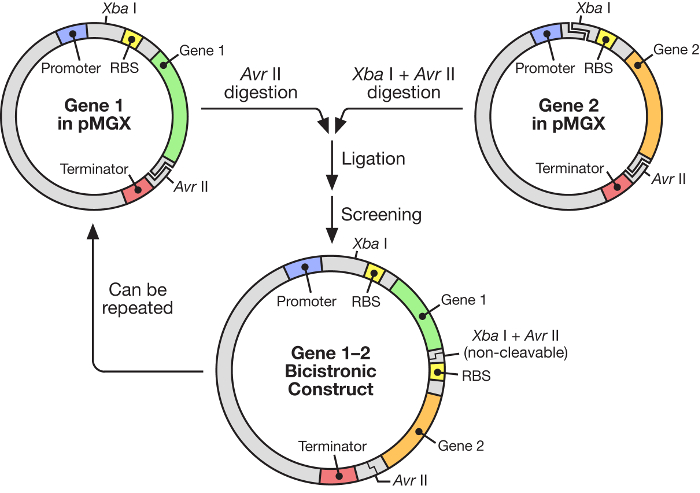
Abbildung 1: pMGX-Workflow, der den Aufbau eines polycistronischen Vektors zeigt. Das pMGX-System bietet eine flexible, einfach zu bedienende Strategie für den Bau von synthetischen Operons unter der Kontrolle eines induzierbaren T7-Promotors.E.com/files/ftp_upload/55187/55187fig1large.jpg "target =" _ blank "> Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
In diesem Manuskript wird die Verwendung eines hochwirksamen Systems für die Konstruktion von multigenen synthetischen Operons unter der Kontrolle einer induzierbaren T7-RNA-Polymerase ( Fig. 1 ) beschrieben. Dieses System erlaubt es, dass viele Gene gleichzeitig aus einem Plasmid exprimiert werden. Es basiert auf einem Plasmidsystem, das ursprünglich pKH22 genannt wurde und das für eine Reihe von verschiedenen Anwendungen 6 , 7 , 8 erfolgreich eingesetzt wurde . Hier wird dieser Plasmidsatz um vier verwandte Vektoren erweitert: pMGX-A, ein Expressionsvektor ohne C- oder N-terminale Markierungen und mit dem Ampicillin-Resistenzmarker; PMGX-hisA, ein Expressionsvektor, der für ein N-terminales Hexahistidin-Tag und mit dem Ampicillin-Resistenzmarker kodiert; PMGX-K, ein Expressionsvektor lAcking alle C- oder N-terminalen Markierungen und mit dem Kanamycin-Resistenz-Marker; Und pMGX-hisK, ein Expressionsvektor, der für ein N-terminales Hexahistidin-Tag und mit dem Kanamycin-Resistenzmarker kodiert. In dieser Studie wird das Verfahren zur Erzeugung eines polycistronischen Vektors, der fünf Gene enthält, unter Verwendung des pMGX-Systems, insbesondere pMGX-A, zusammen mit der erfolgreichen Produktion jedes einzelnen Proteins in Escherichia coli gezeigt .
Protocol
Representative Results
Discussion
Die Koexpression mehrerer Gene ist immer wichtiger, insbesondere bei der Charakterisierung und Rekonstitution komplexer, multigener Stoffwechselwege 3 , 4 , 5 . Das pMGX-System macht die Multigene-Koexpression in der E. coli- Routine 6 , 7 , 8 und ist für verschiedene Forscher zugänglich. In dieser Studie wurden fünf Protei…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Diese Arbeit wurde vom Naturwissenschaften und Ingenieurforschungsrat von Kanada unterstützt.
Materials
| Enzymes | |||
| Alkaline Phosphatase, Calf Intestinal (CIP) | New England Biolabs | M0290S | |
| AvrII | New England Biolabs | R0174S | |
| EcoRI | New England Biolabs | R0101S | |
| NdeI | New England Biolabs | R0111S | |
| XbaI | New England Biolabs | R0145S | |
| Herculase II Fusion DNA Polymerase | Agilent Technologies | 600677 | |
| T4 DNA Ligase | New England Biolabs | M0202S | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 1 kb DNA ladder | New England Biolabs | N3232L | |
| 4-20% Mini-PROTEAN TGX Stain-Free Protein Gels | Bio-Rad | 456-8095 | |
| 50 x TAE | Fisher Thermo Scientific | BP1332-4 | |
| Agar | Fisher Thermo Scientific | BP1423-500 | |
| Agarose | Fisher Thermo Scientific | BP160-500 | |
| Ampicilin | Sigma-Alrich | A9518-5G | |
| BL21 (DE3) chemically comeptent cells | Comeptent cell prepared in house | ||
| B-PER Bacterial Protein Extraction Reagent | Fisher Thermo Scientific | PI78243 | |
| dNTP mix | Agilent Technologies | Supplied with polymerase | |
| Gel Extraction Kit | Omega | D2500-02 | E.Z.N.A Gel Extraction, supplied by VWR Cat 3: CA101318-972 |
| Glycine | Fisher Thermo Scientific | BP381-1 | |
| His Tag Antibody [HRP], mAb, Mouse | GenScript | A00612 | |
| Immobilon Western Chemiluminescent HRP Substrate | EMD Millipore | WBKLS0100 | |
| IPTG | Sigma-Alrich | 15502-10G | |
| LB | Fisher Thermo Scientific | BP1426-500 | |
| Methanol | Fisher Thermo Scientific | A411-20 | |
| Pasteurized instant skim milk powder | Local grocery store | No-name grocery store milk is adequate | |
| Nitrocellulose membrane | Amersham Protran (GE Healthcare Life Sciences) | 10600007 | Membrane PT 0.45 µm 200 mm X 4 m, supplied by VWR Cat #: CA10061-086 |
| Plasmid DNA Isolation Kit | Omega | D6943-02 | E.Z.N.A Plasmid DNA MiniKit I, supplied by VWR Cat #: CA101318-898 |
| pMGX | Boddy Lab | Request from the Boddy Lab Contact cboddy@uottawa.ca | |
| Primers | Intergrated DNA Technologies | Design primers as needed for desired gene | |
| Synthetic Gene | Life Technologies | Design and optimize as needed | |
| Thick Blot Filter Paper | Bio-Rad | 1703932 | |
| Tris base | BioShop | TRS001.1 | |
| Tween-20 | Sigma-Alrich | P9416-50ML | |
| XL1-Blue chemically competent cells | Comeptent cell prepared in house | ||
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| BioSpectrometer | Eppendorf | RK-83600-07 | |
| Gel box – PAGE | Bio-Rad | 1658005 | Mini-PROTEIN Tetra Vertical Electrophoresis Cell |
| Gel Imager | Alpha Innotech | AlphaImager EC | |
| Incubator-oven | Fisher Thermo Scientific | 11-690-650D | Isotemp |
| Incubator-shaker | Fisher Thermo Scientific | SHKE6000-7 | MaxQ 6000 |
| Personna Razors | Fisher Thermo Scientific | S04615 | |
| Power Pack | Bio-Rad | S65533Q | FB300 |
| Transilluminator | VWR International | M-10E,6W | |
| Thermocylcer | Eppendorf | Z316091 | Mastercycler Personal, supplied by Sigma |
| UV Face-Shield | 18-999-4542 | ||
| Waterbath | Fisher Thermo Scientific | 15-460-2SQ | |
| Western Transfer Apparatus | Bio-Rad | 1703935 | Mini-Trans Blot Cell |
Riferimenti
- Purnick, P. E. M., Weiss, R. The second wave of synthetic biology: from modules to systems. Nat. Rev. Mol. Cell Biol. 10 (6), 410-422 (2009).
- Bieniossek, C., et al. Automated unrestricted multigene recombineering for multiprotein complex production. Nat. Methods. 6 (6), 447-450 (2009).
- Jochimsen, B., et al. Five phosphonate operon gene products as components of a multi-subunit complex of the carbon-phosphorus lyase pathway. Proc. Natl. Acad. Sci. USA. 108 (28), 11393-11398 (2011).
- Martin, V. J. J., Pitera, D. J., Withers, S. T., Newman, J. D., Keasling, J. D. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. Nat. Biotechnol. 21 (7), 796-802 (2003).
- Ajikumar, P. K., et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli. Science. 330 (6000), 70-74 (2010).
- Boddy, C. N., Hotta, K., Tse, M. L., Watts, R. E., Khosla, C. Precursor-directed biosynthesis of epothilone in Escherichia coli. J. Am. Chem. Soc. 126 (24), 7436-7437 (2004).
- Lundgren, B. R., Boddy, C. N. Sialic acid and N-acyl sialic acid analog production by fermentation of metabolically and genetically engineered Escherichia coli. Org. Biomol. Chem. 5 (12), 1903-1909 (2007).
- Horsman, M. E., Lundgren, B. R., Boddy, C. N. N-Acetylneuraminic Acid Production in Escherichia coli Lacking N-Acetylglucosamine Catabolic Machinery. Chem. Eng. Commun. 203 (10), 1326-1335 (2016).
- Romier, C., et al. Co-expression of protein complexes in prokaryotic and eukaryotic hosts: experimental procedures, database tracking and case studies. Acta Crystallogr. D, Biol. Crystallogr. 62 (Pt 10), 1232-1242 (2006).
- Tolia, N. H., Joshua-Tor, L. Strategies for protein coexpression in Escherichia coli. Nat. Methods. 3 (1), 55-64 (2006).
- Chanda, P. K., Edris, W. A., Kennedy, J. D. A set of ligation-independent expression vectors for co-expression of proteins in Escherichia coli. Protein Expr. Purif. 47 (1), 217-224 (2006).
- Kim, K. -. J., et al. Two-promoter vector is highly efficient for overproduction of protein complexes. Protein Sci. 13 (6), 1698-1703 (2004).
- Tan, S., Kern, R. C., Selleck, W. The pST44 polycistronic expression system for producing protein complexes in Escherichia coli. Protein Expr. Purif. 40 (2), 385-395 (2005).
- Chen, X., Pham, E., Truong, K. TEV protease-facilitated stoichiometric delivery of multiple genes using a single expression vector. Protein Sci. 19 (12), 2379-2388 (2010).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J. Vis. Exp. (63), e3998 (2012).
- Lee, P. Y., Costumbrado, J., Hsu, C. -. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J. Vis. Exp. (62), (2012).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J. Vis. Exp. (48), (2011).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Molecular Cloning. J. Vis. Exp Available from: https://www.jove.com/science-education/5074/molecular-cloning (2016)
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. The Western Blot. J. Vis. Exp Available from: https://www.jove.com/science-education/5065/the-western-blot (2016)
- Laible, M., Boonrod, K. Homemade site directed mutagenesis of whole plasmids. J. Vis. Exp. (27), (2009).

