תסיסה מבוקרת אור לייצור כימיקלים וחלבון מיקרוביאליים
Summary
שליטה אופטוגנטית בחילוף החומרים המיקרוביאלי מציעה שליטה דינמית גמישה על תהליכי תסיסה. הפרוטוקול כאן מראה כיצד להגדיר תסיסה מווסתת אור כחול לייצור כימי וחלבון בקנה מידה נפחי שונה.
Abstract
מפעלי תאים מיקרוביאליים מציעים חלופה בת קיימא לייצור כימיקלים וחלבונים רקומביננטיים ממזון מתחדש. עם זאת, עומס יתר על מיקרואורגניזם עם שינויים גנטיים יכול להפחית את הכושר והפרודוקטיביות של המארח. ניתן להתגבר על בעיה זו באמצעות שליטה דינמית: ביטוי בלתי ניתן לערעור של אנזימים ומסלולים, בדרך כלל באמצעות תוספים כימיים או מבוססי חומרים מזינים, כדי לאזן את הצמיחה והייצור של התאים. אופטוגנטיקה מציעה שיטה לא פולשנית, מאוד טונה והפכה של ויסות דינמי של ביטוי גנים. כאן, אנו מתארים כיצד להגדיר תסיסה מבוקרת אור של קולי Escherichia מהונדס ו Saccharomyces cerevisiae לייצור כימיקלים או חלבונים רקומביננטיים. אנו דנים כיצד ליישם אור בזמנים ובמינונים נבחרים כדי לנתק את הצמיחה והייצור של מיקרוביאלית לשליטה משופרת בתסיסה ופרודוקטיביות, כמו גם את שיקולי האופטימיזציה העיקריים לקבלת התוצאות הטובות ביותר. בנוסף, אנו מתארים כיצד ליישם בקרות אור לניסויים ביו-ריאקטור בקנה מידה מעבדה. פרוטוקולים אלה מקלים על אימוץ בקרות אופטוגנטיות במיקרואורגניזמים מהונדסים לביצועי תסיסה משופרים.
Introduction
אופטוגנטיקה, השליטה בתהליכים ביולוגיים עם חלבונים מגיבים לאור, מציעה אסטרטגיה חדשה לשליטה דינמית בתסיסות מיקרוביות לייצור כימי וחלבון 1,2. הנטל של מסלולים מטבוליים מהונדסים ואת הרעילות של כמה מתווכים ומוצרים לעתים קרובות פוגע צמיחת התא3. לחצים כאלה יכולים להוביל הצטברות ביומסה לקויה ופרודוקטיביות מופחתת3. אתגר זה ניתן לטפל על ידי חלוקה זמנית תסיסה לשלב צמיחה וייצור, אשר מקדישים משאבים מטבוליים הצטברות ביומסה או סינתזת מוצר בהתאמה4. לאחרונה הראינו כי המעבר מצמיחה לייצור בתסיסה דו-שלבית זו יכול להיות מושרה עם שינויים בתנאי תאורה5,6,7. הטונה הגבוהה, הפיכות והאורתוגונליות של תשומות אור8 מציעות יתרונות ייחודיים לתסיסות מבוקרות אור שקשה או בלתי אפשרי לשכפל עם ממריצים כימיים המשמשים לשליטה דינמית בתסיסות דו-פאזיות קונבנציונליות4,9,10,111.
חלבון EL222 מגיב אור כחול נגזר ליטורליס אריתרובקטריה שימש לפיתוח מספר מעגלים אופטוגנטיים להנדסה מטבולית ב Saccharomyces cerevisiae5,7,12,13. EL222 מכיל תחום חיישן מתח אור-חמצן (LOV) שעובר שינוי קונפורמציה עם הפעלת אור כחול (465 ננומטר), המאפשר לו להיקשר לרצף הדנ”א ההכרה שלו (C120)13. היתוך EL222 לתחום ההפעלה הנגיפי VP16 (VP16-EL222) מביא לגורם שעתוק מגיב לאור כחול שיכול להפעיל באופן הפיך ביטוי גנים ב- S. cerevisiae7 ובאורגניזמים אחרים14 מהמקדם הסינתטי PC120. מספר מעגלים המבוססים על EL222 פותחו ושימשו לייצור כימי ב- S. cerevisiae, כגון מערכת OptoEXP הבסיסית המופעלת באמצעות אור7, שבה גן העניין בא לידי ביטוי ישירות מ- PC120 (איור 1A). עם זאת, החששות מפני חדירת אור בצפיפות התאים הגבוהה שבדרך כלל נתקלים בהם בשלב הייצור של התסיסות, הניעה אותנו לפתח מעגלים הפוכים המושרים בחושך, כגון מעגלי OptoINVRT ו- OptoQ-INVRT (איור 1B)5,7,13. מערכות אלה לרתום את הגלקטוז (GAL) או חומצה קווינית (Q) regulons מ S. cerevisiae ו N. crassa, בהתאמה, שליטה על הדיכויים המקבילים שלהם (GAL80 ו QS) עם VP16-EL222, כדי להדחיק ביטוי גנים באור ולגרום לו חזק בחושך. שילוב מעגלי OptoEXP ו-OptoINVRT מביא לשליטה דו-כיוונית בביטוי הגנים, ומאפשר תסיסה דו-שלבית שבה שלב הצמיחה מושרה באור כחול, ושלב הייצור עם חושך (איור 2A)5,7.
שימוש באור במקום בחושך כדי לגרום לביטוי גנים במהלך שלב הייצור ירחיב מאוד את היכולות של בקרות אופטוגנטיות, אך גם ידרוש התגברות על מגבלות חדירת האור של צפיפות התאים הגבוהה שבדרך כלל נתקלת בה בשלב זה של תסיסה. לשם כך, פיתחנו מעגלים, המכונים OptoAMP ו- OptoQ-AMP, המגבירים את תגובת התמלול לגירוי אור כחול. מעגלים אלה משתמשים במוטנטים פראיים או רגישים יתר על המידה של VP16-EL222 כדי לשלוט בייצור של מפעילי התמלול Gal4p או QF2 של regulons GAL או Q, בהתאמה, השגת רגישות משופרת וביטוי גנים חזק יותר עם אור 12,13 (איור 1C). מעגלי OptoAMP יכולים להשיג אינדוקציה מלאה והומוגנית של אור ב-5 L bioreactors בצפיפות אופטית (נמדדת ב-600 ננומטר; OD600) ערכים של לפחות 40 עם רק ~0.35% של תאורה (5% מינון אור רק ~ 7% של פני השטח בתפזורת). זה מדגים רמה גבוהה יותר של רגישות לעומת OptoEXP, אשר דורש קרוב 100% תאורה12. היכולת לגרום ביעילות לביטוי גנים עם אור בצפיפות תאים גבוהה פותחת הזדמנויות חדשות לשליטה דינמית בתסיסות. זה כולל תסיסה תפעולית ביותר משני שלבים זמניים, כגון תסיסה תלת-פאזית, שבה שלבי צמיחה, אינדוקציה וייצור נקבעים עם לוחות זמנים ייחודיים של אור כדי לייעל את הייצור הכימי (איור 2B)12.
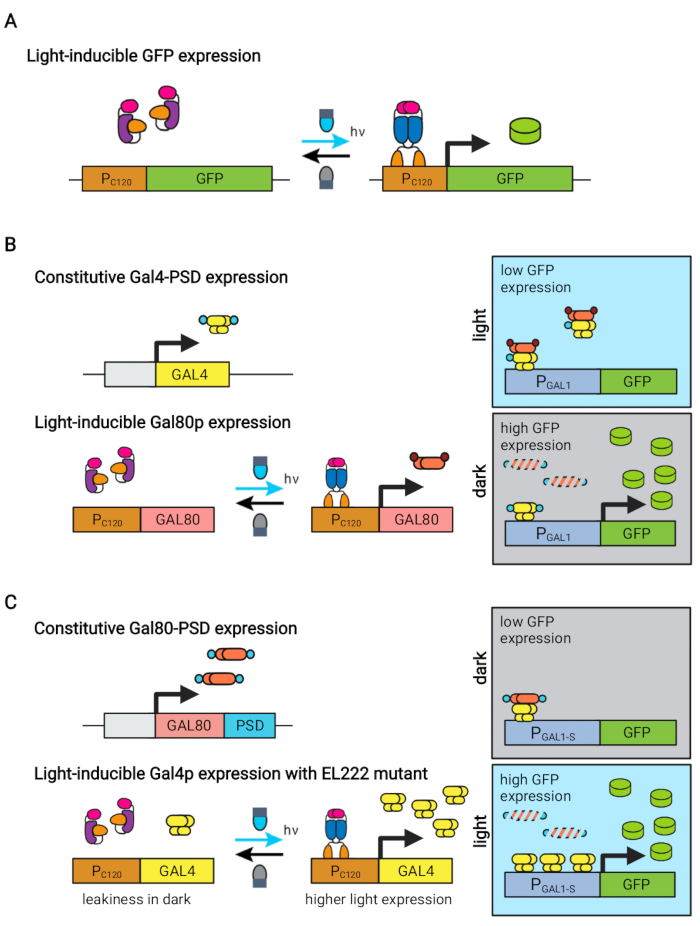
איור 1: מעגלים אופטוגנטיים לשליטה דינמית ב-S. cerevisiae. מעגלי OptoEXP, OptoINVRT ו-OptoAMP מבוססים על מערכת VP16-EL222 הרגישה לאור. (א) במעגל OptoEXP, חשיפה לאור כחול גורמת לשינוי קונפורמציה ודימום של VP16-EL222, החושף תחום מחייב DNA ומאפשר שעתוק מ- PC120. הנתון שונה מז’או ואח’ 7. (ב) מעגלי OptoINVRT רותמים את התגמולונים GAL (המוצגים) או Q כדי לגרום לביטוי בחושך. במעגלים מבוססי GAL, VP16-EL222 ו- GAL4 באים לידי ביטוי באופן מכונן, בעוד PC120 מניע ביטוי של מדכא GAL80 (במעגלים מבוססי Q, GAL4 ו- GAL80 מוחלפים על ידי QF2 ו- QS, בהתאמה, ומקדם סינתטי המכיל QUAS משמש במקום מקדם GAL). לאור זאת, Gal80p מונע הפעלה של הגן של עניין מ PGAL1. בחושך, GAL80 אינו בא לידי ביטוי ומושפל במהירות על ידי היתוך אותו לתחום degron מכונן (תחום חום קטן), המאפשר הפעלה של PGAL1 על ידי Gal4p. הנתון שונה מז’או ואח’ 5. (ג) מעגלי OptoAMP משתמשים גם ב-VP16-EL222 כדי לשלוט ברגולונים GAL (מוצגים) או Q. במעגלים אלה, מדכא GAL80 (או QS) מבוטא באופן מכונן ומותך לדגרון רגיש לצילום (תחום כחול קטן) המבטיח דיכוי הדוק בחושך. PC120 וביטוי בקרת מוטציה VP16-EL222 רגיש של GAL4 (או QF2) עם אור, אשר מפעיל בעוצמה PGAL1 (או מקדם המכיל QUAS) באור. מעגלים שמקורם ב-GAL יכולים להשתמש בצורות מהונדסות של PGAL1, כגון PGAL1-M או PGAL1-S, אשר הגבירו את הפעילות, כמו גם מקדמים מסוג בר הנשלטים על ידי ה-GAL regulon (PGAL1, PGAL10, PGAL2, PGAL7). האיור שונה מ- Zhao et al.12. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
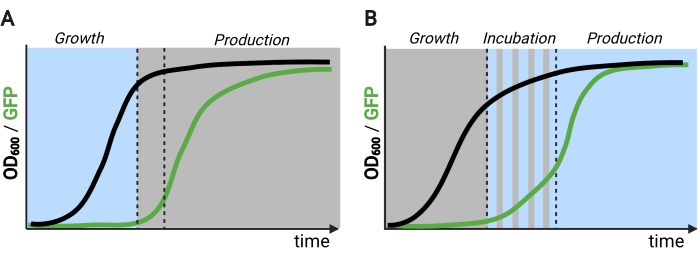
איור 2: תסיסה דו-שלבית ותלת-פאזית לאורך זמן. (A) תסיסה דו-פאזית המופעלת עם מעגלים הפוכים מורכבת משלב צמיחה מונחה אור ושלב ייצור כהה. בשלב הצמיחה, ביומסה מצטברת כאשר מסלול הייצור נשאר מודחק. עם ההגעה OD600 הרצוי, תאים מועברים אל החושך כדי להתאים מטבולית לפני להיות resuspended במדיה טרייה עבור שלב הייצור. (ב) בתהליך תלת-שלבי, שלבי הצמיחה, הדגירה והייצור מוגדרים על ידי לוחות זמנים ייחודיים של אור, שעשויים להיות מורכבים מתקופה של צמיחה חשוכה, דגירה פועמת ושלב ייצור מואר לחלוטין. דמות שנוצרה עם ביורנדר. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
מעגלים אופטוגנטיים פותחו גם לשליטה דינמית בייצור כימי וחלבון ב– E. coli. מעגלי OptoLAC שולטים במדכא LacI החיידקי באמצעות מעגל pDawn בעל תגובת האור, המבוסס על מערכת שני הרכיבים YF1/FixJ6 (איור 3). בדומה ל- OptoINVRT5, מעגלי OptoLAC נועדו להדחיק ביטוי גנים באור ולגרום לו בחושך. רמות הביטוי באמצעות מעגלי OptoLAC יכולות להתאים או לחרוג מאלה שהושגו עם אינדוקציה סטנדרטית של איזופרופיל β-d-1-thiogalactopyranoside (IPTG), ובכך לשמור על כוח האינדוקציה הכימית תוך מתן טונה והפכה משופרות6. לכן, מעגלי OptoLAC מאפשרים שליטה אופטוגנטית יעילה להנדסה מטבולית ב– E. coli.
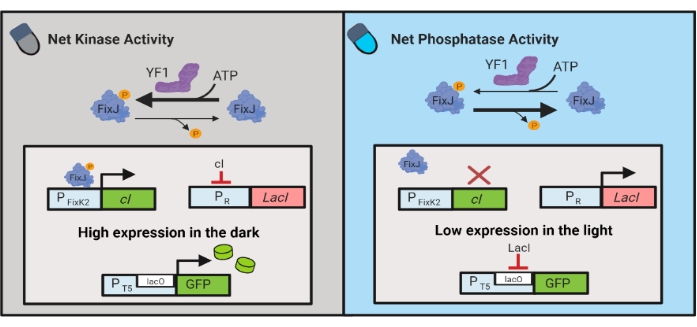
איור 3: מעגלי OptoLAC לשליטה דינמית ב-E. coli. מעגלי OptoLAC מתאימים את מערכת ה-pDawn ואת אופרון הלק כדי להשיג הפעלה בחושך ודיכוי באור. בחושך, YF1 זרחן FixJ, אשר לאחר מכן מפעיל את מקדם PFixK2 להביע את מדכא cI . מדכא ה – cI מונע ביטוי של מדחיק lacI ממקדם ה- PR , המאפשר שעתוק של גן העניין ממקדם המכיל לאקו. לעומת זאת, אור כחול מפחית את פעילות קינאז נטו YF1, היפוך זרחן FixJ ובכך ביטוי cI , אשר מפחית את הביטוי של lacI ומונע ביטוי מהמקדם המכיל לאקו. הנתון שונה מ Lalwani et al.6. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
אנו מתארים כאן את הפרוטוקולים הבסיסיים לתסיסות מבוקרות אור של S. cerevisiae ו – E. coli לייצור כימי או חלבון. עבור שמרים וחיידקים כאחד, אנו מתמקדים תחילה בתסיסות עם שלב צמיחה מונחה אור ושלב ייצור המושרה בחושך המופעל על ידי מעגלי OptoINVRT ו- OptoLAC. לאחר מכן, אנו מתארים פרוטוקול לתסיסה תלת-פאזית (צמיחה, אינדוקציה, ייצור) מבוקרת אור המופעלת על ידי מעגלי OptoAMP. יתר על כן, אנו מתארים כיצד להרחיב תסיסה מבוקרת אופטוגנטית ממיקרו-לוחות ל-bioreactors בקנה מידה של מעבדה. עם פרוטוקול זה, אנו שואפים לספק מדריך מלא וקל לשחזור לביצוע תסיסה מבוקרת אור לייצור כימי או חלבון.
Protocol
Representative Results
Discussion
שליטה דינמית כבר מזמן יושם כדי לשפר את התשואות עבור הנדסה מטבולית וייצור חלבון רקומביננטי4. שינויים בביטוי אנזימטי מיושמים בדרך כלל באמצעות ממריצים כימיים כגון IPTG21, galactose22 ו tetracycline23, אך גם תווכו באמצעות תנאי תהליך כגון טמפרטורה ו- pH. שליט…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
מחקר זה נתמך על ידי משרד האנרגיה של ארה”ב, משרד המדע, המשרד למחקר ביולוגי וסביבתי פרס DE-SC0019363, פרס הקריירה של NSF CBET-1751840, קרנות הצדקה של פיו ופרס המורה-מלומד של קמיל דרייפוס.
Materials
| Light-controlled chemical production using S. cerevisiae | |||
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Agar powder | Thermo Fisher Scientific | 303991049 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| EZ-L439 OptoINVRT7 Plasmid | N/A | N/A | See Reference 1 |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| PmeI | New England Biolabs | R0560L | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| Replica-plating device | Thomas Scientific | F37848-0000 | |
| Replica-plating pads | Sunrise Science Products | 3005-012 | |
| SC-His powder | Sunrise Science Products | 1303-030 | |
| SC Complete powder | Sunrise Science Products | 1459-100 | |
| Sterile sealing film | Excel Scientific | STR-SEAL-PLT | |
| YPD agar plates | VWR | 100217-054 | |
| Zeocin | Thermo Fisher Scientific | R25005 | |
| Light-controlled protein production using E. coli | |||
| 6X SDS Sample Buffer | Cepham Life Sciences | 10502 | |
| 12% Acrylamide protein gels | Thermo Fisher Scientific | NP0341BOX | |
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| Coomassie Brilliant Blue G-250 | Thermo Fisher Scientific | 20279 | |
| Electrophoresis cell | Bio-Rad | 1658004 | |
| Electrophoresis power supply | Bio-Rad | 1645050 | |
| LB broth (Miller) | Fisher Scientific | BP97235 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NaCl | Thomas Scientific | SX0425-1 | |
| OptoLAC plasmids | N/A | N/A | See Reference 2 |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Thermomixer | Eppendorf | 5382000015 | |
| Tris base | Fisher Scientific | BP1521 | |
| Three-phase fermentation using S. cerevisiae | |||
| Same materials as "Light-controlled chemical production using S. cerevisiae" protocol plus the following: | |||
| EZ-L580 OptoAMP4 Plasmid | N/A | N/A | See Reference 10 |
| Chemical production in a light-controlled bioreactor | |||
| Aluminum foil | Reynolds | B004NG90YO | |
| Antifoam | Sigma-Aldrich | A8311 | |
| Bioreactor with control station | Eppendorf | B120110001 | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Bleach | VWR Scientific | 89501-620 (CS) | |
| Blue LED panel | HQRP | 884667106091218 | |
| BPT tubing | Fisher Scientific | 14-170-15 | |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Hydrochloric acid (HCl) | Fisher Scientific | 7647-01-0 | |
| M9 Minimal Salts | Thermo Fisher Scientific | A1374401 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NH4OH Solution | Sigma-Aldrich | I0503-1VL | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SC Complete powder | Sunrise Science Products | 1459-100 |
Riferimenti
- Figueroa, D., Rojas, V., Romero, A., Larrondo, L. F., Salinas, F. The rise and shine of yeast optogenetics. Yeast. 38 (2), 131-146 (2021).
- Pouzet, S., et al. The promise of optogenetics for bioproduction: Dynamic control strategies and scale-up instruments. Bioingegneria. 7 (4), 151 (2020).
- Venayak, N., Anesiadis, N., Cluett, W. R., Mahadevan, R. Engineering metabolism through dynamic control. Current Opinion in Biotechnology. 34, 142-152 (2015).
- Lalwani, M. A., Zhao, E. M., Avalos, J. L. Current and future modalities of dynamic control in metabolic engineering. Current Opinion in Biotechnology. 52, 56-65 (2018).
- Zhao, E. M., et al. Design and characterization of rapid optogenetic circuits for dynamic control in yeast metabolic engineering. ACS Synthetic Biology. 9 (12), 3254-3266 (2020).
- Lalwani, M. A., et al. Optogenetic control of the lac operon for bacterial chemical and protein production. Nature Chemical Biology. 17 (1), 71-79 (2021).
- Zhao, E. M., et al. Optogenetic regulation of engineered cellular metabolism for microbial chemical production. Nature. 555 (7698), 683-687 (2018).
- Baumschlager, A., Khammash, M. Synthetic biological approaches for optogenetics and tools for transcriptional light-control in bacteria. Advanced Biology. 5 (5), 2000256 (2021).
- Dvorak, P., et al. Exacerbation of substrate toxicity by IPTG in Escherichia coli BL21(DE3) carrying a synthetic metabolic pathway. Microbial Cell Factories. 14, 201 (2015).
- Hartline, C. J., Schmitz, A. C., Han, Y., Zhang, F. Dynamic control in metabolic engineering: Theories, tools, and applications. Metabolic Engineering. 63, 126-140 (2021).
- Ni, C., Dinh, C. V., Prather, K. L. J. Dynamic control of metabolism. Annual Review of Chemical and Biomolecular Engineering. 12, 519-560 (2021).
- Zhao, E. M., et al. Optogenetic amplification circuits for light-induced metabolic control. ACS Synthetic Biology. 10 (5), 1143-1154 (2021).
- Lalwani, M. A., Zhao, E. M., Wegner, S. A., Avalos, J. L. The Neurospora crassa Inducible Q System Enables Simultaneous Optogenetic Amplification and Inversion in Saccharomyces cerevisiae for Bidirectional Control of Gene Expression. ACS Synthetic Biology. 10 (8), 2060-2075 (2021).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature Chemical Biology. 10 (3), 196-202 (2014).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Marx, H., Mecklenbräuker, A., Gasser, B., Sauer, M., Mattanovich, D. Directed gene copy number amplification in Pichia pastoris by vector integration into the ribosomal DNA locus. FEMS Yeast Research. 9 (8), 1260-1270 (2009).
- Nordén, K., et al. Increasing gene dosage greatly enhances recombinant expression of aquaporins in Pichia pastoris. BMC Biotechnology. 11, 47 (2011).
- Zhao, E. M., et al. Light-based control of metabolic flux through assembly of synthetic organelles. Nature Chemical Biology. 15 (6), 589-597 (2019).
- Dowee, W. J., Miller, J. F., Ragsdale, C. W. High efficiency transformation of E. coli by high voltage electroporation. Nucleic Acids Research. 16 (13), 6127-6145 (1988).
- Zhou, K., Edgar, S., Stephanopoulos, G. Engineering microbes to synthesize plant isoprenoids. Methods in Enzymology. 575, 225-245 (2016).
- Arfman, N., Worrell, V., Ingram, L. O. Use of the tac promoter and lacI(q) for the controlled expression of Zymomonas mobilis fermentative genes in Escherichia coli and Zymomonas mobilis. Journal of Bacteriology. 174 (22), 7370-7378 (1992).
- Steen, E. J., et al. Metabolic engineering of Saccharomyces cerevisiae for the production of n-butanol. Microbial Cell Factories. 7 (1), 1-8 (2008).
- Tan, S. Z., Manchester, S., Prather, K. L. J. Controlling central carbon metabolism for improved pathway yields in Saccharomyces cerevisiae. ACS Synthetic Biology. 5 (2), 116-124 (2015).
- Jayaraman, P., et al. Blue light-mediated transcriptional activation and repression of gene expression in bacteria. Nucleic Acids Research. 44 (14), 6994 (2016).
- Fernandez-Rodriguez, J., Moser, F., Song, M., Voigt, C. A. Engineering RGB color vision into Escherichia coli. Nature Chemical Biology. 13 (7), 706-708 (2017).
- Ding, Q., et al. Light-powered Escherichia coli cell division for chemical production. Nature Communications. 11 (1), 1-14 (2020).
- Senoo, S., Tandar, S. T., Kitamura, S., Toya, Y., Shimizu, H. Light-inducible flux control of triosephosphate isomerase on glycolysis in Escherichia coli. Biotechnology and Bioengineering. 116 (12), 3292-3300 (2019).
- Ramakrishnan, P., Tabor, J. J. Repurposing synechocystis PCC6803 UirS-UirR as a UV-violet/green photoreversible transcriptional regulatory tool in E. Coli. ACS Synthetic Biology. 5 (7), 733-740 (2016).
- Tabor, J. J., Levskaya, A., Voigt, C. A. Multichromatic control of gene expression in escherichia coli. Journal of Molecular Biology. 405 (2), 315-324 (2011).
- Stewart, C. J., McClean, M. N. Design and implementation of an automated illuminating, culturing, and sampling system for microbial optogenetic applications. Journal of Visualized Experiments:JoVE. (120), e54894 (2017).
- Grødem, E. O. S., Sweeney, K., McClean, M. N. Automated calibration of optoPlate LEDs to reduce light dose variation in optogenetic experiments. BioTechniques. 69 (4), 313-316 (2020).
- Gerhardt, K. P., et al. An open-hardware platform for optogenetics and photobiology. Scientific Reports. 6, (2016).
- Bugaj, L. J., Lim, W. A. High-throughput multicolor optogenetics in microwell plates. Nature Protocols. 14 (7), 2205-2228 (2019).
- Steel, H., Habgood, R., Kelly, C., Papachristodoulou, A. In situ characterisation and manipulation of biological systems with Chi.Bio. PLoS Biology. 18 (7), (2020).
- Carrasco-López, C., García-Echauri, S. A., Kichuk, T., Avalos, J. L. Optogenetics and biosensors set the stage for metabolic cybergenetics. Current Opinion in Biotechnology. 65, 296-309 (2020).
- Milias-Argeitis, A., Rullan, M., Aoki, S. K., Buchmann, P., Khammash, M. Automated optogenetic feedback control for precise and robust regulation of gene expression and cell growth. Nature Communications. 7 (1), 1-11 (2016).
- Melendez, J., et al. Real-time optogenetic control of intracellular protein concentration in microbial cell cultures. Integrative Biology: Quantitative Biosciences From Nano to Macro. 6 (3), 366-372 (2014).
- Milias-Argeitis, A., et al. In silico feedback for in vivo regulation of a gene expression circuit. Nature Biotechnology. 29 (12), 1114-1116 (2011).
- Castillo-Hair, S. M., Baerman, E. A., Fujita, M., Igoshin, O. A., Tabor, J. J. Optogenetic control of Bacillus subtilis gene expression. Nature Communications. 10 (1), 1-11 (2019).
- Xia, A., et al. Optogenetic modification of pseudomonas aeruginosa enables controllable twitching motility and host infection. ACS Synthetic Biology. 10 (3), 531-541 (2021).
- Pu, L., Yang, S., Xia, A., Jin, F. Optogenetics manipulation enables prevention of biofilm formation of engineered pseudomonas aeruginosa on surfaces. ACS Synthetic Biology. 7 (1), 200-208 (2018).

