Fermentações controladas por luz para produção de produtos químicos e proteicos microbianos
Summary
O controle optogenético do metabolismo microbiano oferece controle dinâmico flexível sobre os processos de fermentação. O protocolo aqui mostra como configurar fermentações reguladas por luz azul para produção química e proteica em diferentes escalas volumosas.
Abstract
As fábricas de células microbianas oferecem uma alternativa sustentável para a produção de produtos químicos e proteínas recombinantes a partir de matérias-primas renováveis. No entanto, sobrecarregar um microrganismo com modificações genéticas pode reduzir o condicionamento físico e a produtividade do hospedeiro. Esse problema pode ser superado usando o controle dinâmico: expressão indutível de enzimas e caminhos, tipicamente usando aditivos à base de químicos ou nutrientes, para equilibrar o crescimento e a produção celular. A optogenética oferece um método não invasivo, altamente tável e reversível de regular dinamicamente a expressão genética. Aqui, descrevemos como configurar fermentações controladas por luz de Escherichia coli e Saccharomyces cerevisiae para a produção de produtos químicos ou proteínas recombinantes. Discutimos como aplicar luz em horários selecionados e dosagens para desacoplar o crescimento e a produção microbiana para melhor controle de fermentação e produtividade, bem como as principais considerações de otimização para melhores resultados. Além disso, descrevemos como implementar controles de luz para experimentos bioreatores em escala de laboratório. Esses protocolos facilitam a adoção de controles optogenéticos em microrganismos projetados para melhor desempenho de fermentação.
Introduction
A optogenética, o controle de processos biológicos com proteínas leves responsivas, oferece uma nova estratégia para controlar dinamicamente as fermentações microbianas para a produção química e proteica1,2. A carga de vias metabólicas projetadas e a toxicidade de alguns intermediários e produtos muitas vezes prejudicam o crescimento celular3. Tais tensões podem levar à má acumulação de biomassa e à redução da produtividade3. Esse desafio pode ser enfrentado dividindo temporalmente as fermentações em uma fase de crescimento e produção, que dedicam recursos metabólicos ao acúmulo de biomassa ou à síntese do produto, respectivamente4. Recentemente, mostramos que a transição do crescimento para a produção nesta fermentação em duas fases pode ser induzida com mudanças nas condições de iluminação5,6,7. A alta sintonia, reversibilidade e ortogonalidade dos insumos leves8 oferecem vantagens únicas às fermentações controladas pela luz que são difíceis ou impossíveis de replicar com indutores químicos usados no controle dinâmico das fermentações convencionais em duas fases4,9,10,11.
A proteína EL222 responsiva de luz azul derivada de Erythrobacter litoralis tem sido usada para desenvolver vários circuitos optogenéticos para engenharia metabólica em Saccharomyces cerevisiae5,7,12,13. El222 contém um domínio sensor de tensão de oxigênio leve (LOV) que sofre uma mudança conformacional sobre a ativação da luz azul (465 nm), que permite que ele se ligue à sua sequência de DNA cognato (C120)13. Fundir EL222 ao domínio de ativação do VP16 viral (VP16-EL222) resulta em um fator de transcrição responsiva de luz azul que pode ativar reversivelmente a expressão genética em S. cerevisiae7 e outros organismos14 do promotor sintético PC120. Vários circuitos baseados no EL222 foram desenvolvidos e utilizados para a produção química em S. cerevisiae, como o sistema OptoEXP ativado pela luz básica7, no qual o gene de interesse é diretamente expresso a partir de PC120 (Figura 1A). No entanto, preocupações com a penetração da luz nas altas densidades celulares tipicamente encontradas na fase de produção de fermentações nos motivaram a desenvolver circuitos invertidos que são induzidos no escuro, como os circuitos OptoINVRT e OptoQ-INVRT (Figura 1B)5,7,13. Estes sistemas aproveitam os regulons galactose (GAL) ou quinic acid (Q) de S. cerevisiae e N. crassa, respectivamente, controlando seus repressores correspondentes (GAL80 e QS) com VP16-EL222, para reprimir a expressão genética na luz e induzi-la fortemente no escuro. A combinação de circuitos OptoEXP e OptoINVRT resulta em controle bidirecional da expressão genética, permitindo fermentações em duas fases nas quais a fase de crescimento é induzida com luz azul, e a fase de produção com escuridão (Figura 2A)5,7.
Usar a luz em vez de escuridão para induzir a expressão genética durante a fase de produção expandiria muito as capacidades de controles optogenéticos, mas também exigiria superar as limitações de penetração de luz das altas densidades celulares tipicamente encontradas nesta fase de fermentação. Para isso, desenvolvemos circuitos, conhecidos como OptoAMP e OptoQ-AMP, que amplificam a resposta transcricional à estimulação da luz azul. Esses circuitos usam mutantes do tipo selvagem ou hipersensíveis do VP16-EL222 para controlar a produção dos ativadores transcricionais Gal4p ou QF2 dos regulons GAL ou Q, respectivamente, alcançando maior sensibilidade e expressão genética mais forte com luz12,13 (Figura 1C). Os circuitos optoAMP podem alcançar indução de luz completa e homogênea em bioreatores de 5 L a uma densidade óptica (medida a 600 nm; OD600) valores de pelo menos 40 com apenas ~0,35% de iluminação (5% de dose leve em apenas ~7% da superfície a granel). Isso demonstra um maior grau de sensibilidade em comparação com o OptoEXP, que requer cerca de 100% de iluminação12. A capacidade de induzir efetivamente a expressão genética com luz em altas densidades celulares abre novas oportunidades para o controle dinâmico das fermentações. Isso inclui fermentações operacionais em mais de duas fases temporais, como fermentações trifásicas, nas quais as fases de crescimento, indução e produção são estabelecidas com cronogramas de luz únicos para otimizar a produção química (Figura 2B)12.
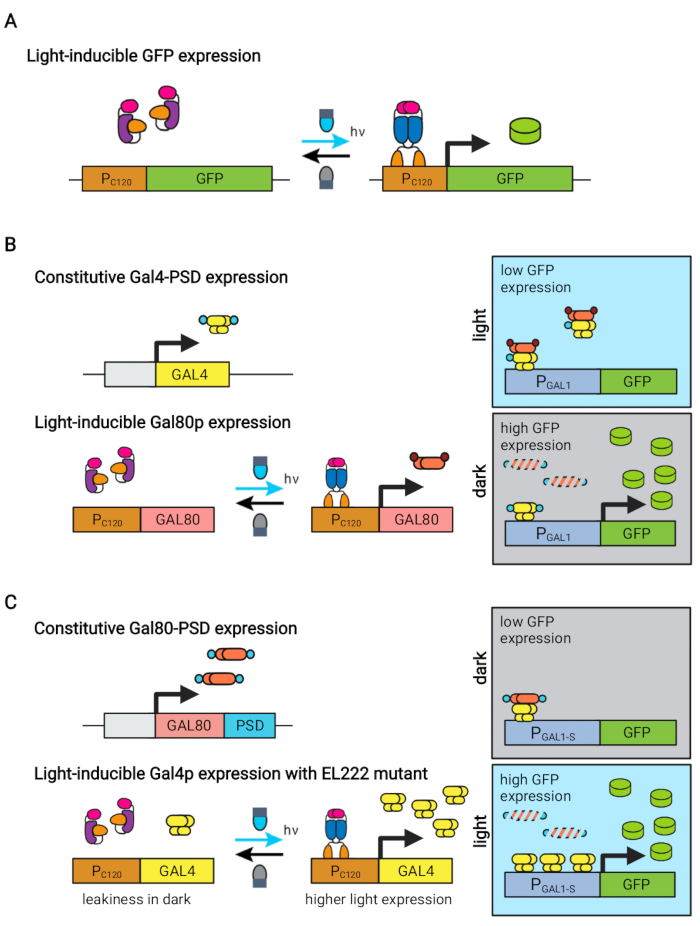
Figura 1: Circuitos optogenéticos para controle dinâmico de S. cerevisiae. Os circuitos OptoEXP, OptoINVRT e OptoAMP são baseados no sistema VP16-EL222 sensível à luz. (A) No circuito OptoEXP, a exposição à luz azul causa uma alteração conformacional e a dimerização do VP16-EL222, que expõe um domínio de vinculação de DNA e permite a transcrição do PC120. O número foi modificado de Zhao et al.7. (B) Os circuitos OptoINVRT aproveitam os regulons GAL (mostrado) ou Q para induzir a expressão no escuro. Nos circuitos baseados em GAL, VP16-EL222 e GAL4 são expressos constitutivamente, enquanto a expressão de drives PC120 do repressor GAL80 (em circuitos baseados em Q, GAL4 e GAL80 são substituídas por QF2 e QS, respectivamente, e um promotor sintético contendo QUAS é usado em vez de um promotor gal). À luz, Gal80p impede a ativação do gene de interesse do PGAL1. No escuro, GAL80 não é expresso e rapidamente degradado, fundindo-o a um domínio degron constitutivo (pequeno domínio marrom), que permite a ativação de PGAL1 por Gal4p. O número foi modificado de Zhao et al.5. (C) Os circuitos OptoAMP também usam VP16-EL222 para controlar os regulons GAL (mostrado) ou Q. Nestes circuitos, o repressor GAL80 (ou QS) é expresso e fundido constitutivamente e fundido a um degron sensível a fotos (pequeno domínio azul) garantindo uma repressão apertada no escuro. PC120 e uma expressão de controle mutante hipersensível VP16-EL222 de GAL4 (ou QF2) com luz, que ativa fortemente O PGAL1 (ou um promotor contendo QUAS) na luz. Os circuitos derivados de GAL podem usar formas projetadas de PGAL1, como PGAL1-M ou PGAL1-S, que aumentaram a atividade, bem como promotores do tipo selvagem controlados pelo regulon GAL (PGAL1, PGAL10, PGAL2, PGAL7). A figura foi modificada de Zhao et al.12. Clique aqui para ver uma versão maior desta figura.
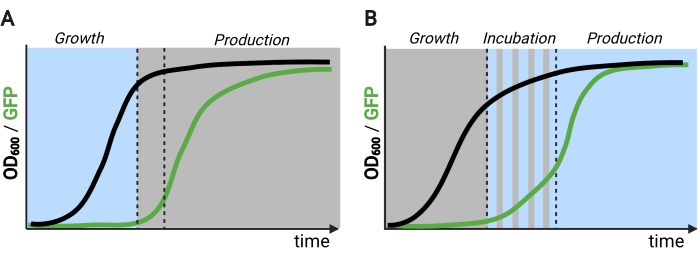
Figura 2: Fermentações de duas e três fases através do tempo. (A) Fermentações em duas fases operadas com circuitos invertidos consistem em uma fase de crescimento orientada pela luz e uma fase de produção escura. Na fase de crescimento, a biomassa se acumula à medida que a via de produção permanece reprimida. Ao atingir o OD600 desejado, as células são deslocadas para o escuro para ajustar metabolicamente antes de serem resuspendidas em novas mídias para a fase de produção. (B) Em um processo trifásica, as fases de crescimento, incubação e produção são definidas por cronogramas de luz únicos, que podem consistir em um período de crescimento escuro, incubação pulsada e fase de produção totalmente iluminada. Figura criada com Biorender. Clique aqui para ver uma versão maior desta figura.
Circuitos optogenéticos também foram desenvolvidos para controle dinâmico da produção química e proteica em E. coli. Os circuitos optoLAC controlam o repressor LacI bacteriano usando o circuito pDawn responsivo à luz, que é baseado no sistema de dois componentes YF1/FixJ6 (Figura 3). Semelhante ao OptoINVRT5, os circuitos OptoLAC são projetados para reprimir a expressão genética na luz e induzi-la no escuro. Os níveis de expressão usando circuitos OptoLAC podem corresponder ou exceder aqueles alcançados com indução isopropílico padrão β-d-1-thiogalactopyranoside (IPTG), mantendo assim a força da indução química ao mesmo tempo que oferece maior sintonia e reversibilidade6. Portanto, os circuitos OptoLAC permitem um controle optogenético eficaz para a engenharia metabólica em E. coli.
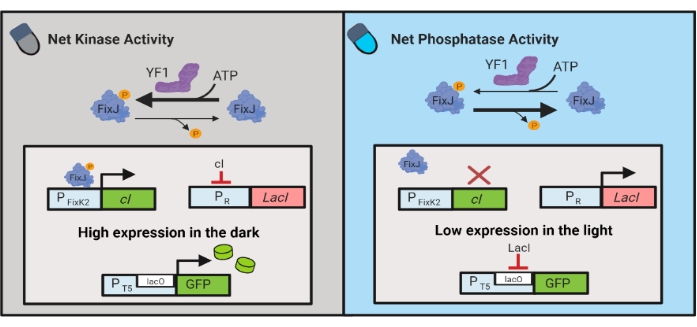
Figura 3: Circuitos OptoLAC para controle dinâmico de E. coli. Os circuitos OptoLAC adaptam o sistema pDawn e operon de lac para alcançar ativação no escuro e repressão à luz. No escuro, o YF1 fosforila fixJ, que então ativa o promotor PFixK2 para expressar o repressor cI . O repressor de IC impede a expressão do repressor lacI do promotor de PR , que permite a transcrição do gene de interesse de um promotor contendo lacO. Por outro lado, a luz azul reduz a atividade de quinase líquida YF1, invertendo a fosforilação fixJ e, portanto, a expressão cI , que deprime a expressão do lacI e impede a expressão do promotor contendo lacO. O número foi modificado de Lalwani et al.6. Clique aqui para ver uma versão maior desta figura.
Descrevemos aqui os protocolos básicos para fermentações controladas pela luz de S. cerevisiae e E. coli para produção química ou proteica. Para a levedura e as bactérias, primeiro focamos em fermentações com uma fase de crescimento impulsionada pela luz e uma fase de produção induzida pela escuridão habilitada pelos circuitos OptoINVRT e OptoLAC. Posteriormente, descrevemos um protocolo para uma fermentação trifásica (crescimento, indução, produção) controlada por luz habilitada pelos circuitos OptoAMP. Além disso, descrevemos como escalar fermentações optogeneticamente controladas de microplacos a bioreatores em escala de laboratório. Com este protocolo, pretendemos fornecer um guia completo e facilmente reprodutível para a realização de fermentações controladas por luz para produção química ou proteica.
Protocol
Representative Results
Discussion
O controle dinâmico tem sido aplicado há muito tempo para melhorar os rendimentos da engenharia metabólica e da produção de proteínas recombinantes4. Mudanças na expressão enzimática são mais tipicamente implementadas usando indutores químicos como IPTG21, galactose22 e tetraciclina23, mas também foram mediadas usando condições de processo como temperatura e pH. O controle optogenético da expressão genética el…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Esta pesquisa foi apoiada pelo Departamento de Energia dos EUA, Escritório de Ciência, Office of Biological and Environmental Research Award Número DE-SC0019363, o NSF CAREER Award CBET-1751840, The Pew Charitable Trusts e o Camille Dreyfus Teacher-Scholar Award.
Materials
| Light-controlled chemical production using S. cerevisiae | |||
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Agar powder | Thermo Fisher Scientific | 303991049 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| EZ-L439 OptoINVRT7 Plasmid | N/A | N/A | See Reference 1 |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| PmeI | New England Biolabs | R0560L | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| Replica-plating device | Thomas Scientific | F37848-0000 | |
| Replica-plating pads | Sunrise Science Products | 3005-012 | |
| SC-His powder | Sunrise Science Products | 1303-030 | |
| SC Complete powder | Sunrise Science Products | 1459-100 | |
| Sterile sealing film | Excel Scientific | STR-SEAL-PLT | |
| YPD agar plates | VWR | 100217-054 | |
| Zeocin | Thermo Fisher Scientific | R25005 | |
| Light-controlled protein production using E. coli | |||
| 6X SDS Sample Buffer | Cepham Life Sciences | 10502 | |
| 12% Acrylamide protein gels | Thermo Fisher Scientific | NP0341BOX | |
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| Coomassie Brilliant Blue G-250 | Thermo Fisher Scientific | 20279 | |
| Electrophoresis cell | Bio-Rad | 1658004 | |
| Electrophoresis power supply | Bio-Rad | 1645050 | |
| LB broth (Miller) | Fisher Scientific | BP97235 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NaCl | Thomas Scientific | SX0425-1 | |
| OptoLAC plasmids | N/A | N/A | See Reference 2 |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Thermomixer | Eppendorf | 5382000015 | |
| Tris base | Fisher Scientific | BP1521 | |
| Three-phase fermentation using S. cerevisiae | |||
| Same materials as "Light-controlled chemical production using S. cerevisiae" protocol plus the following: | |||
| EZ-L580 OptoAMP4 Plasmid | N/A | N/A | See Reference 10 |
| Chemical production in a light-controlled bioreactor | |||
| Aluminum foil | Reynolds | B004NG90YO | |
| Antifoam | Sigma-Aldrich | A8311 | |
| Bioreactor with control station | Eppendorf | B120110001 | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Bleach | VWR Scientific | 89501-620 (CS) | |
| Blue LED panel | HQRP | 884667106091218 | |
| BPT tubing | Fisher Scientific | 14-170-15 | |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Hydrochloric acid (HCl) | Fisher Scientific | 7647-01-0 | |
| M9 Minimal Salts | Thermo Fisher Scientific | A1374401 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NH4OH Solution | Sigma-Aldrich | I0503-1VL | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SC Complete powder | Sunrise Science Products | 1459-100 |
Riferimenti
- Figueroa, D., Rojas, V., Romero, A., Larrondo, L. F., Salinas, F. The rise and shine of yeast optogenetics. Yeast. 38 (2), 131-146 (2021).
- Pouzet, S., et al. The promise of optogenetics for bioproduction: Dynamic control strategies and scale-up instruments. Bioingegneria. 7 (4), 151 (2020).
- Venayak, N., Anesiadis, N., Cluett, W. R., Mahadevan, R. Engineering metabolism through dynamic control. Current Opinion in Biotechnology. 34, 142-152 (2015).
- Lalwani, M. A., Zhao, E. M., Avalos, J. L. Current and future modalities of dynamic control in metabolic engineering. Current Opinion in Biotechnology. 52, 56-65 (2018).
- Zhao, E. M., et al. Design and characterization of rapid optogenetic circuits for dynamic control in yeast metabolic engineering. ACS Synthetic Biology. 9 (12), 3254-3266 (2020).
- Lalwani, M. A., et al. Optogenetic control of the lac operon for bacterial chemical and protein production. Nature Chemical Biology. 17 (1), 71-79 (2021).
- Zhao, E. M., et al. Optogenetic regulation of engineered cellular metabolism for microbial chemical production. Nature. 555 (7698), 683-687 (2018).
- Baumschlager, A., Khammash, M. Synthetic biological approaches for optogenetics and tools for transcriptional light-control in bacteria. Advanced Biology. 5 (5), 2000256 (2021).
- Dvorak, P., et al. Exacerbation of substrate toxicity by IPTG in Escherichia coli BL21(DE3) carrying a synthetic metabolic pathway. Microbial Cell Factories. 14, 201 (2015).
- Hartline, C. J., Schmitz, A. C., Han, Y., Zhang, F. Dynamic control in metabolic engineering: Theories, tools, and applications. Metabolic Engineering. 63, 126-140 (2021).
- Ni, C., Dinh, C. V., Prather, K. L. J. Dynamic control of metabolism. Annual Review of Chemical and Biomolecular Engineering. 12, 519-560 (2021).
- Zhao, E. M., et al. Optogenetic amplification circuits for light-induced metabolic control. ACS Synthetic Biology. 10 (5), 1143-1154 (2021).
- Lalwani, M. A., Zhao, E. M., Wegner, S. A., Avalos, J. L. The Neurospora crassa Inducible Q System Enables Simultaneous Optogenetic Amplification and Inversion in Saccharomyces cerevisiae for Bidirectional Control of Gene Expression. ACS Synthetic Biology. 10 (8), 2060-2075 (2021).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature Chemical Biology. 10 (3), 196-202 (2014).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Marx, H., Mecklenbräuker, A., Gasser, B., Sauer, M., Mattanovich, D. Directed gene copy number amplification in Pichia pastoris by vector integration into the ribosomal DNA locus. FEMS Yeast Research. 9 (8), 1260-1270 (2009).
- Nordén, K., et al. Increasing gene dosage greatly enhances recombinant expression of aquaporins in Pichia pastoris. BMC Biotechnology. 11, 47 (2011).
- Zhao, E. M., et al. Light-based control of metabolic flux through assembly of synthetic organelles. Nature Chemical Biology. 15 (6), 589-597 (2019).
- Dowee, W. J., Miller, J. F., Ragsdale, C. W. High efficiency transformation of E. coli by high voltage electroporation. Nucleic Acids Research. 16 (13), 6127-6145 (1988).
- Zhou, K., Edgar, S., Stephanopoulos, G. Engineering microbes to synthesize plant isoprenoids. Methods in Enzymology. 575, 225-245 (2016).
- Arfman, N., Worrell, V., Ingram, L. O. Use of the tac promoter and lacI(q) for the controlled expression of Zymomonas mobilis fermentative genes in Escherichia coli and Zymomonas mobilis. Journal of Bacteriology. 174 (22), 7370-7378 (1992).
- Steen, E. J., et al. Metabolic engineering of Saccharomyces cerevisiae for the production of n-butanol. Microbial Cell Factories. 7 (1), 1-8 (2008).
- Tan, S. Z., Manchester, S., Prather, K. L. J. Controlling central carbon metabolism for improved pathway yields in Saccharomyces cerevisiae. ACS Synthetic Biology. 5 (2), 116-124 (2015).
- Jayaraman, P., et al. Blue light-mediated transcriptional activation and repression of gene expression in bacteria. Nucleic Acids Research. 44 (14), 6994 (2016).
- Fernandez-Rodriguez, J., Moser, F., Song, M., Voigt, C. A. Engineering RGB color vision into Escherichia coli. Nature Chemical Biology. 13 (7), 706-708 (2017).
- Ding, Q., et al. Light-powered Escherichia coli cell division for chemical production. Nature Communications. 11 (1), 1-14 (2020).
- Senoo, S., Tandar, S. T., Kitamura, S., Toya, Y., Shimizu, H. Light-inducible flux control of triosephosphate isomerase on glycolysis in Escherichia coli. Biotechnology and Bioengineering. 116 (12), 3292-3300 (2019).
- Ramakrishnan, P., Tabor, J. J. Repurposing synechocystis PCC6803 UirS-UirR as a UV-violet/green photoreversible transcriptional regulatory tool in E. Coli. ACS Synthetic Biology. 5 (7), 733-740 (2016).
- Tabor, J. J., Levskaya, A., Voigt, C. A. Multichromatic control of gene expression in escherichia coli. Journal of Molecular Biology. 405 (2), 315-324 (2011).
- Stewart, C. J., McClean, M. N. Design and implementation of an automated illuminating, culturing, and sampling system for microbial optogenetic applications. Journal of Visualized Experiments:JoVE. (120), e54894 (2017).
- Grødem, E. O. S., Sweeney, K., McClean, M. N. Automated calibration of optoPlate LEDs to reduce light dose variation in optogenetic experiments. BioTechniques. 69 (4), 313-316 (2020).
- Gerhardt, K. P., et al. An open-hardware platform for optogenetics and photobiology. Scientific Reports. 6, (2016).
- Bugaj, L. J., Lim, W. A. High-throughput multicolor optogenetics in microwell plates. Nature Protocols. 14 (7), 2205-2228 (2019).
- Steel, H., Habgood, R., Kelly, C., Papachristodoulou, A. In situ characterisation and manipulation of biological systems with Chi.Bio. PLoS Biology. 18 (7), (2020).
- Carrasco-López, C., García-Echauri, S. A., Kichuk, T., Avalos, J. L. Optogenetics and biosensors set the stage for metabolic cybergenetics. Current Opinion in Biotechnology. 65, 296-309 (2020).
- Milias-Argeitis, A., Rullan, M., Aoki, S. K., Buchmann, P., Khammash, M. Automated optogenetic feedback control for precise and robust regulation of gene expression and cell growth. Nature Communications. 7 (1), 1-11 (2016).
- Melendez, J., et al. Real-time optogenetic control of intracellular protein concentration in microbial cell cultures. Integrative Biology: Quantitative Biosciences From Nano to Macro. 6 (3), 366-372 (2014).
- Milias-Argeitis, A., et al. In silico feedback for in vivo regulation of a gene expression circuit. Nature Biotechnology. 29 (12), 1114-1116 (2011).
- Castillo-Hair, S. M., Baerman, E. A., Fujita, M., Igoshin, O. A., Tabor, J. J. Optogenetic control of Bacillus subtilis gene expression. Nature Communications. 10 (1), 1-11 (2019).
- Xia, A., et al. Optogenetic modification of pseudomonas aeruginosa enables controllable twitching motility and host infection. ACS Synthetic Biology. 10 (3), 531-541 (2021).
- Pu, L., Yang, S., Xia, A., Jin, F. Optogenetics manipulation enables prevention of biofilm formation of engineered pseudomonas aeruginosa on surfaces. ACS Synthetic Biology. 7 (1), 200-208 (2018).

