Ljuskontrollerade jäsningar för mikrobiell kemisk och proteinproduktion
Summary
Optogenetisk kontroll av mikrobiell metabolism erbjuder flexibel dynamisk kontroll över jäsningsprocesser. Protokollet här visar hur man ställer in blå ljusreglerade jäsningar för kemisk och proteinproduktion i olika volymetriska skalor.
Abstract
Mikrobiella cellfabriker erbjuder ett hållbart alternativ för att producera kemikalier och rekombinanta proteiner från förnybara råvaror. Överbelastning av en mikroorganism med genetiska modifieringar kan dock minska värdens kondition och produktivitet. Detta problem kan övervinnas genom att använda dynamisk kontroll: induktiva uttryck av enzymer och vägar, vanligtvis med kemiska eller näringsbaserade tillsatser, för att balansera cellulär tillväxt och produktion. Optogenetik erbjuder en icke-invasiv, mycket tunable och reversibel metod för dynamisk reglering av genuttryck. Här beskriver vi hur man ställer in ljuskontrollerade jäsningar av konstruerade Escherichia coli och Saccharomyces cerevisiae för produktion av kemikalier eller rekombinanta proteiner. Vi diskuterar hur man applicerar ljus vid utvalda tidpunkter och doser för att frikoppla mikrobiell tillväxt och produktion för förbättrad jäsningskontroll och produktivitet, samt de viktigaste optimeringsövervägandena för bästa resultat. Dessutom beskriver vi hur man implementerar ljuskontroller för bioreaktorexperiment i labbskala. Dessa protokoll underlättar antagandet av optogenetiska kontroller i konstruerade mikroorganismer för förbättrad jäsningsprestanda.
Introduction
Optogenetik, bekämpning av biologiska processer med ljuskänsliga proteiner, erbjuder en ny strategi för att dynamiskt kontrollera mikrobiella jäsningar för kemisk och proteinproduktion1,2. Bördan av konstruerade metaboliska vägar och toxiciteten hos vissa intermediärer och produkter försämrar ofta celltillväxten3. Sådana påfrestningar kan leda till dålig ackumulering av biomassa och minskad produktivitet3. Denna utmaning kan hanteras genom att tidsmässigt dela upp jäsningar i en tillväxt- och produktionsfas, som ägnar metaboliska resurser åt biomassaackumulering respektive produktsyntes4. Vi visade nyligen att övergången från tillväxt till produktion i denna tvåfasfermentering kan induceras med förändringar i belysningsförhållandena5,6,7. Den höga omtunningsbarheten, reversibiliteten och ortogonaliteten hos ljusingångar8 erbjuder unika fördelar för ljuskontrollerade jäsningar som är svåra eller omöjliga att replikera med kemiska inducerare som används vid dynamisk kontroll av konventionella tvåfasfermenteringar4,9,10,11.
Det blåljus responsiva EL222-proteinet som härrör från Erythrobacter litoralis har använts för att utveckla flera optogenetiska kretsar för metabolisk teknik i Saccharomyces cerevisiae5,7,12,13. EL222 innehåller en ljus-syre-spänningssensor (LOV) domän som genomgår en konformationell förskjutning vid blå ljusaktivering (465 nm), vilket gör att den kan binda till sin cognate DNA-sekvens (C120)13. Att smälta EL222 till den virala VP16 aktiveringsdomänen (VP16-EL222) resulterar i en blåljusresponsiv transkriptionsfaktor som på ett reversibelt sätt kan aktivera genuttryck i S. cerevisiae7 och andra organismer14 från den syntetiska promotorn PC120. Flera kretsar baserade på EL222 har utvecklats och använts för kemisk produktion i S. cerevisiae, såsom det grundläggande ljusaktiverade OptoEXP-systemet7, där den av intressegenen uttrycks direkt från PC120 (figur 1A). Oron för lätt penetration vid de höga celltätheter som vanligtvis påträffas i produktionsfasen av jäsningar motiverade oss dock att utveckla inverterade kretsar som induceras i mörkret, såsom OptoINVRT- och OptoQ-INVRT-kretsarna (figur 1B)5,7,13. Dessa system utnyttjar galaktos (GAL) eller kininsyra (Q) reguloner från S. cerevisiae respektive N. crassa, kontrollerar deras motsvarande undertryckare (GAL80 och QS) med VP16-EL222, för att undertrycka genuttryck i ljuset och starkt inducera det i mörkret. Kombinationen av OptoEXP- och OptoINVRT-kretsar resulterar i dubbelriktad kontroll av genuttryck, vilket möjliggör tvåfasfermentering där tillväxtfasen induceras med blått ljus och produktionsfasen med mörker (figur 2A)5,7.
Att använda ljus istället för mörker för att inducera genuttryck under produktionsfasen skulle kraftigt utöka kapaciteten hos optogenetiska kontroller men skulle också kräva att övervinna de lätta penetrationsbegränsningarna för de höga celltätheter som vanligtvis uppstår i denna fas av jäsning. För detta ändamål har vi utvecklat kretsar, kända som OptoAMP och OptoQ-AMP, som förstärker transkriptionssvaret på blå ljusstimulering. Dessa kretsar använder vilda eller överkänsliga mutanter av VP16-EL222 för att kontrollera produktionen av transkriptionsaktivatorerna Gal4p respektive QF2 av GAL- respektive Q-regulonerna, vilket ger ökad känslighet och starkare genuttryck med ljus12,13 (figur 1C). OptoAMP-kretsar kan uppnå fullständig och homogen ljusinduktion i 5 L bioreaktorer med optisk densitet (mätt vid 600 nm; OD600) värden på minst 40 med endast ~ 0,35% av belysningen (5% ljusdos på endast ~ 7% av bulkytan). Detta visar en högre grad av känslighet jämfört med OptoEXP, vilket kräver nära 100% belysning12. Förmågan att effektivt inducera genuttryck med ljus vid högcellstäthet öppnar nya möjligheter till dynamisk kontroll av jäsningar. Detta inkluderar drift av jäsningar i mer än två temporala faser, såsom trefasfermensningar, där tillväxt-, induktions- och produktionsfaser upprättas med unika ljusscheman för att optimera kemisk produktion (figur 2B)12.
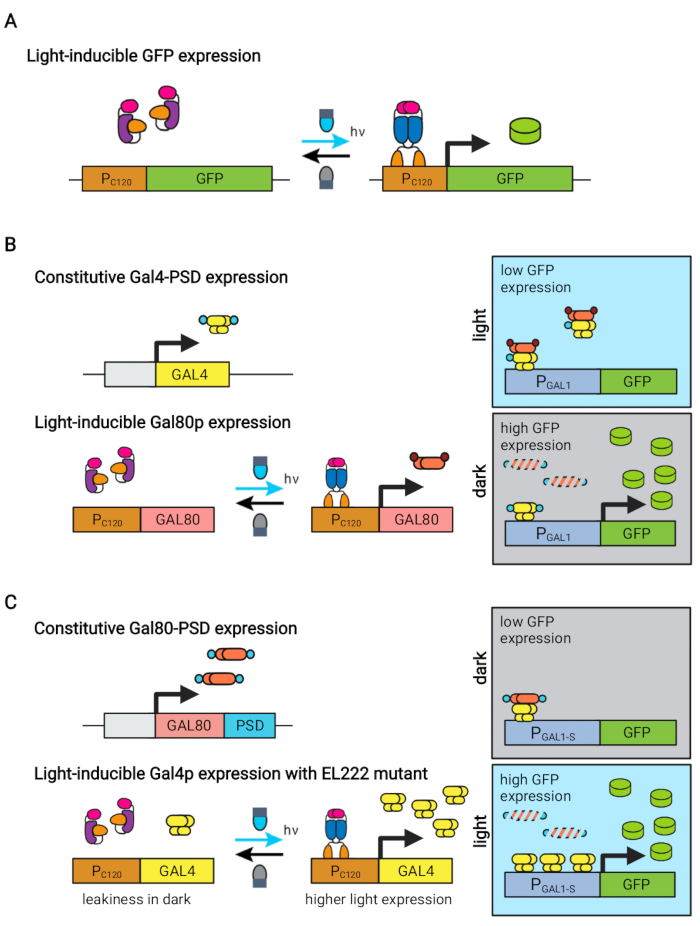
Figur 1: Optogenetiska kretsar för dynamisk kontroll av S. cerevisiae. OptoEXP-, OptoINVRT- och OptoAMP-kretsarna är baserade på det ljuskänsliga VP16-EL222-systemet. (A) I OptoEXP-kretsen orsakar exponering för blått ljus en konformationsförändring och dimerisering av VP16-EL222, som exponerar en DNA-bindande domän och möjliggör transkription från PC120. Figuren har ändrats från Zhao et al.7. (B) OptoINVRT-kretsar utnyttjar GAL -regulonerna (visas) eller Q för att framkalla uttryck i mörker. I GAL-baserade kretsar uttrycks VP16-EL222 och GAL4 konstituerande, medan PC120-frekvensomtryck av GAL80-tryckorn (i Q-baserade kretsar ersätts GAL4 och GAL80 av QF2 respektive QS, och en syntetisk QUAS-innehållande promotor används istället för en GAL-promotor). I ljuset förhindrar Gal80p aktivering av den av intressegenen från PGAL1. I mörkret uttrycks inte GAL80 och bryts snabbt ned genom att den smälter till en konstituerande degrondomän (liten brun domän), vilket möjliggör aktivering av PGAL1 av Gal4p. Figuren har ändrats från Zhao et al.5. (C) OptoAMP-kretsar använder också VP16-EL222 för att styra GAL (visas) eller Q-regulons. I dessa kretsar uttrycks och smälts GAL80-tryckpressorn (eller QS) konstituerande till en fotokänslig degron (liten blå domän) som säkerställer hårt förtryck i mörkret. PC120 och ett överkänsligt VP16-EL222 mutant kontroll uttryck av GAL4 (eller QF2) med ljus, som starkt aktiverar PGAL1 (eller en QUAS-innehållande promotor) i ljuset. GAL-härledda kretsar kan använda konstruerade former av PGAL1, såsom PGAL1-M eller PGAL1-S, som har ökad aktivitet, liksom vilda promotorer som styrs av GAL-regulon (PGAL1, PGAL10, PGAL2, PGAL7). Figuren har ändrats från Zhao et al.12. Klicka här för att se en större version av denna siffra.
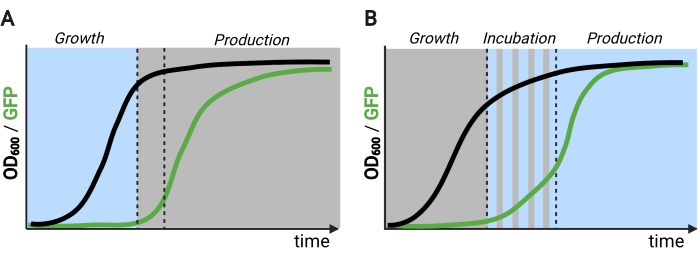
Figur 2: Två- och trefasfermensningar genom tiden. (A) Tvåfasfermensningar som drivs med inverterade kretsar består av en ljusdriven tillväxtfas och en mörk produktionsfas. I tillväxtfasen ackumuleras biomassa när produktionsvägen förblir undertryckt. När man når önskad OD600 flyttas cellerna till mörkret för att metaboliskt justera innan de återanvänds i färska medier för produktionsfasen. (B) I en trefasprocess definieras tillväxt-, inkubations- och produktionsfaserna av unika ljusscheman, som kan bestå av en mörk tillväxtperiod, pulsad inkubation och fullt upplyst produktionsfas. Figur skapad med Biorender. Klicka här för att se en större version av den här figuren.
Optogenetiska kretsar har också utvecklats för dynamisk kontroll av kemisk och proteinproduktion i E. coli. OptoLAC-kretsar styr bakterien LacI-tryckorn med hjälp av den ljuskänsliga pDawn-kretsen, som är baserad på YF1/FixJ tvåkomponentsystem6 (bild 3). I likhet med OptoINVRT5 är OptoLAC-kretsar utformade för att undertrycka genuttryck i ljuset och inducera det i mörkret. Uttrycksnivåer med optoLAC-kretsar kan matcha eller överträffa de som uppnås med standard isopropyl β-d-1-tiogalactopyranoside (IPTG) induktion, vilket bibehåller styrkan av kemisk induktion samtidigt som den erbjuder förbättrad bedövningsförmåga och reversibilitet6. Därför möjliggör OptoLAC-kretsar effektiv optogenetisk kontroll för metabolisk teknik i E. coli.
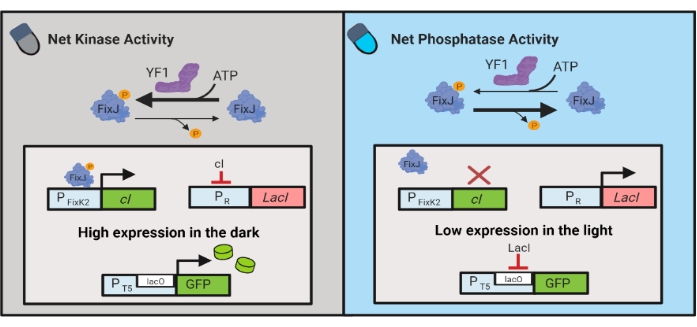
Bild 3: OptoLAC-kretsar för dynamisk styrning av E. coli. OptoLAC-kretsarna anpassar pDawn-systemet och lac operon för att uppnå aktivering i mörker och förtryck i ljuset. I mörkret, YF1 fosforylater FixJ, som sedan aktiverar PFixK2 promotorn för att uttrycka cI-undertryckaren . cI-trycksättaren förhindrar uttryck av lacI-tryckorn från PR-promotorn , vilket tillåter transkription av den av intressegenen från en lacO-innehållande promotor. Omvänt minskar blått ljus YF1 nettokinasaktivitet, vänder FixJ fosforylering och därmed cI uttryck, som dereprimerar uttryck av lacI och förhindrar uttryck från lacO-innehållande promotorn. Figuren har ändrats från Lalwani et al.6. Klicka här för att se en större version av den här figuren.
Vi beskriver här de grundläggande protokollen för ljuskontrollerade jäsningar av S. cerevisiae och E. coli för kemisk eller proteinproduktion. För både jäst och bakterier fokuserar vi först på jäsningar med en ljusdriven tillväxtfas och en mörkinducerad produktionsfas som möjliggörs av OptoINVRT- och OptoLAC-kretsar. Därefter beskriver vi ett protokoll för en trefas (tillväxt, induktion, produktion) ljusstyrd jäsning som möjliggörs av OptoAMP-kretsar. Dessutom beskriver vi hur man kan skala upp optogenetiskt kontrollerade jäsningar från mikroplattor till laboratorieskala bioreaktorer. Med detta protokoll strävar vi efter att tillhandahålla en komplett och lätt reproducerbar guide för att utföra ljuskontrollerade jäsningar för kemisk eller proteinproduktion.
Protocol
Representative Results
Discussion
Dynamisk kontroll har länge tillämpats för att förbättra avkastningen för metabolisk teknik och rekombinant proteinproduktion4. Förändringar i enzymatiska uttryck genomförs oftast med hjälp av kemiska inducerare som IPTG21, galaktos22 och tetracyklin23, men har också medierats med processförhållanden som temperatur och pH. Optogenetisk kontroll av genuttryck eliminerar behovet av förändringar i jäsningsparametr…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Denna forskning stöddes av U.S. Department of Energy, Office of Science, Office of Biological and Environmental Research Award Number DE-SC0019363, NSF CAREER Award CBET-1751840, The Pew Charitable Trusts och Camille Dreyfus Teacher-Scholar Award.
Materials
| Light-controlled chemical production using S. cerevisiae | |||
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Agar powder | Thermo Fisher Scientific | 303991049 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| EZ-L439 OptoINVRT7 Plasmid | N/A | N/A | See Reference 1 |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| PmeI | New England Biolabs | R0560L | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| Replica-plating device | Thomas Scientific | F37848-0000 | |
| Replica-plating pads | Sunrise Science Products | 3005-012 | |
| SC-His powder | Sunrise Science Products | 1303-030 | |
| SC Complete powder | Sunrise Science Products | 1459-100 | |
| Sterile sealing film | Excel Scientific | STR-SEAL-PLT | |
| YPD agar plates | VWR | 100217-054 | |
| Zeocin | Thermo Fisher Scientific | R25005 | |
| Light-controlled protein production using E. coli | |||
| 6X SDS Sample Buffer | Cepham Life Sciences | 10502 | |
| 12% Acrylamide protein gels | Thermo Fisher Scientific | NP0341BOX | |
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| Coomassie Brilliant Blue G-250 | Thermo Fisher Scientific | 20279 | |
| Electrophoresis cell | Bio-Rad | 1658004 | |
| Electrophoresis power supply | Bio-Rad | 1645050 | |
| LB broth (Miller) | Fisher Scientific | BP97235 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NaCl | Thomas Scientific | SX0425-1 | |
| OptoLAC plasmids | N/A | N/A | See Reference 2 |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Thermomixer | Eppendorf | 5382000015 | |
| Tris base | Fisher Scientific | BP1521 | |
| Three-phase fermentation using S. cerevisiae | |||
| Same materials as "Light-controlled chemical production using S. cerevisiae" protocol plus the following: | |||
| EZ-L580 OptoAMP4 Plasmid | N/A | N/A | See Reference 10 |
| Chemical production in a light-controlled bioreactor | |||
| Aluminum foil | Reynolds | B004NG90YO | |
| Antifoam | Sigma-Aldrich | A8311 | |
| Bioreactor with control station | Eppendorf | B120110001 | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Bleach | VWR Scientific | 89501-620 (CS) | |
| Blue LED panel | HQRP | 884667106091218 | |
| BPT tubing | Fisher Scientific | 14-170-15 | |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Hydrochloric acid (HCl) | Fisher Scientific | 7647-01-0 | |
| M9 Minimal Salts | Thermo Fisher Scientific | A1374401 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NH4OH Solution | Sigma-Aldrich | I0503-1VL | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SC Complete powder | Sunrise Science Products | 1459-100 |
Riferimenti
- Figueroa, D., Rojas, V., Romero, A., Larrondo, L. F., Salinas, F. The rise and shine of yeast optogenetics. Yeast. 38 (2), 131-146 (2021).
- Pouzet, S., et al. The promise of optogenetics for bioproduction: Dynamic control strategies and scale-up instruments. Bioingegneria. 7 (4), 151 (2020).
- Venayak, N., Anesiadis, N., Cluett, W. R., Mahadevan, R. Engineering metabolism through dynamic control. Current Opinion in Biotechnology. 34, 142-152 (2015).
- Lalwani, M. A., Zhao, E. M., Avalos, J. L. Current and future modalities of dynamic control in metabolic engineering. Current Opinion in Biotechnology. 52, 56-65 (2018).
- Zhao, E. M., et al. Design and characterization of rapid optogenetic circuits for dynamic control in yeast metabolic engineering. ACS Synthetic Biology. 9 (12), 3254-3266 (2020).
- Lalwani, M. A., et al. Optogenetic control of the lac operon for bacterial chemical and protein production. Nature Chemical Biology. 17 (1), 71-79 (2021).
- Zhao, E. M., et al. Optogenetic regulation of engineered cellular metabolism for microbial chemical production. Nature. 555 (7698), 683-687 (2018).
- Baumschlager, A., Khammash, M. Synthetic biological approaches for optogenetics and tools for transcriptional light-control in bacteria. Advanced Biology. 5 (5), 2000256 (2021).
- Dvorak, P., et al. Exacerbation of substrate toxicity by IPTG in Escherichia coli BL21(DE3) carrying a synthetic metabolic pathway. Microbial Cell Factories. 14, 201 (2015).
- Hartline, C. J., Schmitz, A. C., Han, Y., Zhang, F. Dynamic control in metabolic engineering: Theories, tools, and applications. Metabolic Engineering. 63, 126-140 (2021).
- Ni, C., Dinh, C. V., Prather, K. L. J. Dynamic control of metabolism. Annual Review of Chemical and Biomolecular Engineering. 12, 519-560 (2021).
- Zhao, E. M., et al. Optogenetic amplification circuits for light-induced metabolic control. ACS Synthetic Biology. 10 (5), 1143-1154 (2021).
- Lalwani, M. A., Zhao, E. M., Wegner, S. A., Avalos, J. L. The Neurospora crassa Inducible Q System Enables Simultaneous Optogenetic Amplification and Inversion in Saccharomyces cerevisiae for Bidirectional Control of Gene Expression. ACS Synthetic Biology. 10 (8), 2060-2075 (2021).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature Chemical Biology. 10 (3), 196-202 (2014).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Marx, H., Mecklenbräuker, A., Gasser, B., Sauer, M., Mattanovich, D. Directed gene copy number amplification in Pichia pastoris by vector integration into the ribosomal DNA locus. FEMS Yeast Research. 9 (8), 1260-1270 (2009).
- Nordén, K., et al. Increasing gene dosage greatly enhances recombinant expression of aquaporins in Pichia pastoris. BMC Biotechnology. 11, 47 (2011).
- Zhao, E. M., et al. Light-based control of metabolic flux through assembly of synthetic organelles. Nature Chemical Biology. 15 (6), 589-597 (2019).
- Dowee, W. J., Miller, J. F., Ragsdale, C. W. High efficiency transformation of E. coli by high voltage electroporation. Nucleic Acids Research. 16 (13), 6127-6145 (1988).
- Zhou, K., Edgar, S., Stephanopoulos, G. Engineering microbes to synthesize plant isoprenoids. Methods in Enzymology. 575, 225-245 (2016).
- Arfman, N., Worrell, V., Ingram, L. O. Use of the tac promoter and lacI(q) for the controlled expression of Zymomonas mobilis fermentative genes in Escherichia coli and Zymomonas mobilis. Journal of Bacteriology. 174 (22), 7370-7378 (1992).
- Steen, E. J., et al. Metabolic engineering of Saccharomyces cerevisiae for the production of n-butanol. Microbial Cell Factories. 7 (1), 1-8 (2008).
- Tan, S. Z., Manchester, S., Prather, K. L. J. Controlling central carbon metabolism for improved pathway yields in Saccharomyces cerevisiae. ACS Synthetic Biology. 5 (2), 116-124 (2015).
- Jayaraman, P., et al. Blue light-mediated transcriptional activation and repression of gene expression in bacteria. Nucleic Acids Research. 44 (14), 6994 (2016).
- Fernandez-Rodriguez, J., Moser, F., Song, M., Voigt, C. A. Engineering RGB color vision into Escherichia coli. Nature Chemical Biology. 13 (7), 706-708 (2017).
- Ding, Q., et al. Light-powered Escherichia coli cell division for chemical production. Nature Communications. 11 (1), 1-14 (2020).
- Senoo, S., Tandar, S. T., Kitamura, S., Toya, Y., Shimizu, H. Light-inducible flux control of triosephosphate isomerase on glycolysis in Escherichia coli. Biotechnology and Bioengineering. 116 (12), 3292-3300 (2019).
- Ramakrishnan, P., Tabor, J. J. Repurposing synechocystis PCC6803 UirS-UirR as a UV-violet/green photoreversible transcriptional regulatory tool in E. Coli. ACS Synthetic Biology. 5 (7), 733-740 (2016).
- Tabor, J. J., Levskaya, A., Voigt, C. A. Multichromatic control of gene expression in escherichia coli. Journal of Molecular Biology. 405 (2), 315-324 (2011).
- Stewart, C. J., McClean, M. N. Design and implementation of an automated illuminating, culturing, and sampling system for microbial optogenetic applications. Journal of Visualized Experiments:JoVE. (120), e54894 (2017).
- Grødem, E. O. S., Sweeney, K., McClean, M. N. Automated calibration of optoPlate LEDs to reduce light dose variation in optogenetic experiments. BioTechniques. 69 (4), 313-316 (2020).
- Gerhardt, K. P., et al. An open-hardware platform for optogenetics and photobiology. Scientific Reports. 6, (2016).
- Bugaj, L. J., Lim, W. A. High-throughput multicolor optogenetics in microwell plates. Nature Protocols. 14 (7), 2205-2228 (2019).
- Steel, H., Habgood, R., Kelly, C., Papachristodoulou, A. In situ characterisation and manipulation of biological systems with Chi.Bio. PLoS Biology. 18 (7), (2020).
- Carrasco-López, C., García-Echauri, S. A., Kichuk, T., Avalos, J. L. Optogenetics and biosensors set the stage for metabolic cybergenetics. Current Opinion in Biotechnology. 65, 296-309 (2020).
- Milias-Argeitis, A., Rullan, M., Aoki, S. K., Buchmann, P., Khammash, M. Automated optogenetic feedback control for precise and robust regulation of gene expression and cell growth. Nature Communications. 7 (1), 1-11 (2016).
- Melendez, J., et al. Real-time optogenetic control of intracellular protein concentration in microbial cell cultures. Integrative Biology: Quantitative Biosciences From Nano to Macro. 6 (3), 366-372 (2014).
- Milias-Argeitis, A., et al. In silico feedback for in vivo regulation of a gene expression circuit. Nature Biotechnology. 29 (12), 1114-1116 (2011).
- Castillo-Hair, S. M., Baerman, E. A., Fujita, M., Igoshin, O. A., Tabor, J. J. Optogenetic control of Bacillus subtilis gene expression. Nature Communications. 10 (1), 1-11 (2019).
- Xia, A., et al. Optogenetic modification of pseudomonas aeruginosa enables controllable twitching motility and host infection. ACS Synthetic Biology. 10 (3), 531-541 (2021).
- Pu, L., Yang, S., Xia, A., Jin, F. Optogenetics manipulation enables prevention of biofilm formation of engineered pseudomonas aeruginosa on surfaces. ACS Synthetic Biology. 7 (1), 200-208 (2018).

