Surface Enhanced Raman Spectroscopy detección de biomoléculas mediante EBL Fabricados Nanoestructurados Sustratos
Summary
We describe the fabrication and characterization of nano-biological systems interfacing nanostructured substrates with immobilized proteins and aptamers. The relevant experimental steps involving lithographic fabrication of nanostructured substrates, bio-functionalization, and surface-enhanced Raman spectroscopy (SERS) characterization, are reported. SERS detection of surface-immobilized proteins, and probing of protein-ligand and aptamer-ligand binding is demonstrated.
Abstract
Se informa de fabricación y caracterización de sistemas nano-biológica conjugadas interfaz nanoestructuras metálicas sobre soportes sólidos con biomoléculas inmovilizadas. La secuencia completa de pasos experimentales pertinentes se describe, que implica la fabricación de los sustratos nanoestructurados utilizando litografía de haz de electrones, la inmovilización de biomoléculas sobre los sustratos, y su caracterización utilizando una mayor superficie de la espectroscopia Raman (SERS). Se utilizan tres diseños diferentes de sistemas de nano-biológica, incluyendo la proteína A, proteína de unión a glucosa, y un aptámero de unión a ADN de la dopamina. En los dos últimos casos, la unión de ligandos respectivos, D-glucosa y la dopamina, también se incluye. Los tres tipos de biomoléculas se inmovilizan sobre sustratos nanoestructurados por diferentes métodos, y los resultados de formación de imágenes SERS se reportan. Las capacidades de SERS para detectar modos vibracionales de proteínas inmovilizadas en la superficie, así como para capturar el ligando de una proteína-La unión d aptámero-ligando se demuestran. Los resultados también ilustran la influencia de la geometría de la nanoestructura de la superficie, biomoléculas estrategia de inmovilización, la actividad Raman de las moléculas y la presencia o ausencia de la unión en los espectros SERS adquirido ligando.
Introduction
Capacidades para desarrollar y caracterizar los sistemas nano-biológica conjugadas interfaz nanoestructuras sólidos y polímeros biológicos se están convirtiendo cada vez más importante para nuevos avances en la próxima generación de bio-sensores y bio-tecnologías de accionamiento 1,2. Esto implica estudios multidisciplinarios a través de una serie de campos de investigación, tales como la fabricación de componentes pertinentes de estado sólido (electrodos micro-nano o, revestimientos nanoingeniería, nanocables, o nanopartículas) 2,3,4; la inmovilización de biomoléculas en las superficies para crear bioconjugados deseados 5,6,7; y el seguimiento de las interfaces de nano-biológica 1. En la mayoría de los casos, la selección de fabricación óptima, bio-funcionalización, y los métodos de caracterización es fuertemente relacionados entre sí. Claramente, la elección de las técnicas de nanofabricación sería impulsado por los requisitos de los componentes de estado sólido del sistema, siendo dependiente en gran medida del método de detección, que en tuRn es determinada por la naturaleza de los biopolímeros implicados y de la finalidad de la supervisión de la interfaz.
Fuera de una amplia variedad de técnicas aplicadas a caracterizar los sistemas Bioconjugate 1,3, una mayor superficie de espectroscopia Raman (SERS) ha surgido como un método muy prometedor para la detección de especies químicas y biológicas en superficies 8,9,10,11. SERS emplea la dispersión inelástica de luz monocromática por biomoléculas inmovilizada en la superficie (Figura 1) que permiten la captura de firmas únicas correspondientes a las vibraciones moleculares. Esta capacidad para distinguir entre las diferentes moléculas sin la participación de etiquetas, química compleja, o pasos que consumen tiempo, hace SERS un método potencialmente muy eficiente de bio-detección. Otra ventaja importante de SERS es su alta sensibilidad. La excitación de plasmones de superficie localizados por la luz que interactúa con nanoestructuras de metales nobles (sustratos SERS) aumenta dramáticamente el intensity de dispersión Raman por el analito, lo que permite la detección de cantidades muy pequeñas de moléculas, a partir de monocapas hasta el límite de una sola molécula 8,9,10,11. Finalmente, la mayoría de las biomoléculas requieren soluciones acuosas para ser estable. Porque el agua a menudo tiene actividad Raman limitado, señal de fondo a partir de muestras acuosas se minimiza 9. Aplicaciones de la SERS han mostrado un aumento exponencial en la última década 10. Sin embargo, un reto muy discutido de SERS es que la mejora electromagnética de dispersión Raman depende críticamente del tamaño, la forma y el espaciamiento de nanoestructuras metálicas donde las olas plasmónicas se inducen 11,12,13. A fin de lograr SERS eficientes y reproducibles mediciones, el control sobre la geometría se requiere sustrato a las dimensiones a nanoescala.
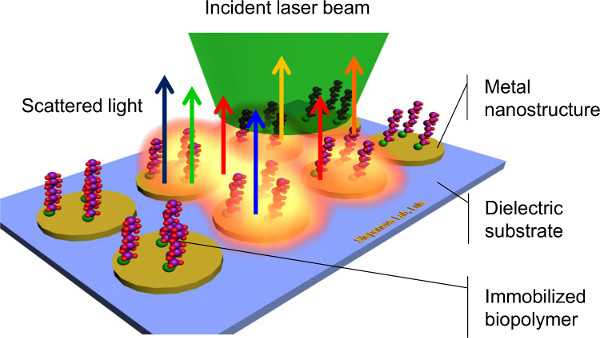
Figura 1. Scheme de la espectroscopia de Raman de superficie mayor.
Numerosos métodos empleados para fabricar sustratos SERS 11,12,13 se pueden dividir en métodos de abajo arriba y de arriba hacia abajo. Los métodos de la primera tipo emplean varios procesos de auto-montaje o la síntesis química dirigida para producir nanoestructuras. A menudo abordan ejemplos incluyen la inmovilización de nanopartículas monodispersas sobre soportes sólidos 11,12,13, térmica, pulverización catódica o deposición electroquímica de películas metálicas rugosas 11,12, y diversos métodos de síntesis química 13. Aunque tales técnicas tienden a ser relativamente sencilla y económica, la mayoría de ellos son desafiados por una falta de control sobre la ubicación de las estructuras, y limitada reproducibilidad de muestra a muestra.
En contraste, las técnicas de litografía de arriba hacia abajo emplean instrumentos manipulables, como haces de partículas para crear patrones deseados en las superficies. Uno de los más utilizadosmétodos de nanolitografía, litografía por haz de electrones (EBL), ofrece un control excelente sobre las características hasta por debajo de 10 nm y también una flexibilidad para permitir diferentes diseños de sustrato sobre soportes sólidos 11,12. En EBL, un haz de electrones enfocado a un lugar de unos pocos nanómetros de diámetro en las exploraciones a través de una superficie de un material sensible de electrones (resistir) causando un cambio químico en las regiones expuestas. Para tono positivo resiste como polimetilmetacrilato (PMMA), electrones resultados de exposición viga en la escisión de las cadenas poliméricas que componen la resisten, lo que lleva a un aumento de la solubilidad en un disolvente apropiado (desarrollador). El proceso de litografía por haz de electrones incluye spin-recubrimiento de una capa uniforme de resistir sobre un sustrato; la exposición del material de resistir objetivo en una cámara de vacío con un haz de electrones; y el desarrollo de la muestra para eliminar las regiones solubles.
Soportes dieléctricos debajo de nanoestructuras metálicas, tales como sílice fundida, tienen been demostrado que aumenta significativamente las intensidades en SERS debido a la localización de las ondas plasmónicas comparación con otros materiales tales como el silicio 14,15. Sin embargo patrón EBL en sustratos dieléctricos, especialmente en la nanoescala, implica retos importantes debido a la acumulación de carga durante la exposición. Anteriormente, hemos demostrado 16,17 que estas dificultades se pueden superar mediante la colocación de capas de polímero conductor por encima de la resistencia. La Figura 2 muestra un esquema del proceso global de fabricación usando la exposición y el desarrollo EBL seguido por la deposición de metal y despegue para producir nanoestructuras metálicas sobre fusionado soportes de sílice. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2. Esquema de correoLectron litografía por haz, la deposición de metal, y el proceso de despegue etapas empleadas para fabricar nanoestructuras metálicas sobre sustratos dieléctricos 16-19. En este trabajo, presentamos toda la secuencia de pasos del proceso que implican sustratos SERS fabricación por EBL, bio-funcionalización de los sustratos, y colección de los espectros de Raman. Tres diseños explorados en nuestros trabajos recientes 18,19 se abordan (ver figuras 3 y 4, y en la Tabla 1). En Diseño 1, proteína recombinante A se inmoviliza sobre bio-funcionalizado Au nanoestructuras sobre un soporte de sílice fundida (FS) 18, y la detección SERS de la proteína se demostró. En Diseño 2, la proteína 21,26,27 con y sin el ligando (D-glucosa) de unión a glucosa-recombinante se inmoviliza por medio de etiquetas de histidina en los espacios entre las nanoestructuras de Ag sobre FS recubiertos de Ni, y la unión de la glucosa a la proteína se detecta. En Diseño 3, tiolado DN unión dopamina-Un aptámero 19,23 se inmoviliza sobre Au nanoestructuras sobre FS, y la unión de la dopamina por aptámero inmovilizado se demostró. Inclusive de todos los pasos experimentales relevantes de la preparación del sustrato a la adquisición de espectros de Raman, y representativa de diferentes biomoléculas y estrategias de inmovilización, estos ejemplos son útiles para una amplia variedad de aplicaciones, desde la investigación de exploración interrogar interfaces de nano-biológica por SERS al desarrollo de SERS biosensores de moléculas pequeñas que emplean en proteínas o vinculante como método de reconocimiento de aptámero-ligando. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3. Esquemas de tres diseños representativas utilizando diferentes biomoléculas, los métodos de immobilización, y materiales de sustrato: (A) la proteína A inmovilizada sobre los metales nobles nano-puntos funcionalizados por una monocapa autoensamblada (SAM) de ácido 11-mercaptodecanoic (MUA) en agua DI; (B) la proteína de unión de glucosa marcado con histidina (GBP) complejado con D-glucosa inmovilizada sobre la superficie del sustrato entre el metal nano-puntos nobles; (C) terminada en tiol de unión a la dopamina aptámero completado con la dopamina (DBA) inmovilizada en metal noble nano-puntos. Véanse más detalles en la Tabla 1. En Diseño 2 se ilustra por el panel (B), una muestra sin el correspondiente ligando también se preparó para la comparación. Haga clic aquí para ver una versión más grande de esta figura.

La Figura 4.Biomoléculas empleados en tres diseños: (A) la proteína A; Proteína (B) de unión de glucosa y D-glucosa; (C) de unión a DNA aptámero y la dopamina dopamina. Las estructuras terciarias de proteínas en (a) y (b) se han tomado de Protein Data Bank, AP ID 1BDD 20 y 2HPH 21, respectivamente, y dibujado con VMD para LINUXAMD64, versión 1.9.1 22. La estructura secundaria del aptámero en (c) se predice a partir de la secuencia de 23 usando ValFold 24 software y dibujado con PseudoViewer 3,0 25. Las letras G, A, T y C corresponden a la guanina, adenina, timina, citosina y nucleótidos, respectivamente. Haga clic aquí para ver una versión más grande de esta figura.
| Diseño 1 </td> | Diseño 2 | Diseño 3 | |
| Biopolímeros | Proteína A | Proteína de unión a glucosa (GBP) | Aptámero de unión de la dopamina (DBA) |
| Aglutinante | Ácido 11-mercaptoundecanoico (MUA) monocapa autoensamblada (SAM) | Etiquetas de histidina | Enlazadores tiol |
| Ligando | Ninguno | D-glucosa | La dopamina |
| Solución | Desionizada (DI) | Tampón de fosfato potásico | Tris (hidroximetil) aminometano (TRIS) y ácido etilendiaminotetraacético tampón (EDTA); Salina tamponada con fosfato (PBS) |
| Sustrato | Au estructuras en FS | Estructuras Ag en FS recubierto con Ni | Au estructuras en FS |
| Ar con dibujosea | 4 micras x 10 micras | 4 m x 8 micras | 4 micras x 10 micras |
| Patrón | Au puntos, 50 nm de paso | Puntos de Ag, 40 nm de paso | Hexágonos Au, pitch 200 nm |
| Hexágonos Ag, pitch 200 nm | Almohadillas no estructurados Au | ||
| Almohadillas no estructurados Ag | |||
| Dosis de exposición EBL | Puntos: | Puntos: 105 mC / cm 2 | Hexágonos: 180 mC / cm 2 |
| Matriz I 120 mC / cm 2 | Hexágonos: 170 mC / cm 2 | ||
| Matriz II 96 mC / cm 2 | |||
| Matriz III 72mC / cm 2 | |||
| Excitación láser de longitud de onda | 532 nm | 532 nm | 780 nm |
Tabla 1. Tres proyectos de sistemas de nano-biológica.
Protocol
Representative Results
Discussion
SERS está ganando reconocimiento como una muy poderosa técnica de bio-detección que ofrece muchas ventajas únicas. La relación con las vibraciones moleculares permite identificar de manera selectiva "huellas digitales" de sustancias específicas de análisis de espectros SERS, mientras que la muy alta sensibilidad permite detectar cantidades muy pequeñas de analito 9,10,11,35. Además, SERS es una técnica no destructiva que también es relativamente insensible al agua, y por lo tanto se adapta muy bien para sondear materiales biológicos en su entorno natural, 9 acuosa. Los resultados presentados enfatizan estas ventajas también demuestran como más fuerte potencial de SERS como una técnica libre de etiquetas muy flexible de bio-detección. En tres diseños que emplean monocapas de diferentes biomoléculas sustrato inmovilizado, modos Raman se han detectado que podrían confianza atribuye a los analitos particulares. Que la detección de estas biomoléculas, or sus respectivos ligandos, se ha demostrado el empleo de superficies planas de sílice fundida como el soporte para sustratos SERS, hace que los diseños compatibles con la electrónica y ajustes actuales de microfluidos, prometiendo numerosas aplicaciones en relación con las arquitecturas de bio-electrónica de interfaz materiales biológicos con superficies de electrónica emergente y dispositivos electroquímicos 2,3. Es importante destacar que, en dos de tres diseños de detección SERS se ha demostrado para la unión específica de las pequeñas moléculas, tales como la glucosa y la dopamina, el empleo de monocapas de la proteína y aptámero inmovilizado en la superficie, respectivamente, como los elementos de reconocimiento.
Sin embargo, varios aspectos deben ser atendidos con el fin de lograr un SERS bio-detección eficaz en el ajuste "on-chip". En primer lugar, un desafío bien conocido que es común para la mayoría de biomoléculas es su propensión a degradarse, en particular cuando se expone a condiciones no naturales tales como ENVI secamedio o intenso de luz láser. A lo largo del protocolo, hemos hecho hincapié en la importancia de mantener siempre las muestras bio-funcionalizado sumergidos en soluciones adecuadas durante todo el experimento, desde la preparación de las muestras para la adquisición de espectros Raman. Para este último, una cámara a prueba de agua de encargo ha sido diseñado (Figura 7) para evitar la evaporación del líquido durante las exposiciones de láser. La duración de la exposición y de la intensidad del láser también se debe limitar, como se describe en el paso 5.3 del protocolo para evitar el daño de las muestras.
Los resultados de la detección SERS se encuentran sensible a la geometría del sustrato empleado, y en particular la separación inter-característica de las nanoestructuras metálicas. Como se desprende de las figuras 8 y 9, la intensidad SERS de Diseño 1 muestras depende en gran medida de la anchura de los huecos entre Au nano-puntos en sílice fundida. Fuera de tres matrices de Au nanopuntos probados in este diseño (Figura 8), la mayor intensidad de Raman se logra con la matriz I, que tiene los huecos estrechos entre las características de Au y por lo tanto proporciona una mejora más eficiente campo electromagnético. Como muestra la Figura 9 ilustra, se requiere el control de las separaciones entre las características a nivel de 10 a 20 nm o menos. Empleando EBL para la fabricación de sustratos SERS, como se ha demostrado aquí, proporciona una resolución eficiente específicamente para el control de las anchuras de las brechas entre las funciones. Con una EBL-tono positivo resistir tales como PMMA, el tamaño de los agujeros en las máscaras de PMMA se puede variar simplemente cambiando las dosis de exposición. Después de lift-off esto resulta en diferentes tamaños de puntos de metal, y la anchura de los vacíos entre los puntos puede ser sintonizado como se desee mediante la selección de la exposición EBL adecuada dosis de 18.
El otro reto es la optimización de la geometría sustrato SERS para la aplicación específica de detección de bio. Aunque el efecto de mejora increases con una disminución de las brechas entre las características, el tamaño relativamente grande de las moléculas biológicas impone limitaciones sobre la estrecha pueden ser los vacíos. Esto es evidente a partir de los resultados para el Diseño 2, donde el método de inmovilización es tal que la proteína se une de manera eficiente sólo a la superficie entre los puntos de metales nobles, pero no a los mismos puntos (véase la Figura 3B). Como se desprende de la Figura 10, los espectros SERS para los cojines de Ag no estructurados no muestran ninguna banda a partir del analito. Aunque las pastillas presentan una estructura nano-cristalino con brechas entre las islas muy delgadas (ver Figura 6F) estas brechas son demasiado estrechas para dar cabida a una molécula de proteína. Sin embargo, se añade otra dimensión de complejidad cuando tiene proteína ligando de unión a detectar. En la Figura 10, las bandas SERS CH son más pronunciadas en los espectros de ligando unido GBP que en el de un ligando libre, lo que puede explicarse hipotéticamente por un cambio en la conformación GBPtras la unión de D-glucosa 2 7,27, resultando en una estructura más rígida con una mayor actividad Raman. Si se comparan los dos sustratos nanoestructurados, la banda CH de la proteína-ligando libre es más fuerte en los espectros SERS obtenido con el sustrato nano-puntos, mientras que tanto las proteínas y glucosa CH bandas de proteína ligando determinada son más pronunciados con los nano-hexágonos sustrato. Se espera que dos factores para dar lugar a estas diferencias, la disponibilidad de espacio entre Ag cuenta que el GBP podría obligar a Ni, y la susceptibilidad de la proteína ligando determinada y ligando libre a la mejora electromagnético de la dispersión Raman en "puntos calientes" entre estas características. Por un lado, el patrón-nano puntos ofrece una zona inter-estelar relativamente grande donde recubrimiento de Ni está disponible para que la proteína se une, lo que puede explicar una banda CH más pronunciada observado para GBP libre de glucosa en Ag sustrato nano-puntos. Por otro lado, debido a su estruc no uniformetura (ver Figura 6D), Ag nano-hexágonos podría ser propensos a mostrar una mejora electromagnética fuerte en huecos estrechos entre las islas de Ag en nano-hexágonos resultando en fuertes bandas CH vibración desde GBP glucosa unida en el sustrato nano-hexágonos. Algunos detalles de esta interacción requieren mayor verificación y optimización de sustratos SERS para analitos complejos que implican proteínas de gran tamaño como el GBP se encuentra todavía en la tubería.
Claramente, la detección SERS de la unión empleando biomoléculas inmovilizadas como un elemento de reconocimiento ligando se facilita cuando sólo el ligando es Raman activo en una región seleccionada, mientras que los otros componentes no son. Este es el caso de Diseño 3, donde se obtienen SERS pronunciadas bandas de dopamina aptámero unido (Figura 11). La pareja aptámero-dopamina exhibe excelente especificidad y el espectro SERS comprende bandas pronunciadas sin señal de fondo significativo.
<p class="jove_content"> Futuro avance de la tecnología SERS etiqueta-fee implicaría extensas pruebas de mejora de la señal SERS biomoléculas "con una amplia gama de diferentes diseños nanoestructura superficie. El uso de escritura directa litografía por haz de electrones para fabricar varios nanoestructuras con un excelente nivel de control sobre el tamaño, forma, y la separación inter-característica, combinada con los protocolos de preparación de muestras presentadas aquí, sería facilitar la comparación y validación cruzada de los resultados obtenidos por diferentes grupos de investigación. Esto sería abordar el importante reto de reproducibilidad cuando sustratos SERS se fabrican empleando alternativa "abajo hacia arriba" métodos 11,12,13, lo que permite un mejor control del tamaño de nanoestructura metálica y la posición hacia una identificación fiable de diseño óptimo sustrato para una amplia variedad de aplicaciones. Escalabilidad de estas técnicas puede posteriormente ser mejorada mediante la combinación de EBL con los métodos de nanolitografía complementarios, tales como nanoimpresión litográfica 19 hacia el futuro la producción en serie de diseños a escala nanométrica optimizado empleando las técnicas EBL sintonizables.Disclosures
The authors have nothing to disclose.
Acknowledgements
The authors would like to thank: David Wishart, Valentyna Semenchenko, Mark McDermott, Michael Woodside, and Albert Cao for their help in developing and preparing the protein conjugates as well as the DNA aptamer; T. M. Fahim Amin, Mosa Sharmin Aktar, and Trevor Olsen for their assistance in the sample preparation, Jonathan Mane for his assistance in generating images of the molecular structures; and the funding sources including the National Research Council of Canada – National Institute for Nanotechnology (NRC-NINT), Natural Sciences and Engineering Research Council of Canada (NSERC), and the University of Alberta for supporting the work.
Materials
| 11-Mercaptoundecanoic acid (MUA) | Sigma Aldrich (www.sigmaalrich.com) |
450561 ALDRICH | Used for surface functionalization in Design 1 |
| Conductive polymer | Mitsubishi Rayon (www.mrc.co.jp) |
aquaSAVE-57xs | A 70 nm thick layer is used as anti-charging coating for EBL exposures |
| D-glucose | Collaborator Lab. | Ligand in Design 2 | |
| Dopamine | Collaborator Lab. | Ligand in Design 3 | |
| Dopamine binding aptamer (DBA) | Integrated DNA Technologies Inc. (www.idtdna.com) |
5'- /Thiol Modifier C6 S-S/ AAAAAAAAAA GTCTCTGTGT GCGCCAGAGA ACACTGGGGC AGATATGGGC CAGCACAGAA TGAGGCCC-3' | Biopolymer in Design 3 |
| Fused silica wafers | Mark Optics www.markoptics.com |
||
| Glucose binding protein (GBP) | Collaborator Lab. (www.ncbi.nlm.nih.gov/protein/gi|145579532) |
PDB ID 2HPH | Biopolymer in Design 2 |
| High vacuum grease | Dow Corning (www.dowcorning.com) |
Used to seal water-proof chamber, step 5.1 | |
| Hydrogen Peroxide 30%, H2O2 | J.T. Baker | Used for pirahna solution, step 1.2 | |
| N-ethyl-N'-(3-(dimethylamino) propyl) carbodiimide (EDC) | Sigma Aldrich www.sigmaaldrich.com |
03450 FLUKA | Used for immobilization of biopolymer in Design 1 |
| N-Hydroxysuccinimide (NHS) | Sigma Aldrich (www.sigmaaldrich.com) |
130672 ALDRICH | Used for immobilization of biopolymer in Design 1 |
| Potassium phosphate buffer | Collaborator Lab. | Buffer used in Raman sampling | |
| Phosphate buffered | Collaborator Lab. | Solvent in Design 3 | |
| saline (PBS) | |||
| Polymethylmethacrylate (PMMA) 950 A2 | MicroChem (www.microchem.com) |
A 90 nm thick layer is used as EBL positive tone resist | |
| Recombinant protein A | Protein Mods Inc (www.proteinmods.com) |
PDB ID 1BDD (www.rcsb.org/pdb/explore/explore.do?structureId=1bdd) |
Biopolymer in Design 1 |
| Sulfuric acid 96%, H2SO4 | J.T. Baker | Used for pirahna solution, step 1.2 | |
| Tris(hydroxymethyl)aminomethane (TRIS) and ethylenediaminetetraacetic acid (EDTA) buffer | Sigma Aldrich (www.sigmaaldrich.com) |
T9285 SIGMA | Buffer in Design 3 |
| Dicing saw | Diamond Touch Technology Inc. | Used to cut FS wafer, step 1.1 | |
| (17301 W Colfax Ave # 152, Golden, CO) | |||
| Electron beam evaporator | Kurt J. Lesker (www.lesker.com) |
Used for Au and Ag evaporation | |
| Electron beam evaporator | Johnsen Ultravac (JUV) (www.ultrahivac.com) |
JuV E-gun | Used for Ni evaporation |
| Microscope cover slips (25 mm) | Fisher Scientific (www.fishersci.ca) |
12-545-102 | Used in water-proof chamber, step 5.1 |
| Microscope slides (3×1 in.) | Fisher Scientific (www.fishersci.ca) |
Used in water-proof chamber, step 5.1 | |
| Raith 150TWO EBL exposure system | Raith Inc. (www.raith.com) |
Raith 150TWO system | Used for EBL exposures, step 2.2 |
| Raman microscope | Thermo Scientific (www.thermoscientific.com) |
Nicolet Almega XR | Used for Raman spectroscopy, step 5.3 |
| Sonicator system | Branson (www.bransonic.com) |
Used for liftoff and solutions mixing | |
| Spinner | Brewer Spinner and Hotplate (www.brewerscience.com) |
Cee 200X and Cee 1300X | Used to spin-coat PMMA and conductive polymer, step 2.1 |
References
- Sapsford, K. E., Tyner, K. M., Dair, B. J., Deschamps, J. R., Medlintz, I. L. Analyzing Nanomaterial Bioconjugates: A Review of Current and Emerging Purification and Characterization Techniques. Anal. Chem. 83, 4453-4488 (2011).
- Walcarius, A., Minteer, S. h. D., Wang, J., Yu, L., Merkoçi, A. Nanomaterials for Bio-Functionalized Electrodes: Recent Trends. J. Mater. Chem. B. 1, 4878-4908 (2013).
- Kim, J., et al. Applications, Techniques, and Microfluidic Interfacing for Nanoscale Biosensing. Microfluid. Nanofluid. 7, 149-167 (2009).
- Rassaei, L., Singh, P. S., Lemay, S. G. Lithography-Based Nanoelectrochemistry. Anal. Chem. 83, 3974-3980 (2011).
- Wong, L. S., Khan, F., Micklefield, J. Selective Covalent Protein Immobilization: Strategies and Applications. Chem. Rev. 109, 4025-4053 (2009).
- Ley, C., Holtmann, D., Mangold, K. -. M., Schrader, J. Immobilization of Histidine-Tagged Proteins on Electrodes. Colloids and Surfaces B: Biointerfaces. 88, 539-551 (2011).
- Kim, D., Herr, A. E. Protein Immobilization Techniques for Microfluidic Assays. Biomicrofluidics. 7, 041501 (2013).
- Anker, J. N., Hall, W. P., Lyandres, O., Shah, N. C., Xhao, J., Van Duyne, R. P. Biosensing with Plasmonic Nanosensors. Nature Materials. 7, 442-453 (2008).
- Bantz, K. C., et al. Recent Progress in SERS Biosensing. Phys.Chem. 13, 11551-11567 (2011).
- Sharma, B., Frontiera, R. R., Henry, A. -. I., Ringe, E., Van Duyne, R. P. SERS: Materials, Applications, and the Future. Mater. Today. 15, 16-25 (2012).
- Kleinman, S. L., Frontiera, R. R., Henry, A. -. I., Dieringer, J. A., Van Duyne, R. P. Creating, Characterizing, and Controlling Chemistry with SERS Hot Spots. Phys.Chem.Chem.Phys. 15, 21-36 (2013).
- Fan, M., Andrade, F. S., Brolo, A. G. A Review on the Fabrication of Substrates for Surface Enhanced Raman Spectroscopy and their Applications in Analytical Chemistry. Anal. Chim. Acta. 693, 7-25 (2011).
- Cao, Y., Li, D., Jiang, F., Yang, Y., Zh, H. Engineering Metal Nanostructure for SERS Application. J. Nanomater. 123812, 1-12 (2013).
- Glembocki, O., Rendell, R., Alexson, D., Prokes, S., Fu, A., Mastro, M. Dielectric-Substrate-Induced Surface-Enhanced Raman Scattering. Phys. Rev. B. 80, 085416 (2009).
- Merlen, A., et al. Surface Enhanced Spectroscopy with Gold Nanostructures on Silicon and Glass Substrates. Surf. Sci. 605, 1214-1218 (2011).
- Muhammad, M., Buswell, S. C., Dew, S. K., Stepanova, M. Nanopatterning of PMMA on Insulating Surfaces with Various Anticharging Schemes Using 30 keV Electron Beam Lithography. J. Vac. Sci. Technol. B. 29, 06F304 (2011).
- Peters, R., Fito, T., Gutierrez-Rivera, L., Dew, S. K., Stepanova, M. Study of Multilayer Systems in Electron Beam Lithography. J. Vac. Sci. Technol. B. 31, 06F407 (2013).
- Gutierrez-Rivera, L., Peters, R., Dew, S., Stepanova, M. Application of EBL Fabricated Nanostrucutred Substrates for SERS Detection of Protein A in Aqueous Solution. J. Vac. Sci. Technol.B. 31, (2013).
- Gutierrez-Rivera, L., Peters, R., Dew, S., Stepanova, M. Surface-Enhanced Raman Spectroscopy Detection of Protein-Ligand Binding Using D-glucose and Glucose Binding Protein on Nanostructured Plasmonic Substrates. , (2014).
- Peters, R. . Fabrication and Testing of Surface-Enhanced Raman Spectroscopy Substrates for the Detection of Biomolecules [MSc Thesis]. , (2014).
- Gouda, H., Torigoe, H., Saito, A., Sato, M., Arata, Y., Shimada, I. Three-Dmensional Solution Structure of the B Domain of Staphylococcal Protein A: Comparisons of the Solution and Crystal Structures. Biochemistry. 31, 9665-9672 (1992).
- Cuneo, M. J., Johnson, S. J., Beese, L. S., Hellinga, H. W. High Resolution Structure of E. Coli Glucose/Galactose Binding Protein Bound with Glucose. Research Collaboratory for Structural Bioinformatics (RCSB) Protein Data Bank. , (2009).
- Humphrey, W., Dalke, A., Schulten, K. VMD – Visual Molecular Dynamics. J. Molec. Graphics. 14, 33-38 (1996).
- Walsh, R., DeRosa, M. C. Retention of Function in the DNA Homolog of the RNA Dopamine Aptamer. Biochem. Biophys. Res. Comm. 388, 732-735 (2009).
- Akitomi, J., Kato, S., Yoshida, Y., Horii, K., Furuich, M., Waga, I. ValFold: Program for the Aptamer Truncation Process. Biomed. Inf. 7, 38-40 (2011).
- Han, K., Lee, Y., Kim, W. PseudoViewer: Automatic Visualization of RNA Pseudoknots. Bioinformatics. 18, S321-S327 (2002).
- Dwyer, M. A., Hellinga, H. W. Periplasmic Binding Proteins: a Versatile Superfamily for Protein Engineering. Curr. Opin. Struct. Biol. 14, 495-504 (2004).
- Benson, D. E., Conrad, D. W. Design of Bioelectronic Interfaces by Exploiting Hinge-Bending Motions in Proteins. Science. 293, 1641-1644 (2001).
- Bozic, S., Chorzempa, J. . Pirahna Cleaning. , (2011).
- Mohammad, M. A. Raith 150TWO SOP. , (2011).
- Schmidt, M. W., et al. General Atomic and Molecular Electronic Structure System. J. Comput. Chem. 14, 1347-1363 (1993).
- Bode, B. M., Gordon, M. S. MacMolPlt: a Graphical User Interface for GAMESS. J. of Mol. Graph. Mod. 16, 133-138 (1998).
- Bright, A., Devi, T. S. R., Gunasekaran, S. Spectroscopical Vibrational Band Assignment and Qualitative Analysis of Biomedical Compounds with Cardiovascular Activity. Int. J. Chem. Tech. Res. 2, 379-388 (2010).
- Bandekar, J. Amide Modes and Protein Conformation. Biochim. Biophys. Acta. 1120, 123-243 (1992).
- Barth, A., Zscherp, C. What Vibrations Tell About Proteins. Quarterly Reviews of Biophysics. 35, 369-340 (2002).
- Chrimes, A. F., Khoshmanesh, K. h., Stoddart, P. R. M. i. t. c. h. e. l. l. A., Kalantar-Zadeh, K. Microfluidics and Raman Microscopy: Current Applications and Future Challenges. Chem. Soc. Rev. 42, 5880-5906 (2013).
- Park, S. -. K., Lee, N. -. S., Lee, S. -. H. Vibrational Analysis of Dopamine Neutral Base based on Density Functional Force Field. Bull.-Korean Chem. Soc. 21, 959-968 (2000).
- Briand, E., Salmain, M., Compère, C., Pradier, C. M. Immobilization of Protein A on SAMs for the elaboration of immunosensors. Coll. Surf. B: Biointerfaces. 53, 215-224 (2006).
- Lakowicz, L. R., et al. Radiative decay engineering: 2. Effects of Silver Island Films on Fluorescence Intensity, Lifetimes, and Resonance Energy Transfer. Analytical biochemistry. 301, 261-277 (2002).

