Détermination du potentiel Zeta via nanoparticulaires translocation Velocities à travers un accordable Nanopore: Utilisation de particules d'ADN modifié comme un exemple
Summary
Ici, nous utilisons un nanopore accordable en polyuréthane intégré dans une technique de détection d'impulsions résistif pour caractériser les nanoparticules de la chimie de surface par la mesure des vitesses de translocation de particules, qui peut être utilisé pour déterminer le potentiel zéta des nanoparticules individuelles.
Abstract
technologies de Nanopore, connus collectivement comme capteurs de pulsations résistifs (RPS), sont utilisées pour détecter, quantifier et caractériser les protéines, les molécules et les nanoparticules. détection d'impulsion résistive accordable (de TRPS) est une adaptation relativement récente au RPS qui incorpore un pore accordable peut être modifiée en temps réel. Ici, nous utilisons TRPS pour surveiller les temps de translocation des nanoparticules d'ADN modifiées comme ils traversent la membrane poreuse réglable en fonction de la concentration d'ADN et de la structure ( par exemple, simple brin en ADN double brin).
TRPS repose sur deux électrodes Ag / AgCl, séparés par une membrane poreuse en élastomère qui établit un courant ionique stable lors d'un champ électrique appliqué. Contrairement à diverses technologies de caractérisation des particules optiques à base, TRPS peuvent caractériser des particules individuelles dans une population de l'échantillon, ce qui permet pour les échantillons multimodales à analyser avec facilité. Ici, nous démontrons mesures du potentiel zêtavia des vitesses de translocation de particules de normes connues et les appliquer à l'échantillon des temps de translocation analyte, conduisant ainsi à la mesure du potentiel zeta de ces analytes.
En plus d'acquérir des valeurs de potentiel zêta moyennes, les échantillons sont tous mesurés en utilisant une perspective particule par particule présentant plus d'informations sur un échantillon donné par l'échantillon répartition de la population, par exemple. De ce fait, cette méthode démontre un potentiel dans les applications de détection pour les domaines médicaux et environnementaux.
Introduction
nanoparticules fonctionnalisés sont de plus en plus populaire en tant que biocapteurs dans les deux domaines de la médecine et de l'environnement. La possibilité de modifier la chimie de surface d'une nanoparticule, avec de l' ADN, par exemple, se révèle utile pour les systèmes de délivrance de médicaments ciblés 1 et de suivi des interactions ADN-protéines 2-4. Une propriété de nanoparticules de plus en plus commun étant utilisé dans des essais biologiques et dans la prestation de produits thérapeutiques est superparamagnétisme 5. particules superparamagnétiques (SPPS) sont extrêmement utiles pour identifier et éliminer les analytes spécifiques à partir de mélanges complexes et peuvent le faire avec la simple utilisation d'un seul aimant. Une fois enlevés, les particules d'analyte lié peuvent être caractérisés et analysés adaptés à l'usage.
Les procédés antérieurs utilisés pour la détection et la caractérisation des nanoparticules comprennent des techniques optiques telles que la diffusion dynamique de lumière (DLS), autrement connu comme la spectroscopie de corrélation de photons. Bien qu'un saluttechnique de débit gh, DLS est limitée à être une technique basée sur la moyenne et lors de l' analyse des échantillons multimodaux sans ajout de logiciels spécialisés, les plus grosses particules produisent un signal beaucoup plus dominante, laissant certaines des plus petites particules complètement inaperçues 6,7. Particule par particule techniques de caractérisation sont donc beaucoup plus favorable pour analyser nanoparticule et les systèmes de nanoparticules fonctionnalisés.
technologies basées RPS sont basés sur l'application d'un champ électrique à un échantillon et la surveillance du mécanisme de transport des particules à travers un nanopore synthétique ou biologique. Une technique relativement récente de détection de nanoparticules et la caractérisation basée sur RPS est résistive accordable impulsion de détection (TRPS) 8-16. TRPS est un système à deux électrodes séparées par une membrane poreuse en élastomère, accordable. Une méthode accordable de pores permet analytes d'une gamme de forme 17 et la taille à mesurer via leur transdes mécanismes de port à travers les pores. Les pores accordables ont déjà été utilisés pour la détection de petites particules (diamètre 70-95 nm) produisant des résultats comparables à d' autres techniques telles que la spectroscopie électronique à transmission (MET) 10. Lorsqu'un champ électrique est appliqué, un courant ionique est observé et que des particules / molécules passent à travers les pores, ils bloquent temporairement les pores, ce qui provoque une réduction du courant qui peut être défini comme un «événement de blocage. Chaque événement de blocus est représentatif d'une seule particule de sorte que chaque particule dans un échantillon peut être caractérisé individuellement en fonction de l'ampleur du blocus, Δ 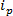 Et toute la largeur à mi-hauteur, LMH, ainsi que d' autres propriétés de blocus. L'analyse des particules individuelles lors de leur passage à travers un nanopore est avantageux pour les échantillons multimodales comme TRPS peuvent réussir et efficacement distinguer une gamme de tailles de particules amongst un seul échantillon. Accordable détection d'impulsion résistive complète taille 10, zeta potentiels 12,18 et la concentration de 15 mesures simultanément en une seule opération et peut donc différencier encore des échantillons de semblable, sinon la même taille par leur charge de surface 19; un avantage par rapport aux techniques de dimensionnement alternatives.
Et toute la largeur à mi-hauteur, LMH, ainsi que d' autres propriétés de blocus. L'analyse des particules individuelles lors de leur passage à travers un nanopore est avantageux pour les échantillons multimodales comme TRPS peuvent réussir et efficacement distinguer une gamme de tailles de particules amongst un seul échantillon. Accordable détection d'impulsion résistive complète taille 10, zeta potentiels 12,18 et la concentration de 15 mesures simultanément en une seule opération et peut donc différencier encore des échantillons de semblable, sinon la même taille par leur charge de surface 19; un avantage par rapport aux techniques de dimensionnement alternatives.
Le potentiel zêta est défini comme étant le potentiel électrostatique au niveau du plan de cisaillement 20, et elle est calculée à partir des vitesses de particules comme ils traversent un pore 19. mesures de potentiel Zeta de particules individuelles donne ainsi un aperçu des mécanismes et le comportement des systèmes de nanoparticules en solution, de précieuses informations pour l'avenir des modèles de test de nanoparticules pour une gamme d'applications translocation. analyse de particules par particule de cette nature permet également la diffusion et la distribution des valeurs de potentiel zêta parmi un échantillon de population à explorer, permettant plus d'informations ocinétique n de réaction (simple brin à l'ADN double brin, par exemple) et des stabilités de particules en solution à atteindre.
Ici, nous décrivons une technique qui détecte et caractérise les deux surfaces SPP non modifiées et modifiées par l'ADN. Le protocole décrit ici est applicable à toute une gamme de nanoparticules inorganiques et biologiques, mais nous démontrons la procédure en utilisant des surfaces d'ADN modifiées en raison de leur large éventail d'applications. Cette technique permet à l'utilisateur de distinguer entre les cibles d'ADN simple brin et double brin sur une surface nanoparticulaire, sur la base des vitesses de translocation de particules à travers un système de pores et donc leurs potentiels zêta.
Protocol
Representative Results
Discussion
Le calcul du potentiel zêta utilisé une méthode basée sur l' étalonnage lié au travail par Arjmandi et al. 21. La durée de la translocation des particules lorsqu'elles traversent un nanopore est mesurée en fonction de la tension appliquée, en utilisant une vitesse moyenne de champ électrique et de particules sur la totalité d'un pore conique régulière. La mobilité électrophorétique est la dérivée de 1 / T (où T est la durée du blocage) par rapport à la tension, …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Les auteurs remercient Izon Science Ltd pour leur soutien. Le travail a été soutenu par la Commission européenne pour la recherche (PCIG11-GA-2012-321836 Nano4Bio).
Materials
| Phosphate buffered Saline (PBS) | Sigma Aldrich, UK | P4417 | 1 tablet dissolved in 200 mL deionised water to make buffer solution. |
| Tween-20 | Sigma Aldrich, UK | P1379 | 0.05% (v/v) in PBS buffer as a surfactant |
| Carboxyl polystyrene nanoparticles | Bangs Laboratories, US | CPC200 | Nominal diamter of 220 nm, raw concentration of 1E12 particles/mL, specific surface charge of 86 µeq/g (equivalent to a surface charge density of 3.2E19 C/nm^2. |
| Streptavidin coated nanoparticles | Ademtech, France | 3121 | Batch had binding capacity of 4352 pmol/mg (188 nM theoretical DNA binding capacity) at a raw concentration of 1.1E11 particles/mL. |
| Biotinylated oligonucleotides | Sigma Aldrich, UK | VC00001 | Supplier spec: Reverse Phase 1 purification (0.05 Scale); Biotin modification at 3' end; Lyophilised powders reconstituted to 100 µM using deionised water, and diluted as required. Sequences: CP 5'ATGGTTAAACCTCAC TACGCGTGGC[Btn]3' |
| Standard olignonucleotides | Sigma Aldrich, UK | VC00001 | Supplier spec: Reverse Phase 1 purification (0.05 Scale); Lyophilised powders reconstituted to 100 µM using deionised water, and diluted as required. Sequences of DNA targets: Fully complementary – 5'GCCACGCGTAGTGAGGTTTAACCAT3', Middle binding – 5'GTAGTGAGGT3', End binding – 5'GTTTAACCAT3', Partially complementary overhanging – 5'GTGAGGTTTAACCAT TTTTTTTTTTTTTTT3'. |
| Izon qNano | Izon Science, NZ | Inherent pressure on system of 47 Pa, | |
| Izon Variable Pressure Module (VPM) | Izon Science, NZ | Each 'cm' of pressure is equivalent to approximately 1000 Pa. | |
| Polyurethane nanopore membranes | Izon Science, NZ | NP150 | Analyte size range 60-480 nm, pore diameter of calculated to be 799 nm at a 45 mm stretch. |
| Magrack 6 | GE Healthcare, UK | 28-9489-64 | |
| Sonic Bath | Fisher Scientific, UK | 10692353 | 80 Watts |
| Vortexer | IKA, Germany | 0003365000 | |
| Rotary Wheel | Labnet International, US | H5500-230 V |
References
- Alexander, C. M., Maye, M. M., Dabrowiak, J. C. DNA-capped nanoparticles designed for doxorubicin drug delivery. Chem Commun. 47 (12), 3418-3420 (2011).
- Billinge, E. R., Platt, M. Aptamer based dispersion assay using tunable resistive pulse sensing (TRPS). Anal Methods. 7 (20), 8534-8538 (2015).
- Bulyk, M. L. Protein Binding Microarrays for the Characterization of Protein-DNA Interactions. Adv Biochem Eng Biotechnol. 104, 65-85 (2007).
- Platt, M., Rowe, W., Knowles, J., Day, P. J., Kell, D. B. Analysis of aptamer sequence activity relationships. Integr Biol. 1 (1), 116-122 (2009).
- Ruiz-Hernández, E., Baeza, A., Vallet-Regí, M. Smart Drug Delivery through DNA/Magnetic Nanoparticle Gates. ACS Nano. 5 (2), 1259-1266 (2011).
- Murdock, R. C., Braydich-stolle, L., Schrand, A. M., Schlager, J. J., Hussain, S. M. Characterization of Nanomaterial Dispersion in Solution Prior to In Vitro Exposure Using Dynamic Light Scattering Technique. Toxicol Sci. 101 (2), 239-253 (2008).
- Hupfield, S., Holsaeter, A. M., Skar, M., Frantzen, C. B., Brandl, M. Liposome size analysis by dynamic/static light scattering upon size exclusion-/field flow fractionation. J Nanosci Nanotechnol. 6 (7), 3025-3031 (2006).
- Roberts, G. S., et al. Tunable pores for measuring concentrations of synthetic and biological nanoparticle dispersions. Biosens Bioelectron. 31 (1), 17-25 (2012).
- Roberts, G. S., Kozak, D., Anderson, W., Broom, M. F., Vogel, R., Trau, M. Tunable nano/micropores for particle detection and discrimination: scanning ion occlusion spectroscopy. Small. 6 (23), 2653-2658 (2010).
- Vogel, R., et al. Quantitative sizing of nano/microparticles with a tunable elastomeric pore sensor. Anal Chem. 83 (9), 3499-3506 (2011).
- Booth, M. A., Vogel, R., Curran, J. M., Harbison, S., Travas-Sejdic, J. Detection of target-probe oligonucleotide hybridization using synthetic nanopore resistive pulse sensing. Biosens Bioelectron. 45, 136-140 (2013).
- Kozak, D., Anderson, W., Vogel, R., Chen, S. Simultaneous size and ζ-potential measurements of individual nanoparticles in dispersion using size-tunable pore sensors. ACS Nano. 6 (8), 6990-6997 (2012).
- Kozak, D., Anderson, W., Vogel, R., Trau, M. Advances in Resistive Pulse Sensors: Devices bridging the void between molecular and microscopic detection. Nano Today. 6 (5), 531-545 (2011).
- Weatherall, E., Willmott, G. R. Applications of tunable resistive pulse sensing. Analyst. 140, 3318-3334 (2015).
- Willmott, G. R., et al. Use of tunable nanopore blockade rates to investigate colloidal dispersions. J Phys Condens Matter. 22 (45), 454116 (2010).
- Blundell, E. L. C. J., Mayne, L. J., Billinge, E. R., Platt, M. Emergence of tunable resistive pulse sensing as a biosensor. Anal Methods. 7, 7055-7066 (2015).
- Platt, M., Willmott, G. R., Lee, G. U. Resistive Pulse Sensing of Analyte-Induced Multicomponent Rod Aggregation Using Tunable Pores. Small. 8 (15), 2436-2444 (2012).
- Vogel, R., Anderson, W., Eldridge, J., Glossop, B., Willmott, G. A variable pressure method for characterizing nanoparticle surface charge using pore sensors. Anal Chem. 84 (7), 3125-3131 (2012).
- Blundell, E. L. C. J., Vogel, R., Platt, M. Particle-by-Particle Charge Analysis of DNA-Modified Nanoparticles Using Tunable Resistive Pulse Sensing. Langmuir. 32 (4), (2016).
- Hunter, R. J. . Zeta Potential in Colloid Science: Principles and Applications. , (1981).
- Arjmandi, N., Van Roy, W., Lagae, L., Borghs, G. Measuring the electric charge and zeta potential of nanometer-sized objects using pyramidal-shaped nanopores. Anal Chem. 84 (20), 8490-8496 (2012).
- Bacri, L., et al. Dynamics of colloids in single solid-state nanopores. J Phys Chem B. 115 (12), 2890-2898 (2011).
- Cabello-Aguilar, S., et al. Dynamics of polymer nanoparticles through a single artificial nanopore with a high-aspect-ratio. Soft Matter. 10 (42), 8413-8419 (2014).
- Billinge, E. R., Muzard, J., Platt, M. Tunable resistive pulse sensing as a tool to monitor analyte induced particle aggregation. Nanomater Nanosci. 1 (1), 11 (2013).
- Li, J., Fan, C., Pei, H., Shi, J., Huang, Q. Smart Drug Delivery Nanocarriers with Self-Assembled DNA Nanostructures. Adv Mater. 25 (32), 4386-4396 (2013).
- Billinge, E. R., Broom, M., Platt, M. Monitoring aptamer-protein interactions using tunable resistive pulse sensing. Anal Chem. 86 (2), 1030-1037 (2014).
- Gold, L., et al. Aptamer-Based Multiplexed Proteomic Technology for Biomarker Discovery. PLoS One. 5 (12), e15004 (2010).
- Park, S. -. J., Taton, T. A., Mirkin, C. A. Array-Based Electrical Detection of DNA with Nanoparticle Probes. Science. 295 (5559), 1503-1506 (2002).

