High-Throughput Live Imaging af mikrokolonier til måling af heterogenitet i vækst og genekspression
Summary
Gær vækst fænotyper er præcist målt gennem meget parallelle time-lapse billeddannelse af immobiliserede celler vokser til mikrokolonier. Samtidig kan stresstolerance, proteinudtryk og proteinlokalisering overvåges og generere integrerede datasæt for at undersøge, hvordan miljømæssige og genetiske forskelle samt genekspressions heterogenitet blandt isogene celler modulerer vækst.
Abstract
Præcise målinger af heterogenitet mellem og inden for belastningen i mikrobielle vækstrater er afgørende for at forstå genetiske og miljømæssige input i stresstolerance, sygdomsfremkaldende evne og andre nøglekomponenter i fitness. Dette manuskript beskriver en mikroskopbaseret analyse, der sporer ca. 105 Saccharomyces cerevisiae mikrokolonier pr. eksperiment. Efter automatiseret time-lapse billeddannelse af gær immobiliseret i en multiwell plade, mikrokoloni vækstrater er let analyseres med brugerdefinerede billedanalyse software. For hver mikrokoloni kan ekspression og lokalisering af fluorescerende proteiner og overlevelse af akut stress også overvåges. Denne analyse giver mulighed for præcis vurdering af stammernes gennemsnitlige vækstrater samt omfattende måling af heterogenitet i vækst, genekspression og stresstolerance i klonpopulationer.
Introduction
Vækst fænotyper bidrager kritisk til gær fitness. Naturlig udvælgelse kan effektivt skelne mellem afstamninger med vækstrater, der varierer i det modsatte af den effektive befolkningsstørrelse, som kan overstige 108 personer1. Desuden er variation i vækstrater blandt individer i en population en evolutionært relevant parameter, da den kan tjene som grundlag for overlevelsesstrategier som bet hedging2,3,4,5,6. Derfor er assays, der giver mulighed for meget nøjagtige målinger af vækstphoenotyper og deres fordelinger, afgørende for undersøgelsen af mikroorganismer. Den mikrokoloniske vækstanalyse, der er beskrevet her, kan generere individuelle vækstratemålinger for ~ 105 mikrokolonier pr. Eksperiment. Denne analyse giver derfor en stærk protokol til at studere gær evolutionære genetik og genomik. Det egner sig særligt godt til at teste, hvordan variation i populationer af genetisk identiske enkeltceller genereres, vedligeholdes og bidrager til befolkningens egnethed7,8,9,10.
Den metode, der er beskrevet her (Figur 1) bruger periodisk optagne, lav-forstørrelse brightfield billeder af celler vokser i flydende medier på en 96- eller 384-godt glas-bund plade til at spore vækst i mikrokolonier. Cellerne klæber til lectin concanavalin A, som dækker bunden af mikroskoppladen og danner todimensionelle kolonier. Fordi mikrokolonier vokser i et monolayer, mikrokoloni område er stærkt korreleret med celle nummer7. Derfor kan nøjagtige skøn over mikrokolonivækst og forsinkelsestid genereres med brugerdefineret billedanalysesoftware, der sporer ændringshastigheden for arealet af hver mikrokoloni. Desuden kan den eksperimentelle opsætning overvåge overflod og endda de subcellulære lokaliseringer af fluorescerende mærkede proteiner udtrykt i disse mikrokolonier. Downstream-behandling af data fra denne mikrokoloniske vækstanalyse kan opnås ved brugerdefineret analyse eller ved eksisterende billedanalysesoftware, såsom Processing Images Easily (PIE)11, en algoritme til robust koloniområdegenkendelse og vækstanalyse med høj overførselshastighed fra lavforstørrelse, brightfield-billeder, som er tilgængelig via GitHub12.
Da vækstestimater, der stammer fra mikrokoloniens vækstanalyse, genereres af et stort antal enkeltkolonimålinger, er de ekstremt nøjagtige, idet standardfejl er flere størrelsesordener mindre end selve estimaterne for et eksperiment af rimelig størrelse. Derfor er analysens kraft til at opdage vækstrateforskelle mellem forskellige genotyper, behandlinger eller miljøforhold høj. Multiwell-pladeformatet gør det muligt at sammenligne mange forskellige miljø- og genotypekombinationer i et enkelt eksperiment. Hvis stammer udgør udtryk for forskellige fluorescerende markører, kan de blandes i samme brønd og skelnes af efterfølgende billedanalyse, hvilket kan øge magten yderligere ved at tillade godt-by-well data normalisering.
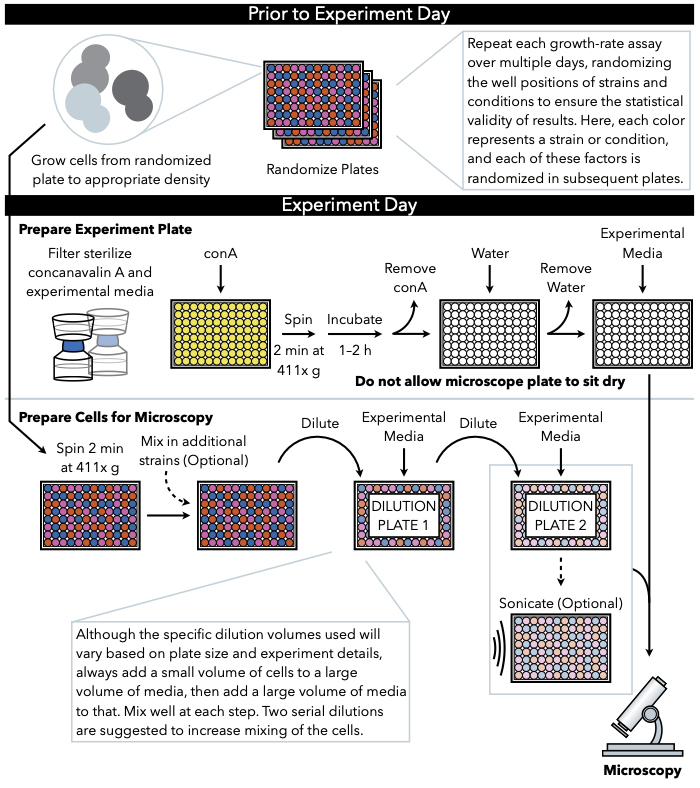
Figur 1: Skematisk repræsentation af protokollen. Denne protokol følger to hovedtrin, som er forberedelsen af forsøgspladen og forberedelsen af cellerne til billedet. Randomisering af plader og vækst af celler bør udføres før og op til eksperimentdagen. Gentagen blanding af celler på hvert trin under fortynding er afgørende i trinene, indtil plating, og derfor anbefales det først at forberede forsøgspladen, så den er klar til plating umiddelbart efter afslutningen af cellefortynding. Klik her for at se en større version af dette tal.
Protocol
Representative Results
Discussion
Den protokol, der er beskrevet her, er en alsidig analyse, der gør det muligt at overvåge cellevækst og genekspression samtidigt på niveau med individuelle mikrokolonier. Ved at kombinere disse to modaliteter giver unikke biologiske indsigter. For eksempel har tidligere arbejde brugt denne analyse til at vise en negativ korrelation mellem udtryk for TSL1-genet og mikrokolonivæksten i isogene wildtypeceller ved at måle både samtidigt7,10. Det er og…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vi takker Naomi Ziv, Sasha Levy og Shuang Li for deres bidrag til udviklingen af denne protokol, David Gresham for delt udstyr og Marissa Knoll for hjælp til videoproduktion. Dette arbejde blev støttet af National Institutes of Health tilskud R35GM118170.
Materials
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are reccomended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
References
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Genetics. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Genetics. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization–a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).

