Imagerie en direct à haut débit des microcolonies pour mesurer l’hétérogénéité de la croissance et de l’expression des gènes
Summary
Les phénotypes de croissance de levure sont précisément mesurés par l’imagerie time-lapse très parallèle des cellules immobilisées se développent dans les microcolonies. Simultanément, la tolérance au stress, l’expression des protéines et la localisation des protéines peuvent être surveillées, générant des ensembles de données intégrés pour étudier comment les différences environnementales et génétiques, ainsi que l’hétérogénéité de l’expression génique entre les cellules isogéniques, modulent la croissance.
Abstract
Des mesures précises de l’hétérogénéité entre les souches et à l’intérieur de la souche dans les taux de croissance microbienne sont essentielles pour comprendre les apports génétiques et environnementaux dans la tolérance au stress, la pathogénie et d’autres composantes clés de la condition physique. Ce manuscrit décrit un test au microscope qui suit environ 105microcoloniesSaccharomyces cerevisiae par expérience. Après l’imagerie automatisée en accéléré de levure immobilisée dans une plaque multiwell, les taux de croissance de la microcolonie sont facilement analysés à l’aide d’un logiciel d’analyse d’image personnalisé. Pour chaque microcolonie, l’expression et la localisation des protéines fluorescentes et la survie du stress aigu peuvent également être surveillées. Cet essai permet une estimation précise des taux de croissance moyens des souches, ainsi qu’une mesure complète de l’hétérogénéité de la croissance, de l’expression des gènes et de la tolérance au stress au sein des populations clonales.
Introduction
Les phénotypes de croissance contribuent de façon critique à la condition physique de levure. La sélection naturelle peut faire une distinction efficace entre les lignées dont les taux de croissance diffèrent par l’inverse de la taille effective de la population, qui peut dépasser 108 individus1. En outre, la variabilité des taux de croissance entre les individus au sein d’une population est un paramètre d’évolution pertinent, car il peut servir de base à des stratégies de survie telles que lacouverture des paris 2,3,4,5,6. Par conséquent, les analyses qui permettent des mesures très précises des phénotypes de croissance et de leurs distributions sont essentielles pour l’étude des micro-organismes. L’analyse de croissance de la microcolonie décrite ici peut générer des mesures individuelles du taux de croissance pour ~105 microcolonies par expérience. Cet essai fournit donc un protocole puissant pour étudier la génétique évolutive de levure et la génomique. Il se prête particulièrement bien à tester comment la variabilité au sein des populations de cellules individuelles génétiquement identiques est générée, maintenue et contribue à la condition physique de la population7,8,9,10.
La méthode décrite ici (figure 1) utilise périodiquement capturées, images de faible grossissement brightfield des cellules qui poussent dans les médias liquides sur une plaque de fond de verre de 96 ou 384 puits pour suivre la croissance en microcolonies. Les cellules adhèrent à la concanavalin A de lectine, qui recouvre le fond de la plaque de microscope, et forment des colonies bidimensionnelles. Puisque les microcolonies se développent dans un monocouche, la zone de microcolonie est fortement corrélée avec le numéro de cellule7. Par conséquent, des estimations précises du taux de croissance de la microcolonie et du temps de décalage peuvent être générées avec un logiciel personnalisé d’analyse d’image qui suit le taux de changement de la zone de chaque microcolonie. En outre, la configuration expérimentale peut surveiller les abondances et même les localisations subcellulaires des protéines étiquetées fluorescentes exprimées dans ces microcolonies. Le traitement en aval des données de cet essai de croissance de microcolonie peut être réalisé par analyse personnalisée ou par des logiciels existants d’analyse d’image, tels que Processing Images Easily (PIE)11, un algorithme pour la reconnaissance robuste de la zone de colonie et l’analyse de croissance à haut débit à partir de faible grossissement, images brightfield, qui est disponible via GitHub12.
Étant donné que les estimations du taux de croissance dérivées de l’analyse de microcolonie-croissance sont générées à partir d’un grand nombre de mesures d’une seule colonie, elles sont extrêmement précises, avec des erreurs standard plusieurs ordres de grandeur plus petits que les estimations elles-mêmes pour une expérience de taille raisonnable. Par conséquent, la puissance de l’analyse pour détecter les différences de taux de croissance entre les différents génotypes, traitements ou conditions environnementales est élevée. Le format multiwell-plaque permet de comparer de nombreuses combinaisons d’environnement et de génotypes différentes en une seule expérience. Si les souches expriment constitutivement différents marqueurs fluorescents, elles peuvent être mélangées dans le même puits et distinguées par une analyse d’image ultérieure, ce qui pourrait augmenter encore la puissance en permettant une normalisation des données bien par puits.
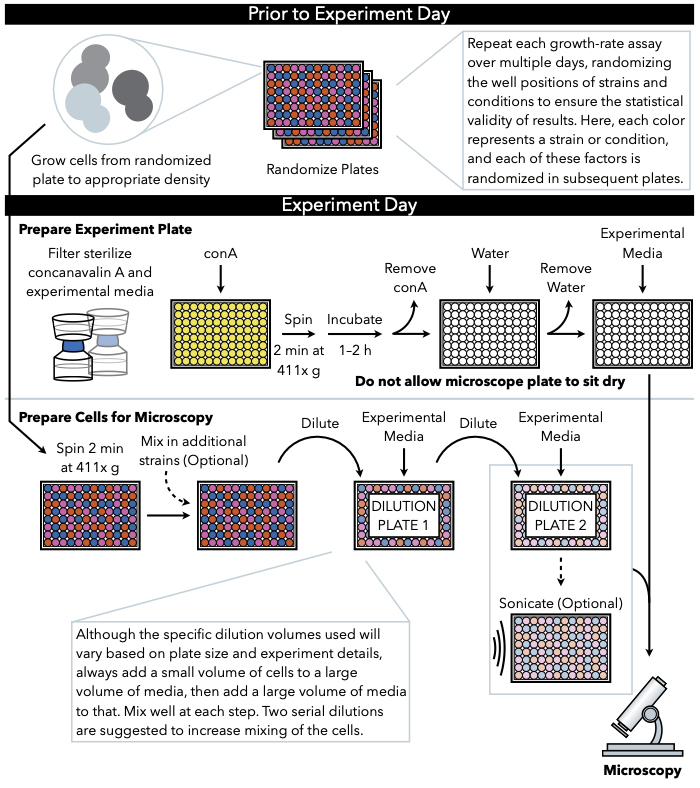
Figure 1: Représentation schématique du protocole. Ce protocole suit deux étapes principales, qui sont la préparation de la plaque expérimentale et la préparation des cellules à l’image. La randomisation des plaques et la croissance des cellules doivent être effectuées avant et jusqu’au jour de l’expérience. Le mélange répété des cellules à chaque étape pendant la dilution est impératif dans les étapes jusqu’au placage, et donc la préparation de la plaque expérimentale d’abord est recommandée afin qu’elle soit prête pour le placage immédiatement après l’achèvement de la dilution cellulaire. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
Protocol
Representative Results
Discussion
Le protocole décrit ici est un test polyvalent qui permet de surveiller simultanément la croissance cellulaire et l’expression des gènes au niveau des microcolonies individuelles. La combinaison de ces deux modalités donne des idées biologiques uniques. Par exemple, des travaux antérieurs ont utilisé cet essai pour montrer une corrélation négative entre l’expression du gène TSL1 et le taux de croissance de la microcolonie dans les cellules isogéniques de type sauvage en mesurant les<sup class="xre…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Nous remercions Naomi Ziv, Sasha Levy et Shuang Li pour leur contribution au développement de ce protocole, David Gresham pour l’équipement partagé, et Marissa Knoll pour leur aide à la production vidéo. Ces travaux ont été soutenus par la subvention R35GM118170 des National Institutes of Health.
Materials
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are reccomended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
References
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Genetics. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Genetics. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization–a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).

