Imagem ao vivo de alta taxa de microcolônias para medir heterogeneidade no crescimento e expressão genética
Summary
Fenótipos de crescimento de leveduras são precisamente medidos através de imagens de lapso de tempo altamente paralelas de células imobilizadas crescendo em microcolônias. Simultaneamente, a tolerância ao estresse, a expressão proteica e a localização da proteína podem ser monitoradas, gerando conjuntos de dados integrados para estudar como as diferenças ambientais e genéticas, bem como a heterogeneidade de expressão genética entre as células isogênicas, modulam o crescimento.
Abstract
Medições precisas da heterogeneidade entre e dentro da tensão nas taxas de crescimento microbiano são essenciais para a compreensão de insumos genéticos e ambientais na tolerância ao estresse, patogenicidade e outros componentes-chave da aptidão. Este manuscrito descreve um ensaio baseado em microscópio que rastreia aproximadamente 105 microcolônias de Saccharomyces por experimento. Após a imagem automatizada de lapso de tempo de levedura imobilizada em uma placa multiwell, as taxas de crescimento de microcolonia são facilmente analisadas com software personalizado de análise de imagem. Para cada microcolonia, a expressão e a localização de proteínas fluorescentes e a sobrevivência do estresse agudo também podem ser monitoradas. Este ensaio permite uma estimativa precisa das taxas médias de crescimento das cepas, bem como uma medição abrangente da heterogeneidade no crescimento, expressão genética e tolerância ao estresse dentro das populações clonais.
Introduction
Fenótipos de crescimento contribuem criticamente para o condicionamento físico da levedura. A seleção natural pode distinguir eficientemente entre linhagens com taxas de crescimento diferidas pelo inverso do tamanho populacional efetivo, que pode exceder 108 indivíduos1. Além disso, a variabilidade das taxas de crescimento entre indivíduos dentro de uma população é um parâmetro evolutivamente relevante, pois pode servir de base para estratégias de sobrevivência como cobertura de apostas2,3,4,5,6. Portanto, ensaios que permitem medições altamente precisas de fenótipos de crescimento e suas distribuições são fundamentais para o estudo de microrganismos. O ensaio de crescimento da microcolônia descrito aqui pode gerar medições individuais de taxa de crescimento para ~105 microcolônias por experimento. Este ensaio fornece, portanto, um protocolo poderoso para estudar a genética evolutiva da levedura e a genômica. Ele se presta particularmente bem a testar como a variabilidade dentro de populações de células únicas geneticamente idênticas é gerada, mantida e contribui para a aptidão populacional7,8,9,10.
O método descrito aqui (Figura 1) usa imagens de campo brilhante de baixa ampliação capturadas periodicamente de células crescendo em mídia líquida em uma placa de fundo de vidro de 96 ou 384 poços para rastrear o crescimento em microcolônias. As células aderem à lectina concanavalina A, que reveste a parte inferior da placa de microscópio, e formam colônias bidimensionais. Como as microcolônias crescem em uma monocamada, a área de microcolonia é altamente correlacionada com o celular número7. Portanto, estimativas precisas da taxa de crescimento da microcolonia e do tempo de defasagem podem ser geradas com software personalizado de análise de imagem que rastreia a taxa de mudança da área de cada microcolonia. Além disso, a configuração experimental pode monitorar as abundâncias e até mesmo as localizações subcelulares de proteínas fluorescentes rotuladas expressas nessas microcolônias. O processamento a jusante de dados deste ensaio de crescimento de microcolonia pode ser alcançado por meio de análise personalizada ou por softwares de análise de imagem existentes, como Processing Images Easily (PIE)11, um algoritmo para reconhecimento robusto da área de colônia e análise de crescimento de alto rendimento a partir de imagens de baixa ampliação e brightfield, que está disponível via GitHub12.
Como as estimativas de taxa de crescimento derivadas do ensaio de crescimento da microcolonia são geradas a partir de um grande número de medições de uma única colônia, elas são extremamente precisas, com erros padrão várias ordens de magnitude menores do que as próprias estimativas para um experimento de tamanho razoável. Portanto, o poder do ensaio para detectar diferenças de taxa de crescimento entre diferentes genótipos, tratamentos ou condições ambientais é alto. O formato multiwell-plate permite que inúmeras combinações diferentes de ambiente e genótipo sejam comparadas em um único experimento. Se as cepas expressam com constitutivamente diferentes marcadores fluorescentes, elas podem ser misturadas no mesmo poço e distinguidas pela análise de imagem subsequente, o que poderia aumentar ainda mais a energia, permitindo a normalização dos dados bem-por-bem.
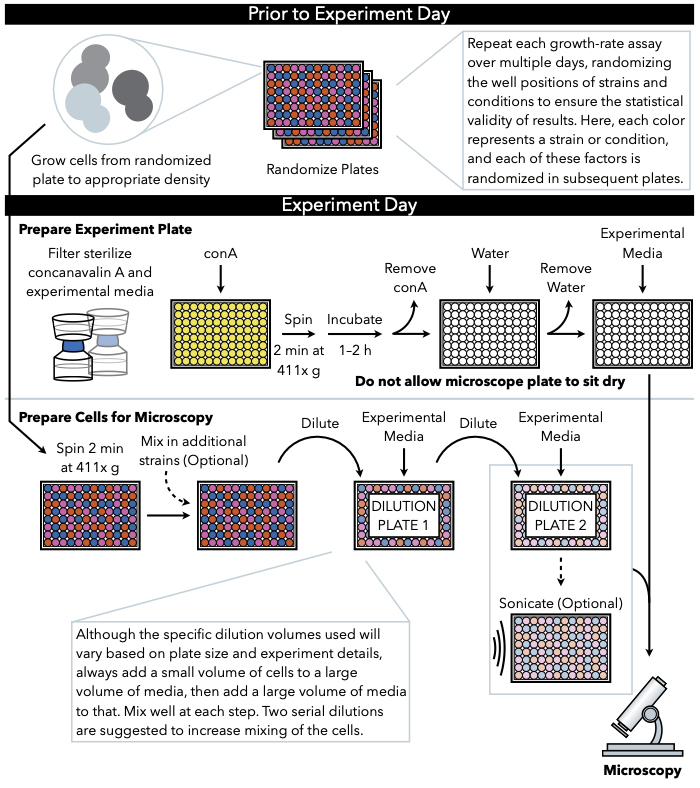
Figura 1: Representação esquemática do protocolo. Este protocolo segue dois passos principais, que são a preparação da placa experimental e a preparação das células para a imagem. A randomização das placas e o crescimento das células devem ser realizados antes e levando ao dia do experimento. A mistura repetida de células em cada etapa durante a diluição é imperativa nas etapas até o revestimento e, portanto, a preparação da placa experimental primeiro é recomendada para que ela esteja pronta para chapeamento imediatamente após a conclusão da diluição celular. Clique aqui para ver uma versão maior desta figura.
Protocol
Representative Results
Discussion
O protocolo descrito aqui é um ensaio versátil que permite que o crescimento celular e a expressão genética sejam monitorados simultaneamente ao nível de microcolônias individuais. A combinação dessas duas modalidades produz insights biológicos únicos. Por exemplo, trabalhos anteriores usaram este ensaio para mostrar uma correlação negativa entre a expressão do gene TSL1 e a taxa de crescimento da microcolonia em células selvagens isogênicas medindo simultaneamente7,</…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Agradecemos a Naomi Ziv, Sasha Levy e Shuang Li por suas contribuições para o desenvolvimento deste protocolo, David Gresham por equipamentos compartilhados, e Marissa Knoll por ajuda na produção de vídeo. Este trabalho foi apoiado pelo Instituto Nacional de Saúde do Instituto R35GM118170.
Materials
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are reccomended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
References
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Genetics. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Genetics. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization–a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).

