Imaging live ad alta produttività di microcolonie per misurare l'eterogeneità nella crescita e nell'espressione genica
Summary
I fenotipi di crescita del lievito sono misurati con precisione attraverso l’imaging time-lapse altamente parallelo di cellule immobilizzate che crescono in microcolonie. Allo stesso tempo, la tolleranza allo stress, l’espressione proteica e la localizzazione delle proteine possono essere monitorate, generando set di dati integrati per studiare come le differenze ambientali e genetiche, così come l’eterogeneità gene-espressione tra le cellule isogeniche, modulano la crescita.
Abstract
Misurazioni precise dell’eterogeneità tra e all’interno dello sforzo nei tassi di crescita microbica sono essenziali per comprendere gli input genetici e ambientali nella tolleranza allo stress, nella patogenicità e in altri componenti chiave dell’idoneità. Questo manoscritto descrive un saggio basato sul microscopio che tiene traccia di circa 105 microcoloni Saccharomyces cerevisiae per esperimento. Dopo l’imaging automatizzato time-lapse del lievito immobilizzato in una piastra multiwell, i tassi di crescita dei microcoloni vengono facilmente analizzati con un software di analisi delle immagini personalizzato. Per ogni microcolonia, è anche possibile monitorare l’espressione e la localizzazione di proteine fluorescenti e la sopravvivenza dello stress acuto. Questo saggio consente una stima precisa dei tassi di crescita medi dei ceppi, nonché una misurazione completa dell’eterogeneità nella crescita, nell’espressione genica e nella tolleranza allo stress all’interno delle popolazioni clonali.
Introduction
I fenotipi di crescita contribuiscono in modo critico alla forma fisica del lievito. La selezione naturale può distinguere in modo efficiente tra lignaggi con tassi di crescita diversi dall’inverso della dimensione effettiva della popolazione, che può superare i 108 individui1. Inoltre, la variabilità dei tassi di crescita tra gli individui all’interno di una popolazione è un parametro evolutivamente rilevante, in quanto può servire come base per strategie di sopravvivenza come la copertura dellepuntate 2,3,4,5,6. Pertanto, i test che consentono misurazioni altamente accurate dei fenotipi di crescita e delle loro distribuzioni sono fondamentali per lo studio dei microrganismi. Il saggio di crescita del microcolonio qui descritto può generare misurazioni individuali del tasso di crescita per ~10 5 microcolonie per esperimento. Questo saggio fornisce quindi un potente protocollo per studiare la genetica evolutiva e la genomica del lievito. Si presta particolarmente bene a testare come la variabilità all’interno delle popolazioni di singole cellule geneticamente identiche viene generata, mantenuta e contribuisce alla forma fisicadella popolazione 7,8,9,10.
Il metodo qui descritto (Figura 1) utilizza immagini a campo luminoso a basso ingrandimento catturate periodicamente di cellule che crescono in mezzi liquidi su una piastra inferiore in vetro da 96 o 384 pozze di vetro per tenere traccia della crescita in microcolonie. Le cellule aderiscono alla lettalina concanavalina A, che rivestise il fondo della piastra del microscopio e forma colonie bidimensionali. Poiché le microcolonie crescono in un monostrato, l’area della microcolonia è altamente correlata con la cella numero7. Pertanto, è possibile generare stime accurate del tasso di crescita del microcolony e del tempo di ritardo con un software di analisi delle immagini personalizzato che tiene traccia del tasso di cambiamento dell’area di ogni microcolony. Inoltre, la configurazione sperimentale può monitorare l’abbondanza e persino le localizzazioni subcellulari di proteine etichettate fluorescentmente espresse in queste microcolonie. L’elaborazione a valle dei dati di questo test di crescita della microcolonia può essere ottenuta mediante analisi personalizzate o con il software di analisi delle immagini esistente, come Processing Images Easily (PIE)11, un algoritmo per il riconoscimento robusto dell’area della colonia e l’analisi della crescita ad alta produttività da immagini a basso ingrandimento e luminose, disponibile tramite GitHub12.
Poiché le stime del tasso di crescita derivate dal saggio di crescita della microcolonia sono generate da un gran numero di misurazioni a colonia singola, sono estremamente accurate, con errori standard di diversi ordini di grandezza più piccoli delle stime stesse per un esperimento di dimensioni ragionevoli. Pertanto, il potere del saggio di rilevare differenze di tasso di crescita tra diversi genotipi, trattamenti o condizioni ambientali è elevato. Il formato multiwell-plate consente di confrontare numerose combinazioni di ambiente e genotipo diverse in un unico esperimento. Se i ceppi esprimono in modo costitutivo marcatori fluorescenti diversi, possono essere mescolati nello stesso pozzo e distinti dalla successiva analisi dell’immagine, che potrebbe aumentare ulteriormente la potenza consentendo la normalizzazione dei dati pozzo per pozzo.
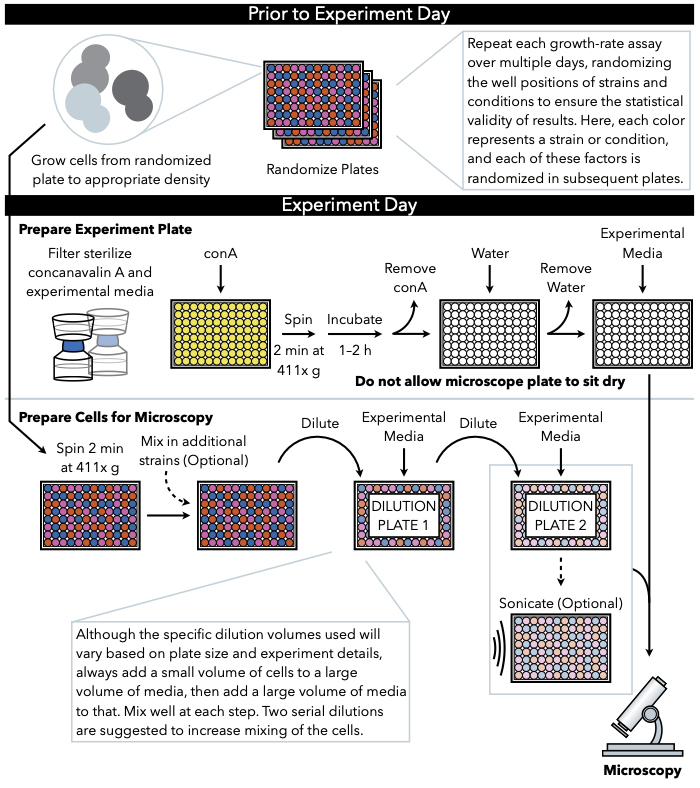
Figura 1: Rappresentazione schematica del protocollo. Questo protocollo segue due fasi principali, che sono la preparazione della piastra sperimentale e la preparazione delle cellule all’immagine. La randomizzazione delle piastre e la crescita delle cellule devono essere condotte prima e prima del giorno dell’esperimento. La miscelazione ripetuta delle cellule in ogni fase durante la diluizione è imperativa nei passaggi fino alla placcatura, e quindi si consiglia di preparare prima la piastra sperimentale in modo che sia pronta per la placcatura immediatamente dopo il completamento della diluizione cellulare. Clicca qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
Il protocollo qui descritto è un saggio versatile che consente di monitorare simultaneamente la crescita cellulare e l’espressione genica a livello delle singole microcolonie. La combinazione di queste due modalità produce intuizioni biologiche uniche. Ad esempio, lavori precedenti hanno usato questo saggio per mostrare una correlazione negativa tra l’espressione del gene TSL1 e il tasso di crescita della microcolonia nelle cellule di tipo selvatico isogenico misurandoentrambi contemporaneame…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Ringraziamo Naomi Ziv, Sasha Levy e Shuang Li per i loro contributi allo sviluppo di questo protocollo, David Gresham per le attrezzature condivise e Marissa Knoll per l’aiuto nella produzione video. Questo lavoro è stato sostenuto dalla sovvenzione dei National Institutes of Health R35GM118170.
Materials
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are reccomended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
References
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Genetics. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Genetics. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization–a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).

