Live imaging av mikrokolonier med hög genomströmning för att mäta heterogenitet i tillväxt och genuttryck
Summary
Jäst tillväxt fenotyper mäts exakt genom mycket parallell tidsfördröjning imaging av immobiliserade celler växer till mikrokolon. Samtidigt kan stresstolerans, proteinuttryck och proteinlokalisering övervakas, vilket genererar integrerade datamängder för att studera hur miljömässiga och genetiska skillnader, liksom genuttrycks heterogenitet bland isogena celler, modulerar tillväxt.
Abstract
Exakta mätningar av mellan- och inomstams heterogenitet i mikrobiell tillväxthastighet är avgörande för att förstå genetiska och miljömässiga indata i stresstolerans, patogenicitet och andra viktiga komponenter i fitness. Detta manuskript beskriver en mikroskopbaserad analys som spårar cirka 105 Saccharomyces cerevisiae mikrokolonier per experiment. Efter automatiserad timelapse-avbildning av jäst immobiliserad i en multiwellplatta analyseras mikrokolonis tillväxthastigheter enkelt med anpassad bildanalysprogramvara. För varje mikrokoloni kan uttryck och lokalisering av fluorescerande proteiner och överlevnad av akut stress också övervakas. Denna analys möjliggör exakt uppskattning av stammars genomsnittliga tillväxttakt, liksom omfattande mätning av heterogenitet i tillväxt, genuttryck och stresstolerans inom klonurpopulationer.
Introduction
Tillväxt fenotyper bidrar kritiskt till jäst fitness. Naturligt urval kan effektivt skilja mellan härstamningar med tillväxthastigheter som skiljer sig åt genom inversen av den effektiva befolkningsstorleken, som kan överstiga 108 individer1. Dessutom är variabilitet av tillväxttakten bland individer inom en population en evolutionärt relevant parameter, eftersom den kan tjäna som grund för överlevnadsstrategier som betsäkring2,3,4,5,6. Därför är analyser som möjliggör mycket exakta mätningar av tillväxt fenotyper och deras fördelningar avgörande för studier av mikroorganismer. Den mikrokoloniska tillväxtanalysen som beskrivs här kan generera individuella tillväxthastighetsmätningar för ~10 5 mikrokolonier per experiment. Denna analys ger därför ett kraftfullt protokoll för att studera jäst evolutionär genetik och genomik. Det lämpar sig särskilt bra för att testa hur variationer inom populationer av genetiskt identiska enstaka celler genereras, underhålls och bidrar till befolkningens kondition7,8,9,10.
Metoden som beskrivs här (Figur 1) använder periodiskt fångade, lågförstoring brightfield bilder av celler som växer i flytande media på en 96- eller 384-brunn glasbottenplatta för att spåra tillväxt till mikrokolonier. Cellerna klibbar sig vid inktinkonanavalin A, som täcker botten av mikroskopplattan och bildar tvådimensionella kolonier. Eftersom mikrokolonerna växer i ett monoskikt är mikrokoloniområdet mycket korrelerat med cellnummer7. Därför kan exakta uppskattningar av mikrokolonisk tillväxthastighet och fördröjningstid genereras med anpassad bildanalysprogramvara som spårar förändringshastigheten för området för varje mikrokoloni. Dessutom kan den experimentella inställningen övervaka överflöd och till och med subcellulära lokaliseringar av fluorescerande märkta proteiner uttryckta i dessa mikrokolonier. Nedströms bearbetning av data från denna mikrofärgade tillväxtanalys kan uppnås genom anpassad analys eller genom befintlig bildanalysprogramvara, till exempel Processing Images Easily (PIE)11, en algoritm för robust igenkänning av koloniområdet och tillväxtanalys med hög genomströmning från lågförstoring, brightfield-bilder, som är tillgänglig via GitHub12.
Eftersom tillväxthastighetsuppskattningar som härrör från mikrokolonitillväxtanalysen genereras från ett stort antal enkolonimätningar är de extremt exakta, med standardfel som är flera storleksordningar mindre än själva uppskattningarna för ett experiment av rimlig storlek. Därför är kraften i analysen för att upptäcka tillväxthastighetsskillnader mellan olika genotyper, behandlingar eller miljöförhållanden hög. Multiwell-plate-formatet gör att många olika miljö- och genotypkombinationer kan jämföras i ett enda experiment. Om stammar utgör olika fluorescerande markörer kan de blandas i samma brunn och särskiljas genom efterföljande bildanalys, vilket kan öka kraften ytterligare genom att tillåta väl-för-brunn-datanormalisering.
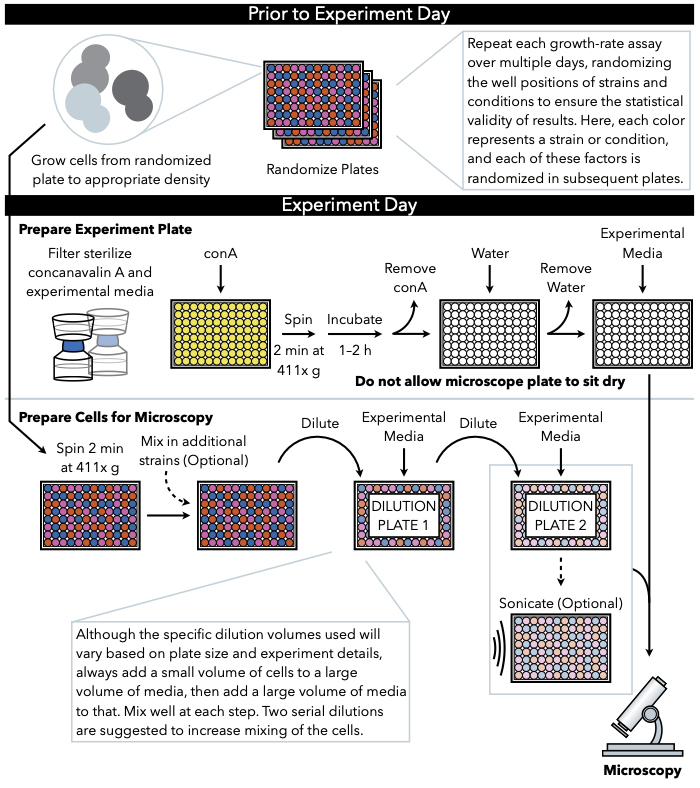
Figur 1: Schematisk representation av protokollet. Detta protokoll följer två huvudsteg, som är förberedelsen av den experimentella plattan och beredningen av cellerna till bild. Randomisering av plattor och tillväxt av celler bör utföras före och fram till experimentdagen. Upprepad blandning av celler i varje steg under utspädning är absolut nödvändigt i stegen fram till plätering, och därför rekommenderas förberedelse av experimentplattan först så att den är klar för plätering omedelbart efter avslutad cellutspädning. Klicka här om du vill visa en större version av den här figuren.
Protocol
Representative Results
Discussion
Protokollet som beskrivs här är en mångsidig analys som gör det möjligt att övervaka celltillväxt och genuttryck samtidigt på nivån för enskilda mikrokolonier. Att kombinera dessa två metoder ger unika biologiska insikter. Till exempel har tidigare arbete använt denna analys för att visa en negativ korrelation mellan uttryck av TSL1-genen och mikrokolonitillväxthastighet i isogena vilda typceller genom att mätabåda samtidigt 7,10. Det är…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vi tackar Naomi Ziv, Sasha Levy och Shuang Li för deras bidrag till att utveckla detta protokoll, David Gresham för delad utrustning och Marissa Knoll för hjälp med videoproduktion. Detta arbete stöddes av National Institutes of Health grant R35GM118170.
Materials
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are reccomended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
References
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Genetics. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Genetics. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization–a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).

