DNA-gebonden RNA-polymerase voor programmeerbare in vitro transcriptie en moleculaire berekening
Summary
We beschrijven de engineering van een nieuw DNA-gebonden T7-RNA-polymerase om in vitro transcriptiereacties te reguleren. We bespreken de stappen voor eiwitsynthese en karakterisering, valideren proof-of-concept transcriptionele regulatie en bespreken de toepassingen ervan in moleculaire computing, diagnostiek en moleculaire informatieverwerking.
Abstract
DNA-nanotechnologie maakt programmeerbare zelfassemblage van nucleïnezuren in door de gebruiker voorgeschreven vormen en dynamieken voor diverse toepassingen mogelijk. Dit werk toont aan dat concepten uit de DNA-nanotechnologie kunnen worden gebruikt om de enzymatische activiteit van het faag-afgeleide T7-RNA-polymerase (RNAP) te programmeren en schaalbare synthetische genregulerende netwerken te bouwen. Ten eerste wordt een oligonucleotide-gebonden T7 RNAP ontworpen via expressie van een N-terminale SNAP-gelabelde RNAP en daaropvolgende chemische koppeling van de SNAP-tag met een benzylguanine (BG)-gemodificeerd oligonucleotide. Vervolgens wordt nucleïnezuurstrengverplaatsing gebruikt om polymerasetranscriptie op aanvraag te programmeren. Bovendien kunnen hulpnucleïnezuurassemblages worden gebruikt als “kunstmatige transcriptiefactoren” om de interacties tussen de DNA-geprogrammeerde T7 RNAP met zijn DNA-sjablonen te reguleren. Dit in vitro transcriptie regulerende mechanisme kan een verscheidenheid aan circuitgedragingen implementeren, zoals digitale logica, feedback, cascadering en multiplexing. De composability van deze genregulerende architectuur vergemakkelijkt ontwerpabstractie, standaardisatie en schaling. Deze functies zullen de snelle prototyping van in vitro genetische apparaten mogelijk maken voor toepassingen zoals bio-sensing, ziektedetectie en gegevensopslag.
Introduction
DNA-computing maakt gebruik van een set ontworpen oligonucleotiden als medium voor berekening. Deze oligonucleotiden zijn geprogrammeerd met sequenties om dynamisch te assembleren volgens door de gebruiker gespecificeerde logica en te reageren op specifieke nucleïnezuurinputs. In proof-of-concept studies bestaat de output van de berekening meestal uit een set fluorescerend gelabelde oligonucleotiden die kunnen worden gedetecteerd via gel-elektroforese of fluorescentieplaatlezers. In de afgelopen 30 jaar zijn steeds complexere DNA-computationele circuits aangetoond, zoals verschillende digitale logische cascades, chemische reactienetwerken en neurale netwerken1,2,3. Om te helpen bij de voorbereiding van deze DNA-circuits, zijn wiskundige modellen gebruikt om de functionaliteit van synthetische gencircuits te voorspellen4,5,en computationele hulpmiddelen zijn ontwikkeld voor orthogonaal DNA-sequentieontwerp6,7,8,9,10 . In vergelijking met op silicium gebaseerde computers omvatten de voordelen van DNA-computers hun vermogen om rechtstreeks met biomoleculen te communiceren, in oplossing te werken bij afwezigheid van een voeding, evenals hun algehele compactheid en stabiliteit. Met de komst van next-generation sequencing zijn de kosten van het synthetiseren van DNA-computers de afgelopen twee decennia sneller gedaald dan de wet van Moore11. Toepassingen van dergelijke DNA-gebaseerde computers beginnen nu op te komen, zoals voor ziektediagnose12,13,voor het aandrijven van moleculaire biofysica14en als gegevensopslagplatforms15.
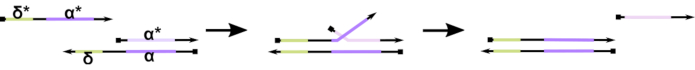
Figuur 1: Mechanisme van door de retentie gemedieerde verplaatsing van DNA-strengen. De toehold, δ, is een vrije, ongebonden reeks op een gedeeltelijke duplex. Wanneer een complementair domein (δ*) op een tweede streng wordt geïntroduceerd, dient het vrije δ domein als houvast voor hybridisatie, waardoor de rest van de streng (ɑ*) zijn concurrent langzaam kan verdringen door een ritsende / unzipping omkeerbare reactie die bekend staat als strengmigratie. Naarmate de lengte van δ toeneemt, neemt de ΔG voor de voorwaartse reactie af en vindt verplaatsing gemakkelijker plaats. Klik hier om een grotere versie van deze figuur te bekijken.
Tot op heden gebruiken de meeste DNA-computers een gevestigd motief op het gebied van dynamische DNA-nanotechnologie dat bekend staat als teenhold-gemedieerde DNA-strengverplaatsing (TMDSD, figuur 1)16. Dit motief bestaat uit een gedeeltelijk dubbelstrengs DNA (dsDNA) duplex met korte “teenvaste” overhangen (d.w.z. 7- tot 10 nucleotiden (nt)). Nucleïnezuur “input” strengen kunnen interageren met de partiële duplexen door de teen. Dit leidt tot de verplaatsing van een van de strengen van de gedeeltelijke duplex, en deze bevrijde streng kan dan dienen als input voor stroomafwaartse gedeeltelijke duplexen. TMDSD maakt dus signaalcassering en informatieverwerking mogelijk. In principe kunnen orthogonale TMDSD-motieven onafhankelijk in oplossing werken, waardoor parallelle informatieverwerking mogelijk is. Er zijn een aantal variaties geweest op de TMDSD-reactie, zoals teenhold-gemedieerde DNA-strenguitwisseling (TMDSE)17,“lekloze” teengrepen met dubbellangedomeinen 18,sequentie-niet-overeenkomende teengrepen19en “handhold”-gemedieerde strengverplaatsing20. Deze innovatieve ontwerpprincipes maken nauwkeuriger afgestemde TMDSD-energetica en dynamiek mogelijk voor het verbeteren van dna-rekenprestaties.
Synthetische gencircuits, zoals transcriptionele gencircuits, zijn ook in staat om21,22,23teberekenen. Deze circuits worden gereguleerd door eiwittranscriptiefactoren, die transcriptie van een gen activeren of onderdrukken door zich te binden aan specifieke regulerende DNA-elementen. In vergelijking met DNA-gebaseerde circuits hebben transcriptionele circuits verschillende voordelen. Ten eerste heeft enzymatische transcriptie een veel hogere omloopsnelheid dan bestaande katalytische DNA-circuits, waardoor meer kopieën van de output per enkele kopie van de ingang worden gegenereerd en een efficiëntere manier van signaalversterking wordt geboden. Bovendien kunnen transcriptionele circuits verschillende functionele moleculen produceren, zoals aptameren of boodschapper-RNA (mRNA) die coderen voor therapeutische eiwitten, als berekeningsoutputs, die voor verschillende toepassingen kunnen worden gebruikt. Een belangrijke beperking van de huidige transcriptionele circuits is echter hun gebrek aan schaalbaarheid. Dit komt omdat er een zeer beperkte set orthogonale eiwitgebaseerde transcriptiefactoren is en het de novo ontwerp van nieuwe eiwittranscriptiefactoren technisch uitdagend en tijdrovend blijft.
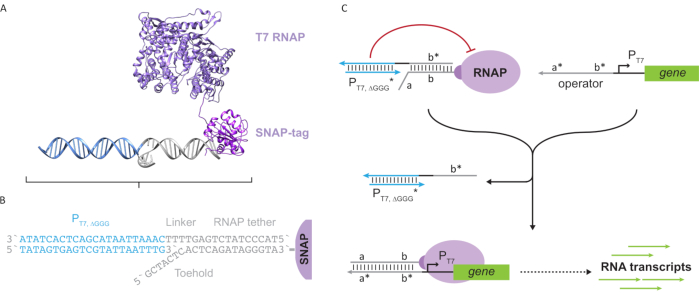
Figuur 2: Abstractie en mechanisme van “tether” en “cage” polymerase complex. (A en B) Een oligonucleotide tether wordt enzymatisch gelabeld op een T7 polymerase door middel van de SNAP-tag reactie. Een kooi bestaande uit een “faux” T7-promotor met een tether-complementoverhang maakt het mogelijk om te hybridiseren naar de tether en transcriptionele activiteit te blokkeren. (C) Wanneer de operator (a*b*) aanwezig is, bindt hij zich aan de teengreep op de oligonucleotide tether (ab) en verplaatst het b* gebied van de kooi, waardoor transcriptie kan plaatsvinden. Dit cijfer is aangepast van Chou en Shih27. Afkortingen: RNAP = RNA polymerase. Klik hier om een grotere versie van deze figuur te bekijken.
Dit artikel introduceert een nieuwe bouwsteen voor moleculaire computing die de functionaliteiten van transcriptionele circuits combineert met de schaalbaarheid van dna-gebaseerde circuits. Deze bouwsteen is een T7 RNAP covalent bevestigd met een enkelstrengs DNA-tether(Figuur 2A). Om deze DNA-gebonden T7 RNAP te synthetiseren, werd het polymerase gefuseerd tot een N-terminal SNAP-tag24 en recombinant uitgedrukt in Escherichia coli. De SNAP-tag werd vervolgens gereageerd met een oligonucleotide gefunctionaliseerd met het BG-substraat. De oligonucleotide-tether maakt de positionering van moleculaire gasten in de nabijheid van het polymerase mogelijk via DNA-hybridisatie. Een van die gasten was een competitieve transcriptionele blokker die een “kooi” wordt genoemd, die bestaat uit een “faux” T7 promotor DNA-duplex zonder gen stroomafwaarts(Figuur 2B). Wanneer de kooi via zijn oligonucleotide-tether aan de RNAP wordt gebonden, blokkeert de kooi de polymeraseactiviteit door andere DNA-sjablonen voor RNAP-binding te overtreffen, waardoor de RNAP in een “OFF” -toestand wordt weergegeven(Figuur 2C).
Om het polymerase tot een “ON” -toestand te activeren, werden T7 DNA-sjablonen met enkelstrengs “operator” -domeinen stroomopwaarts van de T7-promotor van het gen ontworpen. Het operatordomein (d.w.z. domein a*b* Figuur 2C)kan worden ontworpen om de kooi via TMDSD van de RNAP te verplaatsen en de RNAP proximaal aan de T7-promotor van het gen te plaatsen, waardoor transcriptie wordt geïnitieerd. Als alternatief werden ook DNA-sjablonen ontworpen waarbij de operatorsequentie complementair was aan hulpnucleïnezuurstrengen die worden aangeduid als “kunstmatige transcriptiefactoren” (d.w.z. TFA- en TFB-strengen in figuur 3A). Wanneer beide strengen in de reactie worden geïntroduceerd, zullen ze zich op de operatorlocatie verzamelen, waardoor een nieuw pseudo-aaneengesloten domein a * b *ontstaat. Dit domein kan vervolgens de kooi verplaatsen via TMDSD om transcriptie te initiëren(Figuur 3B). Deze strengen kunnen zowel exogene als geproduceerd worden geleverd.
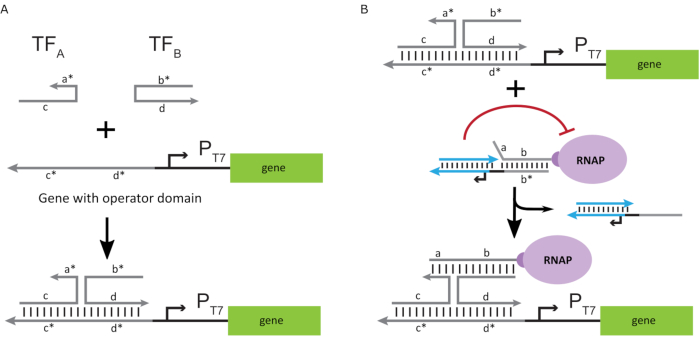
Figuur 3: Selectieve programmering van polymeraseactiviteit door middel van een driecomponentenschakelaaractivator. (A) Wanneer de transcriptiefactoren (TFA en TFB) aanwezig zijn, binden ze zich aan het operatordomein stroomopwaarts van de promotor en vormen ze een pseudo-enkelstrengssequentie ( a *b *) die in staat is de kooi te verplaatsen door middel van teengemedieerde DNA-verplaatsing. (B) Dit a*b*-domein kan de kooi via TMDSD verplaatsen om transcriptie te initiëren. Dit cijfer is aangepast van Chou en Shih27. Afkortingen: TF = transcriptiefactor; RNAP = RNA-polymerase; TMDSD = tenen-gemedieerde DNA-strengverplaatsing. Klik hier om een grotere versie van deze figuur te bekijken.
Het gebruik van op nucleïnezuur gebaseerde transcriptiefactoren voor in vitro transcriptionele regulatie maakt de schaalbare implementatie van geavanceerd circuitgedrag mogelijk, zoals digitale logica, feedback en signaalcassalisatie. Men kan bijvoorbeeld logische poortcascades bouwen door nucleïnezuursequenties zo te ontwerpen dat de transcripties van een stroomopwaarts gen een stroomafwaarts gen activeren. Een toepassing die gebruik maakt van de cascadering en multiplexing die door deze voorgestelde technologie mogelijk wordt gemaakt, is de ontwikkeling van meer geavanceerde moleculaire computercircuits voor draagbare diagnostiek en moleculaire gegevensverwerking. Bovendien kan de integratie van de moleculaire computing en de novo RNA-synthesemogelijkheden nieuwe toepassingen mogelijk maken. Een moleculair circuit kan bijvoorbeeld worden ontworpen om één of een combinatie van door de gebruiker gedefinieerde RNA’s te detecteren als inputs en output therapeutische RNA’s of mRNA’s die coderen voor functionele peptiden of eiwitten voor point-of-care medische toepassingen.
Protocol
Representative Results
Discussion
Deze studie demonstreert een dna-nanotechnologie-geïnspireerde benadering om de activiteit van T7 RNA-polymerase te beheersen door covalent een N-terminaal SNAP-gelabelde recombinant T7 RNAP te koppelen aan een BG-gefunctionaliseerd oligonucleotide, dat vervolgens werd gebruikt om TMDSD-reacties te programmeren. Door het ontwerp werd de SNAP-tag gepositioneerd op het N-eindpunt van het polymerase, omdat het C-eindpunt van wild-type T7 RNAP begraven ligt in de kern van de eiwitstructuur en belangrijke contacten legt met …
Disclosures
The authors have nothing to disclose.
Acknowledgements
L.Y.T.C erkent genereuze steun van de New Frontiers in Research Fund-Exploration (NFRF-E), de Natural Sciences and Engineering Research Council of Canada (NSERC) Discovery Grant en het Medicine by Design Initiative van de Universiteit van Toronto, dat financiering ontvangt van het Canada First Research Excellence Fund (CFREF).
Materials
| 0.5% polysorbate 20 (TWEEN 20) | BioShop | TWN510.5 | |
| 0.5M ethylenediaminetetraacetic acid (EDTA) | Bio Basic | SD8135 | |
| 10 mM sodium phosphate buffer (pH 7) | Bio Basic | PD0435 | Tablets used to make 10 mM buffer |
| 10% ammonium persulfate (APS) | Sigma Aldrich | A3678-100G | |
| 100 kDa Amicon Ultra-15 Centrifugal Filter Unit | Fisher Scientific | UFC910008 | |
| 100% acetone | Fisher Chemical | A18P4 | |
| 100% ethanol (EtOH) | House Brand | 39752-P016-EAAN | |
| 10x in vitro transcription (IVT) buffer | New England Biolabs | B9012 | |
| 10x Tris-Borate-EDTA (TBE) buffer | Bio Basic | A0026 | |
| 1M Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Sigma Aldrich | I5502-1G | |
| 1M sodium bicarbonate buffer | Sigma Aldrich | S6014-500G | |
| 1M Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 648311-1KG | |
| 1X Tris-EDTA (TE) buffer | ThermoFisher | 12090015 | |
| 2M imidazole | Sigma Aldrich | 56750-100G | |
| 2-mercaptoethanol (BME) | Sigma Aldrich | M3148 | |
| 3M sodium acetate | Bio Basic | SRB1611 | |
| 40% acrylamide (19:1) | Bio Basic | A00062 | |
| 4x LDS protein sample loading buffer | Fisher Scientific | NP0007 | |
| 5M sodium chloride (NaCl) | Bio Basic | DB0483 | |
| 5mM dithiothreitol (DTT) | Sigma Aldrich | 43815-1G | |
| 6x gel loading dye | New England Biolabs | B7024S | |
| agarose B powder | Bio Basic | AB0014 | |
| BG-GLA-NHS | New England Biolabs | S9151S | |
| BL21 competent E. coli | Addgene | C2530H | |
| BLUeye prestained protein ladder | FroggaBio | PM007-0500 | |
| bromophenol blue | Bio Basic | BDB0001 | |
| coomassie blue (SimplyBlue SafeStain) | ThermoFisher | LC6060 | |
| cyanine dye (SYBR Gold nucleic acid gel stain) | Fisher Scientific | S11494 | |
| cyanine dye (SYBR Safe nucleic acid gel stain) | Fisher Scientific | S33102 | |
| dry dimethyl sulfoxide (DMSO) | Fisher Scientific | D12345 | |
| formamide | Sigma Aldrich | F9037-100ML | |
| glycerol | Bio Basic | GB0232 | |
| kanamycin sulfate | BioShop | KAN201.5 | |
| lysogeny broth | Sigma Aldrich | L2542-500ML | |
| malachite green oxalate | Sigma Aldrich | 2437-29-8 | |
| N,N,N'N'-Tetramethylethane-1,2-diamine (TEMED) | Sigma Aldrich | T9281-25ML | |
| NuPAGE MES SDS running buffer (20x) | Fisher Scientific | LSNP0002 | |
| NuPAGE Novex 4-12% Bis-Tris gel 1.0 mm 12-well | Life Technologies | NP0322BOX | |
| oligonucleotide (cage antisense) | IDT | N/A | TATAGTGAGTCGTATTAATTTG |
| oligonucleotide (cage sense) | IDT | N/A | TCAGTCACCTATCTGTTTCAAA TTAATACGACTCACTATA |
| oligonucleotide (malachite green aptamer antisense) | IDT | N/A | GGATCCATTCGTTACCTGGCT CTCGCCAGTCGGGATCCTATA GTGAGTCGTATTACAGTTCCAT TATCGCCGTAGTTGGTGTACT |
| oligonucleotide (malachite green aptamer sense) | IDT | N/A | TAATACGACTCACTATAGGATC CCGACTGGCGAGAGCCAGGT AACGAATGGATCC |
| oligonucleotide (Transcription Factor A) | IDT | N/A | AGTACACCAACTACGAGTGAG |
| oligonucleotide (Transcription Factor B) | IDT | N/A | TCAGTCACCTATCTGGCGATAA TGGAACTG |
| oligonucleotide with 3’ Amine modification (tether) | IDT | N/A | GCTACTCACTCAGATAGGTGAC TGA/3AmMO/ |
| Pierce strong ion exchange spin columns | Fisher Scientific | 90008 | |
| plasmid encoding SNAP T7 RNAP and kanamycin resistance genes | Genscript | N/A | custom gene insert |
| protein purification column (HisPur Ni-NTA spin column) | Fisher Scientific | 88226 | |
| rNTP mix | New England Biolabs | N0466S | |
| Roche mini quick DNA spin column | Sigma Aldrich | 11814419001 | |
| Triton X-100 | Sigma Aldrich | T8787-100ML | |
| Ultra Low Range DNA ladder | Fisher Scientific | 10597012 | |
| urea | BioShop | URE001.1 |
References
- Cherry, K. M., Qian, L. Scaling up molecular pattern recognition with DNA-based winner-take-all neural networks. Nature. 559 (7714), 370-376 (2018).
- Qian, L., Winfree, E., Bruck, J. Neural network computation with DNA strand displacement cascades. Nature. 475 (7356), 368-372 (2011).
- Chen, Y. -. J., et al. Programmable chemical controllers made from DNA. Nature Nanotechnology. 8 (10), 755-762 (2013).
- di Bernardo, D., Marucci, L., Menolascina, F., Siciliano, V. Predicting synthetic gene networks. Synthetic Gene Networks: Methods and Protocols. 813, 57-81 (2012).
- Xiang, Y., Dalchau, N., Wang, B. Scaling up genetic circuit design for cellular computing: advances and prospects. Natural Computing. 17 (4), 833-853 (2018).
- Gould, N., Hendy, O., Papamichail, D. Computational tools and algorithms for designing customized synthetic genes. Frontiers in Bioengineering and Biotechnology. 2, (2014).
- MacDonald, J. T., Siciliano, V. Computational sequence design with R2oDNA Designer. Mammalian Synthetic Promoters. 1651, 249-262 (2017).
- Cervantes-Salido, V. M., Jaime, O., Brizuela, C. A., Martínez-Pérez, I. M. Improving the design of sequences for DNA computing: A multiobjective evolutionary approach. Applied Soft Computing. 13 (12), 4594-4607 (2013).
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. Journal of Computational Chemistry. 32 (1), 170-173 (2011).
- Fornace, M. E., Porubsky, N. J., Pierce, N. A. A unified dynamic programming framework for the analysis of interacting nucleic acid strands: enhanced models, scalability, and speed. ACS Synthetic Biology. 9 (10), 2665-2678 (2020).
- Wetterstrand, K. DNA sequencing costs: Data. Genome.gov. , (2020).
- Lopez, R., Wang, R., Seelig, G. A molecular multi-gene classifier for disease diagnostics. Nature Chemistry. 10 (7), 746-754 (2018).
- Pardee, K., et al. low-cost detection of Zika virus using programmable biomolecular components. Cell. 165 (5), 1255-1266 (2016).
- Yurke, B., Turberfield, A. J., Mills, A. P., Simmel, F. C., Neumann, J. L. A DNA-fuelled molecular machine made of DNA. Nature. 406 (6796), 605-608 (2000).
- Lin, K. N., Volkel, K., Tuck, J. M., Keung, A. J. Dynamic and scalable DNA-based information storage. Nature Communications. 11 (1), 2981 (2020).
- Yurke, B., Mills, A. P. Using DNA to power nanostructures. Genetic Programming and Evolvable Machines. 4 (2), 111-122 (2003).
- Zhang, D. Y., Turberfield, A. J., Yurke, B., Winfree, E. Engineering entropy-driven reactions and networks catalyzed by DNA. Science. 318 (5853), 1121-1125 (2007).
- Wang, B., Thachuk, C., Ellington, A. D., Winfree, E., Soloveichik, D. Effective design principles for leakless strand displacement systems. Proceedings of the National Academy of Sciences. 115 (52), 12182-12191 (2018).
- Machinek, R. R. F., Ouldridge, T. E., Haley, N. E. C., Bath, J., Turberfield, A. J. Programmable energy landscapes for kinetic control of DNA strand displacement. Nature Communications. 5 (1), 5324 (2014).
- Cabello-Garcia, J., Bae, W., Stan, G. -. B. V., Ouldridge, T. E. Handhold-mediated strand displacement: a nucleic acid-based mechanism for generating far-from-equilibrium assemblies through templated reactions. bioRxiv. , (2020).
- Brophy, J. A. N., Voigt, C. A. Principles of genetic circuit design. Nature Methods. 11 (5), 508-520 (2014).
- Khalil, A. S., et al. A synthetic biology framework for programming eukaryotic transcription functions. Cell. 150 (3), 647-658 (2012).
- Swank, Z., Laohakunakorn, N., Maerkl, S. J. Cell-free gene-regulatory network engineering with synthetic transcription factors. Proceedings of the National Academy of Sciences. 116 (13), 5892-5901 (2019).
- Howland, S. W., Tsuji, T., Gnjatic, S., Ritter, G., Old, L. J., Wittrup, K. D. Inducing efficient cross-priming using antigen-coated yeast particles. Journal of immunotherapy. 31 (7), 607 (2008).
- Abil, Z., Ellefson, J. W., Gollihar, J. D., Watkins, E., Ellington, A. D. Compartmentalized partnered replication for the directed evolution of genetic parts and circuits. Nature Protocols. 12 (12), 2493-2512 (2017).
- Baugh, C., Grate, D., Wilson, C., Doudna, J. A. 2.8 Å crystal structure of the malachite green aptamer11. Journal of Molecular Biology. 301 (1), 117-128 (2000).
- Chou, L. Y. T., Shih, W. M. In vitro transcriptional regulation via nucleic acid-based transcription factors. ACS Synthetic Biology. 8 (11), 2558-2565 (2019).
- Lykke-Andersen, J., Christiansen, J. The C-terminal carboxy group of T7 RNA polymerase ensures efficient magnesium ion-dependent catalysis. Nucleic Acids Research. 26 (24), 5630-5635 (1998).
- Pu, J., Disare, M., Dickinson, B. C. Evolution of C-terminal modification tolerance in full-length and split T7 RNA Polymerase biosensors. Chembiochem. 20 (12), 1547-1553 (2019).
- Gardner, L. P., Mookhtiar, K. A., Coleman, J. E. Initiation, elongation, and processivity of carboxyl-terminal mutants of T7 RNA polymerase. Biochemistry. 36 (10), 2908-2918 (1997).
- Yin, J., Lin, A. J., Golan, D. E., Walsh, C. T. Site-specific protein labeling by Sfp phosphopantetheinyl transferase. Nature Protocols. 1 (1), 280-285 (2006).
- Warden-Rothman, R., Caturegli, I., Popik, V., Tsourkas, A. Sortase-tag expressed protein ligation: combining protein purification and site-specific bioconjugation into a single step. Analytical Chemistry. 85 (22), 11090-11097 (2013).
- Zhang, W. -. B., Sun, F., Tirrell, D. A., Arnold, F. H. Controlling macromolecular topology with genetically encoded SpyTag-SpyCatcher chemistry. Journal of the American Chemical Society. 135 (37), 13988-13997 (2013).

