Dna-Tethered RNA Polymerase para Transcrição In vitro Programável e Computação Molecular
Summary
Descrevemos a engenharia de uma nova polimerase T7 RNA com DNA para regular reações de transcrição in vitro. Discutimos os passos para a síntese e caracterização proteica, validamos a regulação transcricional de prova de conceito e discutimos suas aplicações em computação molecular, diagnóstico e processamento de informações moleculares.
Abstract
A nanotecnologia de DNA permite a automontagem programável de ácidos nucleicos em formas e dinâmicas prescritas pelo usuário para diversas aplicações. Este trabalho demonstra que conceitos da nanotecnologia de DNA podem ser usados para programar a atividade enzimática da polimerase T7 RNA (RNAP) derivada de phage e construir redes de regulação genética sintética escalável. Primeiro, um T7 RNAP com oligonucleotídeo é projetado através da expressão de um RNAP com marca SNAP n-terminal e posterior acoplamento químico da tag SNAP com um oligonucleotídeo modificado por benziloguanina (BG). Em seguida, o deslocamento da cadeia nucleico-ácido é usado para programar transcrição de polimerase sob demanda. Além disso, conjuntos auxiliares de ácido nucleico podem ser usados como “fatores de transcrição artificial” para regular as interações entre o T7 RNAP programado pelo DNA com seus modelos de DNA. Este mecanismo de regulação de transcrição in vitro pode implementar uma variedade de comportamentos de circuito, como lógica digital, feedback, cascata e multiplexing. A composabilidade dessa arquitetura normativa genética facilita a abstração, padronização e escala do design. Esses recursos permitirão a prototipagem rápida de dispositivos genéticos in vitro para aplicações como biosensação, detecção de doenças e armazenamento de dados.
Introduction
A computação em DNA usa um conjunto de oligonucleotídeos projetados como meio de computação. Esses oligonucleotídeos são programados com sequências para serem montados dinamicamente de acordo com a lógica especificada pelo usuário e responder a entradas específicas de ácido nucleico. Em estudos de prova de conceito, a saída da computação normalmente consiste em um conjunto de oligonucleotídeos fluorescentes rotulados que podem ser detectados através de leitores de eletroforese de gel ou fluorescência. Ao longo dos últimos 30 anos, foram demonstrados circuitos computacionais de DNA cada vez mais complexos, como várias cascatas lógicas digitais, redes de reação química e redes neurais1,2,3. Para auxiliar na preparação desses circuitos de DNA, modelos matemáticos têm sido usados para prever a funcionalidade dos circuitos genéticos sintéticos4,5, e ferramentas computacionais foram desenvolvidas para o projeto de sequência de DNA ortogonal6,7,8,9,10 . Em comparação com computadores baseados em silício, as vantagens dos computadores de DNA incluem sua capacidade de interagir diretamente com biomoléculas, operar em solução na ausência de uma fonte de alimentação, bem como sua compactação e estabilidade globais. Com o advento do sequenciamento da próxima geração, o custo de sintetizar computadores de DNA vem diminuindo nas últimas duas décadas a uma taxa mais rápida do que a Lei11de Moore. As aplicações desses computadores baseados em DNA estão começando a surgir, como para o diagnóstico de doenças12,13, para alimentar a biofísica molecular14, e como plataformas de armazenamento de dados15.
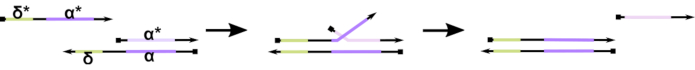
Figura 1: Mecanismo de deslocamento da cadeia de DNA mediado por toehold. O toehold, δ, é uma sequência livre e desvinculada em um duplex parcial. Quando um domínio complementar (δ*) é introduzido em uma segunda vertente, o domínio δ livre serve como um toehold para hibridização, permitindo que o resto da cadeia (ônica*) desloque lentamente seu concorrente através de uma reação reversível zipping/unzipping conhecida como migração de fios. À medida que o comprimento do δ aumenta, o ΔG para a reação para a frente diminui, e o deslocamento acontece mais facilmente. Clique aqui para ver uma versão maior desta figura.
Até o momento, a maioria dos computadores de DNA utilizam um motivo bem estabelecido no campo da nanotecnologia dinâmica de DNA conhecida como deslocamento de fios de DNA mediados por toehold (TMDSD, Figura 1)16. Este motivo consiste em um duplex de DNA parcialmente duplo (dsDNA) exibindo saliências curtas de “toehold” (ou seja, 7 a 10 nucleotídeos (nt)). Os fios de “entrada” de ácido nucleico podem interagir com os duplexes parciais através do dedo do dedo do sol. Isso leva ao deslocamento de um dos fios do duplex parcial, e este fio liberado pode então servir como entrada para duplexes parciais a jusante. Assim, o TMDSD permite a cascata de sinais e o processamento de informações. Em princípio, os motivos ortogonais de TMDSD podem operar de forma independente na solução, permitindo o processamento paralelo de informações. Houve uma série de variações na reação TMDSD, como a troca de fios de DNA mediada por toehold (TMDSE)17, toeholds “sem vazamento” com domínios de dois comprimentos18, dedos incompatíveis de sequência19e deslocamento de fios mediados por “handhold”20. Esses princípios inovadores de design permitem energias tmdsd mais finamente ajustadas e dinâmicas para melhorar o desempenho da computação de DNA.
Circuitos genéticos sintéticos, como circuitos genéticos transcricionais, também são capazes de calcular21,22,23. Esses circuitos são regulados por fatores de transcrição proteica, que ativam ou reprimem transcrição de um gene ligando-se a elementos específicos de DNA regulatório. Comparados com circuitos baseados em DNA, circuitos transcricionais têm várias vantagens. Em primeiro lugar, a transcrição enzimática tem uma taxa de rotatividade muito maior do que os circuitos de DNA catalíticos existentes, gerando assim mais cópias de saída por cópia única de entrada e fornecendo um meio mais eficiente de amplificação de sinal. Além disso, circuitos transcricionais podem produzir diferentes moléculas funcionais, como aptamers ou codificação de RNA (mRNA) mensageiro para proteínas terapêuticas, como saídas de computação, que podem ser exploradas para diferentes aplicações. No entanto, uma grande limitação dos circuitos transcricionais atuais é a falta de escalabilidade. Isso ocorre porque há um conjunto muito limitado de fatores de transcrição ortogonais baseados em proteínas, e o novo desenho de novos fatores de transcrição proteica permanece tecnicamente desafiador e demorado.
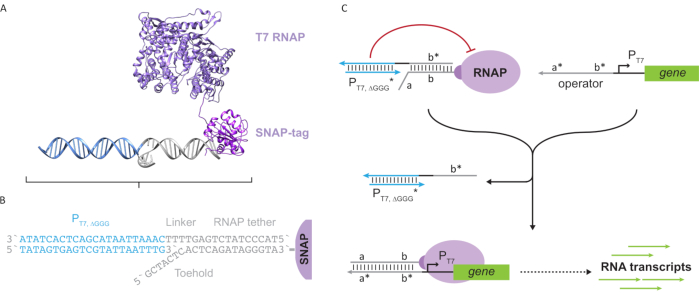
Figura 2: Abstração e mecanismo do complexo de polimerase “tether” e “cage” ( A e B) Uma corda oligonucleotídeo é enzimáticamente rotulada para uma polimerase T7 através da reação snap-tag. Uma gaiola composta por um promotor T7 “falso” com uma saliência complementar de tether permite hibridizar a atividade transcricional de corda e bloco. (C) Quando o operador(a*b*) está presente, ele se liga ao toehold sobre a corda oligonucleotídeo(ab)e desloca a região b* da gaiola, permitindo que a transcrição ocorra. Este número foi modificado de Chou e Shih27. Abreviaturas: RNAP = RNA polymerase. Clique aqui para ver uma versão maior desta figura.
Este artigo introduz um novo bloco de construção para computação molecular que combina as funcionalidades dos circuitos transcricionais com a escalabilidade dos circuitos baseados em DNA. Este bloco de construção é um T7 RNAP covalentemente anexado com uma corda de DNA de um único fio(Figura 2A). Para sintetizar este T7 RNAP ligado ao DNA, a polimerase foi fundida a um SNAP-tag24 n-terminal e recombinantemente expressa em Escherichia coli. A tag SNAP foi então reagida com um oligonucleotídeo funcionalizado com o substrato BG. A corda oligonucleotídeo permite o posicionamento de hóspedes moleculares nas proximidades da polimerase através da hibridização do DNA. Um desses convidados foi um bloqueador transcricional competitivo referido como uma “gaiola”, que consiste em um duplex de DNA “falso” do promotor T7 sem gene a jusante(Figura 2B). Quando vinculada ao RNAP através de sua corda oligonucleotídeo, a gaiola paralisa a atividade de polimerase, superando outros modelos de DNA para a vinculação RNAP, tornando o RNAP em um estado “OFF”(Figura 2C).
Para ativar a polimerase a um estado “ON”, foram projetados modelos de DNA T7 com domínios “operadores” de uma única linha a montante do promotor T7 do gene. O domínio do operador (ou seja, domínio a*b* Figura 2C) pode ser projetado para deslocar a gaiola do RNAP via TMDSD e posicionar o proximal RNAP ao promotor T7 do gene, iniciando assim a transcrição. Alternativamente, foram projetados modelos de DNA onde a sequência do operador era complementar aos fios auxiliares de ácido nucleico que são chamados de “fatores de transcrição artificial” (ou seja, fios TFA e TFB na Figura 3A). Quando ambas as vertentes forem introduzidas na reação, elas se reunirão no site do operador, criando um novo domínio pseudo-contíguo a*b*. Este domínio pode então deslocar a gaiola via TMDSD para iniciar a transcrição(Figura 3B). Estes fios podem ser fornecidos exogenously ou produzidos.
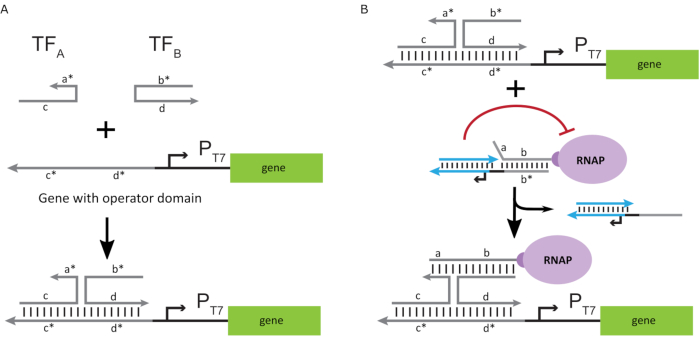
Figura 3: Programação seletiva da atividade de polimerase através de um ativador de interruptor de três componentes. (A) Quando os fatores de transcrição (TFA e TFB) estão presentes, eles se ligam ao domínio do operador a montante do promotor, formando uma sequência pseudo de uma única-strand(a*b*) capaz de deslocar a gaiola através do deslocamento mediado do DNA. (B) Este domínio a*b* pode deslocar a gaiola via TMDSD para iniciar a transcrição. Este número foi modificado de Chou e Shih27. Abreviaturas: TF = fator de transcrição; RNAP = polimerase de RNA; TMDSD = deslocamento de fios de DNA mediados por toehold. Clique aqui para ver uma versão maior desta figura.
O uso de fatores de transcrição nucleicos baseados em ácido para regulação transcricional in vitro permite a implementação escalável de comportamentos sofisticados de circuito, como lógica digital, feedback e cascata de sinais. Por exemplo, pode-se construir cascatas lógicas do portão projetando sequências de ácido nucleico de tal forma que as transcrições de um gene a montante ativam um gene a jusante. Uma aplicação que explora a cascata e multiplexing capazes por essa tecnologia proposta é o desenvolvimento de circuitos de computação molecular mais sofisticados para diagnósticos portáteis e processamento de dados moleculares. Além disso, integrar os recursos de computação molecular e síntese de novo RNA pode permitir novas aplicações. Por exemplo, um circuito molecular pode ser projetado para detectar uma ou uma combinação de RNAs definidas pelo usuário como insumos e saída de RNAs terapêuticos ou mRNAs codificando peptídeos funcionais ou proteínas para aplicações médicas de ponto de cuidado.
Protocol
Representative Results
Discussion
Este estudo demonstra uma abordagem inspirada em nanotecnologia de DNA para controlar a atividade da polimerase T7 RNA, acoplando um T7 RNAP recombinante com marca DE-T7 com um oligonucleotídeo funcionalizado pela BG, que foi posteriormente usado para programar reações TMDSD. Por design, a tag SNAP foi posicionada no N-terminus da polimerase, já que o C-terminus do tipo selvagem T7 RNAP está enterrado dentro do núcleo da estrutura proteica e faz contatos importantes com o modelo de DNA28. Te…
Disclosures
The authors have nothing to disclose.
Acknowledgements
L.Y.T.C reconhece o generoso apoio do New Frontiers in Research Fund-Exploration (NFRF-E), do Natural Sciences and Engineering Research Council of Canada (NSERC) Discovery Grant e da University of Toronto’s Medicine by Design Initiative, que recebe financiamento do Canada First Research Excellence Fund (CFREF).
Materials
| 0.5% polysorbate 20 (TWEEN 20) | BioShop | TWN510.5 | |
| 0.5M ethylenediaminetetraacetic acid (EDTA) | Bio Basic | SD8135 | |
| 10 mM sodium phosphate buffer (pH 7) | Bio Basic | PD0435 | Tablets used to make 10 mM buffer |
| 10% ammonium persulfate (APS) | Sigma Aldrich | A3678-100G | |
| 100 kDa Amicon Ultra-15 Centrifugal Filter Unit | Fisher Scientific | UFC910008 | |
| 100% acetone | Fisher Chemical | A18P4 | |
| 100% ethanol (EtOH) | House Brand | 39752-P016-EAAN | |
| 10x in vitro transcription (IVT) buffer | New England Biolabs | B9012 | |
| 10x Tris-Borate-EDTA (TBE) buffer | Bio Basic | A0026 | |
| 1M Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Sigma Aldrich | I5502-1G | |
| 1M sodium bicarbonate buffer | Sigma Aldrich | S6014-500G | |
| 1M Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 648311-1KG | |
| 1X Tris-EDTA (TE) buffer | ThermoFisher | 12090015 | |
| 2M imidazole | Sigma Aldrich | 56750-100G | |
| 2-mercaptoethanol (BME) | Sigma Aldrich | M3148 | |
| 3M sodium acetate | Bio Basic | SRB1611 | |
| 40% acrylamide (19:1) | Bio Basic | A00062 | |
| 4x LDS protein sample loading buffer | Fisher Scientific | NP0007 | |
| 5M sodium chloride (NaCl) | Bio Basic | DB0483 | |
| 5mM dithiothreitol (DTT) | Sigma Aldrich | 43815-1G | |
| 6x gel loading dye | New England Biolabs | B7024S | |
| agarose B powder | Bio Basic | AB0014 | |
| BG-GLA-NHS | New England Biolabs | S9151S | |
| BL21 competent E. coli | Addgene | C2530H | |
| BLUeye prestained protein ladder | FroggaBio | PM007-0500 | |
| bromophenol blue | Bio Basic | BDB0001 | |
| coomassie blue (SimplyBlue SafeStain) | ThermoFisher | LC6060 | |
| cyanine dye (SYBR Gold nucleic acid gel stain) | Fisher Scientific | S11494 | |
| cyanine dye (SYBR Safe nucleic acid gel stain) | Fisher Scientific | S33102 | |
| dry dimethyl sulfoxide (DMSO) | Fisher Scientific | D12345 | |
| formamide | Sigma Aldrich | F9037-100ML | |
| glycerol | Bio Basic | GB0232 | |
| kanamycin sulfate | BioShop | KAN201.5 | |
| lysogeny broth | Sigma Aldrich | L2542-500ML | |
| malachite green oxalate | Sigma Aldrich | 2437-29-8 | |
| N,N,N'N'-Tetramethylethane-1,2-diamine (TEMED) | Sigma Aldrich | T9281-25ML | |
| NuPAGE MES SDS running buffer (20x) | Fisher Scientific | LSNP0002 | |
| NuPAGE Novex 4-12% Bis-Tris gel 1.0 mm 12-well | Life Technologies | NP0322BOX | |
| oligonucleotide (cage antisense) | IDT | N/A | TATAGTGAGTCGTATTAATTTG |
| oligonucleotide (cage sense) | IDT | N/A | TCAGTCACCTATCTGTTTCAAA TTAATACGACTCACTATA |
| oligonucleotide (malachite green aptamer antisense) | IDT | N/A | GGATCCATTCGTTACCTGGCT CTCGCCAGTCGGGATCCTATA GTGAGTCGTATTACAGTTCCAT TATCGCCGTAGTTGGTGTACT |
| oligonucleotide (malachite green aptamer sense) | IDT | N/A | TAATACGACTCACTATAGGATC CCGACTGGCGAGAGCCAGGT AACGAATGGATCC |
| oligonucleotide (Transcription Factor A) | IDT | N/A | AGTACACCAACTACGAGTGAG |
| oligonucleotide (Transcription Factor B) | IDT | N/A | TCAGTCACCTATCTGGCGATAA TGGAACTG |
| oligonucleotide with 3’ Amine modification (tether) | IDT | N/A | GCTACTCACTCAGATAGGTGAC TGA/3AmMO/ |
| Pierce strong ion exchange spin columns | Fisher Scientific | 90008 | |
| plasmid encoding SNAP T7 RNAP and kanamycin resistance genes | Genscript | N/A | custom gene insert |
| protein purification column (HisPur Ni-NTA spin column) | Fisher Scientific | 88226 | |
| rNTP mix | New England Biolabs | N0466S | |
| Roche mini quick DNA spin column | Sigma Aldrich | 11814419001 | |
| Triton X-100 | Sigma Aldrich | T8787-100ML | |
| Ultra Low Range DNA ladder | Fisher Scientific | 10597012 | |
| urea | BioShop | URE001.1 |
References
- Cherry, K. M., Qian, L. Scaling up molecular pattern recognition with DNA-based winner-take-all neural networks. Nature. 559 (7714), 370-376 (2018).
- Qian, L., Winfree, E., Bruck, J. Neural network computation with DNA strand displacement cascades. Nature. 475 (7356), 368-372 (2011).
- Chen, Y. -. J., et al. Programmable chemical controllers made from DNA. Nature Nanotechnology. 8 (10), 755-762 (2013).
- di Bernardo, D., Marucci, L., Menolascina, F., Siciliano, V. Predicting synthetic gene networks. Synthetic Gene Networks: Methods and Protocols. 813, 57-81 (2012).
- Xiang, Y., Dalchau, N., Wang, B. Scaling up genetic circuit design for cellular computing: advances and prospects. Natural Computing. 17 (4), 833-853 (2018).
- Gould, N., Hendy, O., Papamichail, D. Computational tools and algorithms for designing customized synthetic genes. Frontiers in Bioengineering and Biotechnology. 2, (2014).
- MacDonald, J. T., Siciliano, V. Computational sequence design with R2oDNA Designer. Mammalian Synthetic Promoters. 1651, 249-262 (2017).
- Cervantes-Salido, V. M., Jaime, O., Brizuela, C. A., Martínez-Pérez, I. M. Improving the design of sequences for DNA computing: A multiobjective evolutionary approach. Applied Soft Computing. 13 (12), 4594-4607 (2013).
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. Journal of Computational Chemistry. 32 (1), 170-173 (2011).
- Fornace, M. E., Porubsky, N. J., Pierce, N. A. A unified dynamic programming framework for the analysis of interacting nucleic acid strands: enhanced models, scalability, and speed. ACS Synthetic Biology. 9 (10), 2665-2678 (2020).
- Wetterstrand, K. DNA sequencing costs: Data. Genome.gov. , (2020).
- Lopez, R., Wang, R., Seelig, G. A molecular multi-gene classifier for disease diagnostics. Nature Chemistry. 10 (7), 746-754 (2018).
- Pardee, K., et al. low-cost detection of Zika virus using programmable biomolecular components. Cell. 165 (5), 1255-1266 (2016).
- Yurke, B., Turberfield, A. J., Mills, A. P., Simmel, F. C., Neumann, J. L. A DNA-fuelled molecular machine made of DNA. Nature. 406 (6796), 605-608 (2000).
- Lin, K. N., Volkel, K., Tuck, J. M., Keung, A. J. Dynamic and scalable DNA-based information storage. Nature Communications. 11 (1), 2981 (2020).
- Yurke, B., Mills, A. P. Using DNA to power nanostructures. Genetic Programming and Evolvable Machines. 4 (2), 111-122 (2003).
- Zhang, D. Y., Turberfield, A. J., Yurke, B., Winfree, E. Engineering entropy-driven reactions and networks catalyzed by DNA. Science. 318 (5853), 1121-1125 (2007).
- Wang, B., Thachuk, C., Ellington, A. D., Winfree, E., Soloveichik, D. Effective design principles for leakless strand displacement systems. Proceedings of the National Academy of Sciences. 115 (52), 12182-12191 (2018).
- Machinek, R. R. F., Ouldridge, T. E., Haley, N. E. C., Bath, J., Turberfield, A. J. Programmable energy landscapes for kinetic control of DNA strand displacement. Nature Communications. 5 (1), 5324 (2014).
- Cabello-Garcia, J., Bae, W., Stan, G. -. B. V., Ouldridge, T. E. Handhold-mediated strand displacement: a nucleic acid-based mechanism for generating far-from-equilibrium assemblies through templated reactions. bioRxiv. , (2020).
- Brophy, J. A. N., Voigt, C. A. Principles of genetic circuit design. Nature Methods. 11 (5), 508-520 (2014).
- Khalil, A. S., et al. A synthetic biology framework for programming eukaryotic transcription functions. Cell. 150 (3), 647-658 (2012).
- Swank, Z., Laohakunakorn, N., Maerkl, S. J. Cell-free gene-regulatory network engineering with synthetic transcription factors. Proceedings of the National Academy of Sciences. 116 (13), 5892-5901 (2019).
- Howland, S. W., Tsuji, T., Gnjatic, S., Ritter, G., Old, L. J., Wittrup, K. D. Inducing efficient cross-priming using antigen-coated yeast particles. Journal of immunotherapy. 31 (7), 607 (2008).
- Abil, Z., Ellefson, J. W., Gollihar, J. D., Watkins, E., Ellington, A. D. Compartmentalized partnered replication for the directed evolution of genetic parts and circuits. Nature Protocols. 12 (12), 2493-2512 (2017).
- Baugh, C., Grate, D., Wilson, C., Doudna, J. A. 2.8 Å crystal structure of the malachite green aptamer11. Journal of Molecular Biology. 301 (1), 117-128 (2000).
- Chou, L. Y. T., Shih, W. M. In vitro transcriptional regulation via nucleic acid-based transcription factors. ACS Synthetic Biology. 8 (11), 2558-2565 (2019).
- Lykke-Andersen, J., Christiansen, J. The C-terminal carboxy group of T7 RNA polymerase ensures efficient magnesium ion-dependent catalysis. Nucleic Acids Research. 26 (24), 5630-5635 (1998).
- Pu, J., Disare, M., Dickinson, B. C. Evolution of C-terminal modification tolerance in full-length and split T7 RNA Polymerase biosensors. Chembiochem. 20 (12), 1547-1553 (2019).
- Gardner, L. P., Mookhtiar, K. A., Coleman, J. E. Initiation, elongation, and processivity of carboxyl-terminal mutants of T7 RNA polymerase. Biochemistry. 36 (10), 2908-2918 (1997).
- Yin, J., Lin, A. J., Golan, D. E., Walsh, C. T. Site-specific protein labeling by Sfp phosphopantetheinyl transferase. Nature Protocols. 1 (1), 280-285 (2006).
- Warden-Rothman, R., Caturegli, I., Popik, V., Tsourkas, A. Sortase-tag expressed protein ligation: combining protein purification and site-specific bioconjugation into a single step. Analytical Chemistry. 85 (22), 11090-11097 (2013).
- Zhang, W. -. B., Sun, F., Tirrell, D. A., Arnold, F. H. Controlling macromolecular topology with genetically encoded SpyTag-SpyCatcher chemistry. Journal of the American Chemical Society. 135 (37), 13988-13997 (2013).

