RNA פולימראז קשור DNA לתכנות תמלול במבחנה וחישוב מולקולרי
Summary
אנו מתארים את ההנדסה של פולימראז T7 RNA חדשני הקשור לדנ”א כדי לווסת את תגובות התמלול במבחנה. אנו דנים בצעדים לסינתזה ואפיון חלבונים, מאמתים רגולציה של תמלול הוכחת הרעיון, ודנים ביישומיה במחשוב מולקולרי, אבחון ועיבוד מידע מולקולרי.
Abstract
ננוטכנולוגיה של DNA מאפשרת הרכבה עצמית ניתנת לתכנות של חומצות גרעין לצורות ודינמיקה שנקבעו על ידי המשתמש עבור יישומים מגוונים. עבודה זו מדגימה כי מושגים מננוטכנולוגיה DNA ניתן להשתמש כדי לתכנת את הפעילות אנזימטית של פולימראז T7 RNA נגזר phage (RNAP) ולבנות רשתות רגולטוריות גנים סינתטיים מדרגיים. ראשית, T7 RNAP הקשור לאוליגונוקלאוטיד מתוכנן באמצעות ביטוי של RNAP מתויג N-סופני SNAP וצימוד כימי לאחר מכן של תג SNAP עם אוליגונוקלאוטיד בנצילגואנין (BG). לאחר מכן, עקירת גדיל חומצת גרעין משמשת לתכנות שעתוק פולימראז לפי דרישה. בנוסף, מכלולי חומצת גרעין עזר יכולים לשמש “גורמי שעתוק מלאכותיים” כדי לווסת את האינטראקציות בין RNAP T7 מתוכנת DNA עם תבניות ה- DNA שלה. מנגנון רגולציה זה של תמלול במבחנה יכול ליישם מגוון התנהגויות מעגלים כגון לוגיקה דיגיטלית, משוב, מדורג ומולטיפלקסינג. המורכבות של ארכיטקטורה רגולטורית זו של הגנים מאפשרת הפשטה, סטנדרטיזציה ושינוי קנה מידה של עיצוב. תכונות אלה יאפשרו יצירת סוג של מכשירים גנטיים במבחנה עבור יישומים כגון חישה ביולוגית, זיהוי מחלות ואחסון נתונים.
Introduction
מחשוב דנ”א משתמש בערכה של אוליגונוקלאוטידים מעוצבים כמדיום לחישוב. אוליגונוקלאוטידים אלה מתוכנתים עם רצפים להרכבה דינמית על פי לוגיקה שצוינה על-ידי המשתמש ולהגיב לכניסות ספציפיות של חומצת גרעין. במחקרי הוכחת הרעיון, הפלט של החישוב מורכב בדרך כלל מסט של אוליגונוקלאוטידים שכותרתם פלואורסצנטית שניתן לזהות באמצעות אלקטרופורזה ג’ל או קוראי לוחות פלואורסצנטיות. במהלך 30 השנים האחרונות, מעגלים חישוביים DNA מורכבים יותר ויותר הודגמו, כגון מפלי לוגיקה דיגיטלית שונים, רשתותתגובהכימית, ורשתות עצביות 1,2,3. כדי לסייע בהכנת מעגלי DNA אלה, מודלים מתמטיים שימשו לחיזוי הפונקציונליות של מעגלי גנים סינתטיים4,5, וכלים חישוביים פותחו עבור עיצוב רצף DNA אורתוגונל6,7,8,9,10 . בהשוואה למחשבים מבוססי סיליקון, היתרונות של מחשבי DNA כוללים את יכולתם להתממשק ישירות עם ביומולקולים, לפעול בפתרון בהיעדר ספק כוח, כמו גם את הקומפקטיות והיציבות הכוללות שלהם. עם הופעתו של רצף הדור הבא, העלות של סינתזה של מחשבי DNA כבר יורדת בשני העשורים האחרונים בקצב מהר יותר מאשר חוק מור11. יישומים של מחשבים מבוססי DNA כאלה מתחילים כעת לצוץ, כגון לאבחון מחלות12,13, להפעלת ביופיסיקה מולקולרית14, וכפלטפורמות אחסון נתונים15.
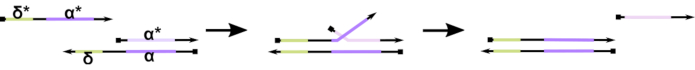
איור 1:מנגנון של תזוזת גדיל DNA בתיווך דריסת רגל. דריסת הבוהן, δ, היא רצף חופשי ולא מאוגד על דופלקס חלקי. כאשר תחום משלים (δ*) מוצג על גדיל שני, הדומיין δ החופשי משמש דריסת רגל להכלאה, ומאפשר לשאר הגדיל (ɑ*) לעקור לאט את המתחרה שלו באמצעות תגובה הפיכה רוכסת / בלתי מתפשרת המכונה העברת גדילים. ככל שאורך δ גדל, ה- ΔG לתגובה הקדמית פוחת, והעקירה מתרחשת ביתר קלות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
עד כה, רוב מחשבי הדנ”א משתמשים במוטיב מבוסס היטב בתחום הננוטכנולוגיה הדינמית של הדנ”א המכונה עקירת גדיל DNA בתיווך בוהן (TMDSD, איור 1)16. מוטיב זה מורכב דופלקס DNA כפול תקוע חלקית (dsDNA) המציגים overhangs קצרים “דריסת רגל” (כלומר, 7- עד 10 נוקלאוטידים (nt)). גדילי “קלט” חומצת גרעין יכולים לקיים אינטראקציה עם הדופלקסים החלקיים דרך דריסת המכהן. זה מוביל לעקירתו של אחד הגדילים מהדופלקס החלקי, והגדיל המשוחרר הזה יכול לשמש כקלט לדופלקסים חלקיים במורד הזרם. לכן, TMDSD מאפשרת סילוק את אותות ועיבוד מידע. באופן עקרוני, מוטיבים אורתוגונליים של TMDSD יכולים לפעול באופן עצמאי בפתרון, ומאפשרים עיבוד מידע מקביל. היו מספר וריאציות על תגובת TMDSD, כגון החלפת גדיל DNA בתיווך דריסת רגל (TMDSE)17, דריסות “ללא דליפות” עם תחומים ארוכים כפולים18, דריסות רצף לא תואמות19, ו “אחיזת יד”, עקירת גדילים בתיווך20. עקרונות עיצוב חדשניים אלה מאפשרים אנרגטיות ודינמיקה של TMDSD מכוונות יותר לשיפור ביצועי מחשוב ה- DNA.
מעגלי גנים סינתטיים, כגון מעגלי גנים תמלול, מסוגלים גם לחשב21,22,23. מעגלים אלה מוסדרים על ידי גורמי שעתוק חלבונים, המפעילים או מדחיקים שעתוק של גן על ידי קשירה לאלמנטים ספציפיים של DNA רגולטורי. בהשוואה למעגלים מבוססי דנ”א, למעגלי התמלול יש מספר יתרונות. ראשית, לתעתיק אנזימטי יש שיעור תחלופה גבוה בהרבה ממעגלי ה- DNA הקטליטיים הקיימים, ובכך יוצר יותר עותקים של פלט לכל עותק יחיד של קלט ומספק אמצעי יעיל יותר להגברת אותות. בנוסף, מעגלי שעתוק יכולים לייצר מולקולות פונקציונליות שונות, כגון aptamers או קידוד RNA שליח (mRNA) עבור חלבונים טיפוליים, כמו יציאות חישוב, אשר ניתן לנצל עבור יישומים שונים. עם זאת, מגבלה מרכזית של מעגלי התמלול הנוכחיים היא חוסר המדרגיות שלהם. הסיבה לכך היא שיש קבוצה מוגבלת מאוד של גורמי שעתוק מבוססי חלבון אורתוגונל, ועיצוב דה נובו של גורמי שעתוק חלבונים חדשים נשאר מאתגר מבחינה טכנית וגוזל זמן רב.
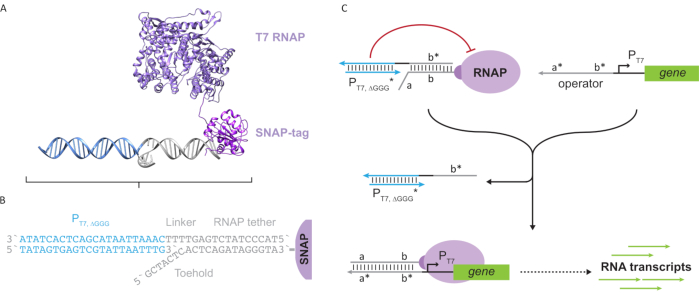
איור 2: הפשטה ומנגנון של קומפלקס פולימראז “לקשור” ו”כלוב”. (A ו- B) קשירה אוליגונוקלאוטיד מסומנת באופן אנזימטי לפולימראז T7 באמצעות תגובת SNAP-tag. כלוב המורכב ממקדם T7 “מזויף” עם תוחם משלים קשירה מאפשר לו להתיידד לקשירה ולחסום פעילות שעתוק. (C)כאשר המפעיל (a*b*) קיים, הוא נקשר לאיכות הבוהן על הרצועה אוליגונוקלאוטיד (ab) ומזיז את אזור b * של הכלוב, ומאפשר שעתוק להתרחש. נתון זה שונה מצ’ו ושי27. קיצורים: RNAP = RNA פולימראז. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
מאמר זה מציג אבן בניין חדשנית למחשוב מולקולרי המשלבת את הפונקציות של מעגלי שעתוק עם מדרגיות של מעגלים מבוססי DNA. אבן בניין זו היא T7 RNAP המחוברת באופן קוולנטי עם רצועת DNA חד-גדילית(איור 2A). כדי לסנתז את ה-T7 RNAP הזה, הפולימראז הותך לתגSNAP-24 של N-terminal והתבטא מחדש באשריצ’יה קולי. תג SNAP הגיב אז עם אוליגונוקלאוטיד פונקציונלי עם מצע BG. הרצועה אוליגונוקלאוטיד מאפשרת מיקום של אורחים מולקולריים בסמיכות לפולימראז באמצעות הכלאת DNA. אורח אחד כזה היה חוסם תמלול תחרותי המכונה “כלוב”, המורכב ודופלקס DNA “מזויף” של מקדם T7 ללא גנים במורד הזרם(איור 2B). כאשר הוא מאוגד לרנ”פ באמצעות קשירת האוליגונוקלאוטיד שלו, הכלוב מעכב פעילות פולימראז על ידי אי-התאמה של תבניות DNA אחרות עבור כריכת RNAP, ועיבוד ה-RNAP במצב “כבוי”(איור 2C).
כדי להפעיל את הפולימראז למצב “ON”, תוכננו תבניות DNA T7 עם תחומי “אופרטור” חד-גדיליים במעלה הזרם של מקדם T7 של הגן. תחום המפעיל (כלומר, תחום a*b* איור 2C) יכול להיות מתוכנן לעקור את הכלוב מה- RNAP באמצעות TMDSD ולמקם את ה- RNAP הקרוב למקדם T7 של הגן, ובכך ליזום שעתוק. לחלופין, תבניות DNA תוכננו גם כאשר רצף המפעיל היה משלים לגדילי חומצת גרעין עזר המכונים “גורמי שעתוק מלאכותיים” (כלומר, גדילי TFA ו- TFB באיור 3A). כאשר שני גדילים מוצגים לתוך התגובה, הם יתאספו באתר המפעיל, יצירת תחום פסאודו-רציף חדש a *b*. תחום זה יכול לאחר מכן לעקור את הכלוב באמצעות TMDSD כדי ליזום תמלול (איור 3B). גדילים אלה יכולים להיות מסופקים או אקסוגני או מיוצר.
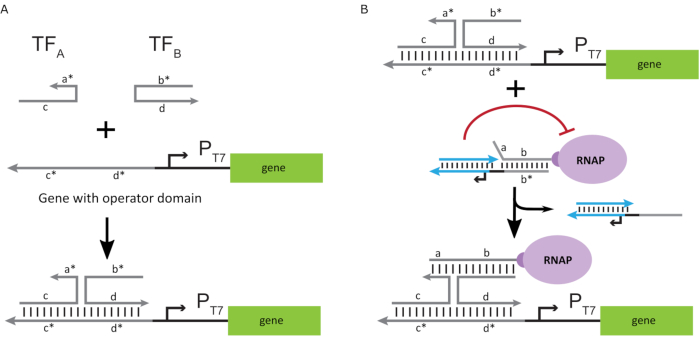
איור 3: תכנות סלקטיבי של פעילות פולימראז באמצעות מפעיל מתג בעל שלושה רכיבים. (A)כאשר קיימים גורמי התמלול (TFA ו- TFB), הם נקשרים לתחום המפעיל במעלה הזרם של המקדם, ויוצרים רצף פסאודו חד-גדילי (a*b*) המסוגל לעקור את הכלוב באמצעות תזוזת דנ”א מתווכת. (B)תחום a*b* זה יכול לעקור את הכלוב באמצעות TMDSD כדי ליזום תמלול. נתון זה שונה מצ’ו ושי27. קיצורים: TF = גורם שעתוק; RNAP = RNA פולימראז; TMDSD = תזוזת גדיל DNA בתיווך דריסת רגל. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
השימוש בגורמי שעתוק מבוססי חומצת גרעין עבור ויסות תמלול במבחנה מאפשר יישום מדרגי של התנהגויות מעגל מתוחכמות כגון לוגיקה דיגיטלית, משוב, ומדדי אותות. לדוגמה, ניתן לבנות מפלי שער לוגיים על ידי תכנון רצפי חומצות גרעין כך שהתעתיקים מגן במעלה הזרם מפעילים גן במורד הזרם. יישום אחד המנצל את המדורגים והמכפילים המסוגלים בטכנולוגיה מוצעת זו הוא פיתוח מעגלי מחשוב מולקולריים מתוחכמים יותר לאבחון נייד ועיבוד נתונים מולקולריים. בנוסף, שילוב יכולות המחשוב המולקולרי וסינתזת דה נובו RNA יכול לאפשר יישומים חדשים. לדוגמה, ניתן לתכנן מעגל מולקולרי כדי לזהות אחד או שילוב של RNAs המוגדרים על-ידי המשתמש כתשומות ופלט של RNAs טיפוליים או mRNAs המקודדים פפטידים פונקציונליים או חלבונים עבור יישומים רפואיים נקודתיים.
Protocol
Representative Results
Discussion
מחקר זה מדגים גישה בהשראת ננוטכנולוגיה DNA כדי לשלוט על הפעילות של T7 RNA פולימראז על ידי צימוד covalently SNAP רקומביננט T7 RNAP עם אוליגונוקלאוטיד פונקציונלי BG, אשר שימש לאחר מכן לתכנת תגובות TMDSD. על ידי עיצוב, SNAP-תג היה ממוקם ב N-terminus של פולימראז, כמו C-terminus של סוג בר T7 RNAP קבור בתוך הליבה מבנה החלבון ועושה…
Disclosures
The authors have nothing to disclose.
Acknowledgements
L.Y.T.C מכיר בתמיכה נדיבה מהחזיתות החדשות בקרן המחקר-חקר (NFRF-E), מענק דיסקברי של המועצה למדעי הטבע והנדסה של קנדה (NSERC) ואוניברסיטת טורונטו לרפואה על ידי יוזמת עיצוב, המקבלת מימון מהקרן למצוינות במחקר הראשון בקנדה (CFREF).
Materials
| 0.5% polysorbate 20 (TWEEN 20) | BioShop | TWN510.5 | |
| 0.5M ethylenediaminetetraacetic acid (EDTA) | Bio Basic | SD8135 | |
| 10 mM sodium phosphate buffer (pH 7) | Bio Basic | PD0435 | Tablets used to make 10 mM buffer |
| 10% ammonium persulfate (APS) | Sigma Aldrich | A3678-100G | |
| 100 kDa Amicon Ultra-15 Centrifugal Filter Unit | Fisher Scientific | UFC910008 | |
| 100% acetone | Fisher Chemical | A18P4 | |
| 100% ethanol (EtOH) | House Brand | 39752-P016-EAAN | |
| 10x in vitro transcription (IVT) buffer | New England Biolabs | B9012 | |
| 10x Tris-Borate-EDTA (TBE) buffer | Bio Basic | A0026 | |
| 1M Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Sigma Aldrich | I5502-1G | |
| 1M sodium bicarbonate buffer | Sigma Aldrich | S6014-500G | |
| 1M Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 648311-1KG | |
| 1X Tris-EDTA (TE) buffer | ThermoFisher | 12090015 | |
| 2M imidazole | Sigma Aldrich | 56750-100G | |
| 2-mercaptoethanol (BME) | Sigma Aldrich | M3148 | |
| 3M sodium acetate | Bio Basic | SRB1611 | |
| 40% acrylamide (19:1) | Bio Basic | A00062 | |
| 4x LDS protein sample loading buffer | Fisher Scientific | NP0007 | |
| 5M sodium chloride (NaCl) | Bio Basic | DB0483 | |
| 5mM dithiothreitol (DTT) | Sigma Aldrich | 43815-1G | |
| 6x gel loading dye | New England Biolabs | B7024S | |
| agarose B powder | Bio Basic | AB0014 | |
| BG-GLA-NHS | New England Biolabs | S9151S | |
| BL21 competent E. coli | Addgene | C2530H | |
| BLUeye prestained protein ladder | FroggaBio | PM007-0500 | |
| bromophenol blue | Bio Basic | BDB0001 | |
| coomassie blue (SimplyBlue SafeStain) | ThermoFisher | LC6060 | |
| cyanine dye (SYBR Gold nucleic acid gel stain) | Fisher Scientific | S11494 | |
| cyanine dye (SYBR Safe nucleic acid gel stain) | Fisher Scientific | S33102 | |
| dry dimethyl sulfoxide (DMSO) | Fisher Scientific | D12345 | |
| formamide | Sigma Aldrich | F9037-100ML | |
| glycerol | Bio Basic | GB0232 | |
| kanamycin sulfate | BioShop | KAN201.5 | |
| lysogeny broth | Sigma Aldrich | L2542-500ML | |
| malachite green oxalate | Sigma Aldrich | 2437-29-8 | |
| N,N,N'N'-Tetramethylethane-1,2-diamine (TEMED) | Sigma Aldrich | T9281-25ML | |
| NuPAGE MES SDS running buffer (20x) | Fisher Scientific | LSNP0002 | |
| NuPAGE Novex 4-12% Bis-Tris gel 1.0 mm 12-well | Life Technologies | NP0322BOX | |
| oligonucleotide (cage antisense) | IDT | N/A | TATAGTGAGTCGTATTAATTTG |
| oligonucleotide (cage sense) | IDT | N/A | TCAGTCACCTATCTGTTTCAAA TTAATACGACTCACTATA |
| oligonucleotide (malachite green aptamer antisense) | IDT | N/A | GGATCCATTCGTTACCTGGCT CTCGCCAGTCGGGATCCTATA GTGAGTCGTATTACAGTTCCAT TATCGCCGTAGTTGGTGTACT |
| oligonucleotide (malachite green aptamer sense) | IDT | N/A | TAATACGACTCACTATAGGATC CCGACTGGCGAGAGCCAGGT AACGAATGGATCC |
| oligonucleotide (Transcription Factor A) | IDT | N/A | AGTACACCAACTACGAGTGAG |
| oligonucleotide (Transcription Factor B) | IDT | N/A | TCAGTCACCTATCTGGCGATAA TGGAACTG |
| oligonucleotide with 3’ Amine modification (tether) | IDT | N/A | GCTACTCACTCAGATAGGTGAC TGA/3AmMO/ |
| Pierce strong ion exchange spin columns | Fisher Scientific | 90008 | |
| plasmid encoding SNAP T7 RNAP and kanamycin resistance genes | Genscript | N/A | custom gene insert |
| protein purification column (HisPur Ni-NTA spin column) | Fisher Scientific | 88226 | |
| rNTP mix | New England Biolabs | N0466S | |
| Roche mini quick DNA spin column | Sigma Aldrich | 11814419001 | |
| Triton X-100 | Sigma Aldrich | T8787-100ML | |
| Ultra Low Range DNA ladder | Fisher Scientific | 10597012 | |
| urea | BioShop | URE001.1 |
References
- Cherry, K. M., Qian, L. Scaling up molecular pattern recognition with DNA-based winner-take-all neural networks. Nature. 559 (7714), 370-376 (2018).
- Qian, L., Winfree, E., Bruck, J. Neural network computation with DNA strand displacement cascades. Nature. 475 (7356), 368-372 (2011).
- Chen, Y. -. J., et al. Programmable chemical controllers made from DNA. Nature Nanotechnology. 8 (10), 755-762 (2013).
- di Bernardo, D., Marucci, L., Menolascina, F., Siciliano, V. Predicting synthetic gene networks. Synthetic Gene Networks: Methods and Protocols. 813, 57-81 (2012).
- Xiang, Y., Dalchau, N., Wang, B. Scaling up genetic circuit design for cellular computing: advances and prospects. Natural Computing. 17 (4), 833-853 (2018).
- Gould, N., Hendy, O., Papamichail, D. Computational tools and algorithms for designing customized synthetic genes. Frontiers in Bioengineering and Biotechnology. 2, (2014).
- MacDonald, J. T., Siciliano, V. Computational sequence design with R2oDNA Designer. Mammalian Synthetic Promoters. 1651, 249-262 (2017).
- Cervantes-Salido, V. M., Jaime, O., Brizuela, C. A., Martínez-Pérez, I. M. Improving the design of sequences for DNA computing: A multiobjective evolutionary approach. Applied Soft Computing. 13 (12), 4594-4607 (2013).
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. Journal of Computational Chemistry. 32 (1), 170-173 (2011).
- Fornace, M. E., Porubsky, N. J., Pierce, N. A. A unified dynamic programming framework for the analysis of interacting nucleic acid strands: enhanced models, scalability, and speed. ACS Synthetic Biology. 9 (10), 2665-2678 (2020).
- Wetterstrand, K. DNA sequencing costs: Data. Genome.gov. , (2020).
- Lopez, R., Wang, R., Seelig, G. A molecular multi-gene classifier for disease diagnostics. Nature Chemistry. 10 (7), 746-754 (2018).
- Pardee, K., et al. low-cost detection of Zika virus using programmable biomolecular components. Cell. 165 (5), 1255-1266 (2016).
- Yurke, B., Turberfield, A. J., Mills, A. P., Simmel, F. C., Neumann, J. L. A DNA-fuelled molecular machine made of DNA. Nature. 406 (6796), 605-608 (2000).
- Lin, K. N., Volkel, K., Tuck, J. M., Keung, A. J. Dynamic and scalable DNA-based information storage. Nature Communications. 11 (1), 2981 (2020).
- Yurke, B., Mills, A. P. Using DNA to power nanostructures. Genetic Programming and Evolvable Machines. 4 (2), 111-122 (2003).
- Zhang, D. Y., Turberfield, A. J., Yurke, B., Winfree, E. Engineering entropy-driven reactions and networks catalyzed by DNA. Science. 318 (5853), 1121-1125 (2007).
- Wang, B., Thachuk, C., Ellington, A. D., Winfree, E., Soloveichik, D. Effective design principles for leakless strand displacement systems. Proceedings of the National Academy of Sciences. 115 (52), 12182-12191 (2018).
- Machinek, R. R. F., Ouldridge, T. E., Haley, N. E. C., Bath, J., Turberfield, A. J. Programmable energy landscapes for kinetic control of DNA strand displacement. Nature Communications. 5 (1), 5324 (2014).
- Cabello-Garcia, J., Bae, W., Stan, G. -. B. V., Ouldridge, T. E. Handhold-mediated strand displacement: a nucleic acid-based mechanism for generating far-from-equilibrium assemblies through templated reactions. bioRxiv. , (2020).
- Brophy, J. A. N., Voigt, C. A. Principles of genetic circuit design. Nature Methods. 11 (5), 508-520 (2014).
- Khalil, A. S., et al. A synthetic biology framework for programming eukaryotic transcription functions. Cell. 150 (3), 647-658 (2012).
- Swank, Z., Laohakunakorn, N., Maerkl, S. J. Cell-free gene-regulatory network engineering with synthetic transcription factors. Proceedings of the National Academy of Sciences. 116 (13), 5892-5901 (2019).
- Howland, S. W., Tsuji, T., Gnjatic, S., Ritter, G., Old, L. J., Wittrup, K. D. Inducing efficient cross-priming using antigen-coated yeast particles. Journal of immunotherapy. 31 (7), 607 (2008).
- Abil, Z., Ellefson, J. W., Gollihar, J. D., Watkins, E., Ellington, A. D. Compartmentalized partnered replication for the directed evolution of genetic parts and circuits. Nature Protocols. 12 (12), 2493-2512 (2017).
- Baugh, C., Grate, D., Wilson, C., Doudna, J. A. 2.8 Å crystal structure of the malachite green aptamer11. Journal of Molecular Biology. 301 (1), 117-128 (2000).
- Chou, L. Y. T., Shih, W. M. In vitro transcriptional regulation via nucleic acid-based transcription factors. ACS Synthetic Biology. 8 (11), 2558-2565 (2019).
- Lykke-Andersen, J., Christiansen, J. The C-terminal carboxy group of T7 RNA polymerase ensures efficient magnesium ion-dependent catalysis. Nucleic Acids Research. 26 (24), 5630-5635 (1998).
- Pu, J., Disare, M., Dickinson, B. C. Evolution of C-terminal modification tolerance in full-length and split T7 RNA Polymerase biosensors. Chembiochem. 20 (12), 1547-1553 (2019).
- Gardner, L. P., Mookhtiar, K. A., Coleman, J. E. Initiation, elongation, and processivity of carboxyl-terminal mutants of T7 RNA polymerase. Biochemistry. 36 (10), 2908-2918 (1997).
- Yin, J., Lin, A. J., Golan, D. E., Walsh, C. T. Site-specific protein labeling by Sfp phosphopantetheinyl transferase. Nature Protocols. 1 (1), 280-285 (2006).
- Warden-Rothman, R., Caturegli, I., Popik, V., Tsourkas, A. Sortase-tag expressed protein ligation: combining protein purification and site-specific bioconjugation into a single step. Analytical Chemistry. 85 (22), 11090-11097 (2013).
- Zhang, W. -. B., Sun, F., Tirrell, D. A., Arnold, F. H. Controlling macromolecular topology with genetically encoded SpyTag-SpyCatcher chemistry. Journal of the American Chemical Society. 135 (37), 13988-13997 (2013).

