RIBO-seq في البكتيريا: مجموعة عينة وبروتوكول إعداد المكتبة لتسلسل NGS
Summary
هنا نحن نصف مراحل جمع العينات وإعداد لريبو-seq في البكتيريا. ويؤدي تسلسل المكتبات المعدة وفقا لهذه المبادئ التوجيهية إلى بيانات كافية لإجراء تحليل معلوماتي حيوي شامل. البروتوكول الذي نقدمه بسيط، ويستخدم معدات مختبرية قياسية ويستغرق سبعة أيام من التحلل إلى الحصول على المكتبات.
Abstract
تقنية التنميط الريبوسوم (RIBO-seq) هي حاليا الأداة الأكثر فعالية لدراسة عملية تخليق البروتين في الجسم الحي. ميزة هذه الطريقة، بالمقارنة مع النهج الأخرى، هي قدرتها على رصد الترجمة عن طريق رسم خرائط دقيقة لموقف وعدد من الريبوسومات على نسخة مرنا.
في هذه المقالة، نقوم بوصف المراحل المتتالية لجمع العينات وإعداد طريقة ريبو-سيك في البكتيريا، مع تسليط الضوء على التفاصيل ذات الصلة بتخطيط وتنفيذ التجربة.
وبما أن ريبو-سيك يعتمد على الريبوسومات السليمة والرنانات المرتبطة بها، فإن الخطوة الرئيسية هي التثبيط السريع للترجمة والتفكك الكافي للخلايا. وبالتالي، نقترح الترشيح وتجميد فلاش في النيتروجين السائل لحصاد الخلايا مع المعالجة المسبقة اختياري مع الكلورامفينيكول لوقف الترجمة في البكتيريا. للتفكك، نقترح طحن الخلايا المجمدة مع هاون والحشرات في وجود أكسيد الألومنيوم لتعطيل ميكانيكيا جدار الخلية. في هذا البروتوكول، لا يلزم وسادة السكروز أو جهاز طرد فائق الانحدار السكروز لتنقية أحادية. بدلا من ذلك، يتم تطبيق فصل مرنا باستخدام البولي أكريلاميد هلام الكهربائي (PAGE) تليها استئصال بصمة ريبوسومال (28-30 NT الفرقة) ويوفر نتائج مرضية. هذا يبسط إلى حد كبير الأسلوب وكذلك يقلل من الوقت ومتطلبات المعدات لهذا الإجراء. لإعداد المكتبة، نوصي باستخدام مجموعة الحمض النووي الريبي الصغيرة المتاحة تجاريا لتسلسل Illumina من نيو إنجلاند Biolabs، باتباع إرشادات الشركة المصنعة مع درجة ما من التحسين.
مكتبات cDNA الناتجة تقدم الكمية والنوعية المناسبة المطلوبة لتسلسل الجيل التالي (NGS). ويؤدي تسلسل المكتبات المعدة وفقا لنتائج البروتوكول الموصوف إلى أن يقرأ 2 إلى 10 ملايين من الملونات التي تم تعيينها بشكل فريد لكل عينة، مما يوفر بيانات كافية للتحليل المعلوماتي الحيوي الشامل. البروتوكول الذي نقدمه سريع وسهل نسبيا ويمكن تنفيذه بمعدات مختبرية قياسية.
Introduction
تم تطوير تقنية التنميط الريبوسوم (RIBO-seq) في مختبر جوناثان ويسمان في جامعة كاليفورنيا ، سان فرانسيسكو1. بالمقارنة مع الأساليب الأخرى المستخدمة لدراسة التعبير الجيني على المستوى الترجمي ، يركز RIBO-seq على كل ربط ريبوسوم ل mRNA ويوفر معلومات حول موقعه والعدد النسبي للريبوسومات على نسخة. أنها تمكن من رصد عملية تخليق البروتين في الجسم الحي، ويمكن أن توفر دقة كودون واحد ودقة السماح لقياس كثافة الريبوسوم على حد سواء، مرنا الفردية وعلى طول النسخ الكامل في الخلية. في الأساس لتقنية ريبو-seq يكمن حقيقة أنه أثناء الترجمة يربط ريبوسوم جزيء مرنا وبالتالي يحمي جزء مدفون من النص من عملية الهضم ريبونوكليز. عند إضافة ريبونوكليز، يتم هضم الحمض النووي الريبي غير المحمي وتظل الشظايا المغلقة بالريبوسومات – التي يبلغ طولها عادة ~ 28-30 nt – سليمة. ويمكن بعد ذلك عزل هذه الشظايا، التي تسمى آثار أقدام ريبوسومية (RF)، وتسلسلها ورسم خرائط لها على النص الذي نشأت عنه مما أدى إلى الكشف عن الموضع الدقيق للريبوسومات. في الواقع، وقد استخدمت القدرة على حماية ريبوسوم شظايا مرنا منذ 1960s لدراسة الربط ribosomal ومواقع بدء الترجمة (TIS)2،3،4. ومع ذلك ، مع التقدم في تكنولوجيا التسلسل العميق ، أصبح RIBO-seq معيارا ذهبيا لرصد الترجمة5 ، والذي ، من خلال المشاركة الريبوسومية ، يمكن أن يوفر معلومات على نطاق الجينوم حول تخليق البروتين6. ريبوسوم التنميط سد الفجوة التكنولوجية التي كانت موجودة بين تحديد النسخ والبروتيوم6.
لإجراء التنميط ريبوسوم نحن بحاجة للحصول على lysate الخلية من الكائن الحي الذي نما في ظل الظروف التي تم التحقيق فيها. قد يؤدي تعطيل هذه الظروف أثناء جمع الخلايا وتحللها إلى توفير بيانات غير موثوقة. لمنع ذلك ، يتم استخدام مثبطات الترجمة والحصاد السريع وتجميد الفلاش في النيتروجين السائل. يمكن أن يتم التحلل الخلايا بواسطة طحن المبردة في التجانس الميكانيكية مثل مطحنة خلاط7،8 أو خافق حبة9، والتحوير من خلال ماصة10 أو مع إبرة11. يمكن إضافة المخزن المؤقت للتحلل قبل أو بعد وقت قصير من سحق الخلايا. في بروتوكولنا نستخدم النيتروجين السائل لقذائف الهاون والحشرات ، وكذلك أكسيد الألومنيوم كنهج ألطف لتعطيل جدار الخلية البكتيرية ، والذي يمنع قص الحمض النووي الريبي الذي غالبا ما يواجه عندما يتم تطبيق طرق مثل السونيشن. بعد السحق ، نضيف عازل تحلل بارد الجليد إلى المحتويات المبردة من قذائف الهاون. اختيار العازلة تحلل المناسبة مهم للحصول على أفضل قرار من آثار أقدام ريبوسومات. منذ قوة الأيونية يؤثر على كل من حجم RF ودقة إطار القراءة، فمن المستحسن حاليا لاستخدام المخازن المؤقتة تحلل مع قوة أيونية منخفضة والقدرة العازلة، حتى لو كان يبدو أن تكوين العازلة لا يؤثر على إشغال ريبوسومات على mRNAs11،12. المكونات الهامة للحاجز التحلل هي أيونات المغنيسيوم ، ووجود الذي يمنع تفكك الوحدات الفرعية ريبوسوم ويمنع التغيرات تشكيلية عفوية في ريبوسوماتالبكتيرية 11،13. أيونات الكالسيوم تلعب أيضا دورا هاما وضرورية لنشاط النيوكليز ميكروكوكال (MNase) المستخدمة في التنميط الريبوسوم البكتيرية طريقة14. إضافة جوانوسين 5′-[β,γ-imido]ثلاثي الفوسفات (GMP-PNP), التناظرية غير قابلة للتحلل من GTP, جنبا إلى جنب مع الكلورامفينيكول يمنع الترجمة خلال تحلل15.
عندما يتم الحصول على lysate، يتم توضيحه عن طريق الطرد المركزي وتقسيمها إلى قسمين، كل لريبو-seq وعالية الإنتاجية مجموع تسلسل مرنا (RNA-seq) لأنها تتم في وقت واحد(الشكل 1). يوفر RNA-seq نقطة مرجعية تمكن من مقارنة البيانات من كل من RIBO-seq و RNA-seq أثناء تحليل البيانات. يتم تعريف المترجم التحقيق من خلال تطبيع آثار أقدام ريبوسومات إلى وفرة مرنا16. يمكن أن تساعد البيانات من RNA-seq أيضا في تحديد الاستنساخ أو تسلسل القطع الأثرية17.
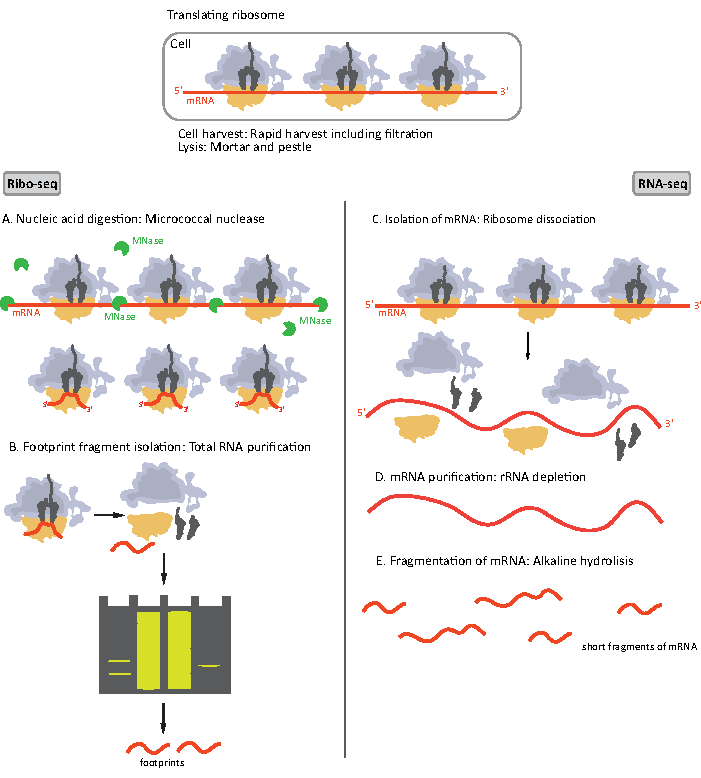
الشكل 1. تخطيطات إعداد عينة مرنا لريبو-seq و RNA-seq. لإعداد مكتبة ريبو-seq، يتم هضم الحمض النووي الريبي مع MNase (A)، تليها اختيار حجم RF من ~ 28-30 NT طول (B)؛ لرنا-seq يتم عزل (C)، استنفدت من rRNA (D)، ويتم تجزئة الحمض النووي الريبي الناتج عشوائيا إلى شظايا من أطوال متفاوتة (E). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الخطوات الأولية لإجراء إعداد عينة لريبو-seq و RNA-seq تختلف قليلا (الشكل 1). بالنسبة للتنميط الريبوسومي ، يحتاج الليسات إلى هضمه بواسطة إندونوكليز محدد لإضعاف جزيئات الحمض النووي الريبي غير المحمية بالريبوسومات. في البروتوكولات القياسية، يتم استرداد أحادية الحصول عليها عن طريق جهاز الطرد الفائق وسادة السكروز أو الطارد الفائق الانحدار السكروز8،14. في هذه المقالة، ونحن نظهر أن هذه الخطوة ليست ضرورية لعزل RF المطلوبة لريبو-seq في البكتيريا، وبالمثل للخلايا eukaryotic18،وأن اختيار حجم شظايا مرنا طول المناسبة من هلام البولي أكريلاميد كافية.
بالنسبة ل RNA-seq ، يتم الحصول على مرنا عن طريق استنفاد الحمض النووي الريبي من إجمالي الحمض النووي الريبي – جزيئات rRNA تهجين إلى مسابير أوليغونوكليوتيد البيوتينية التي ترتبط بالخرز المغناطيسي المغلف بالستريبتافيدين. ثم تتم إزالة مجمعات rRNA-oligonucleotide-حبة من العينة مع مغناطيس مما أدى إلى rRNA المنضب عينة19،20. ثم يتم تجزئة جزيئات الحمض النووي الريبي المنقى عشوائيا بواسطة التحلل المائي القلوية. يتم تحويل الأجزاء التي تم الحصول عليها من مرنا وكذلك آثار أقدام ريبوسومات إلى مكتبات cDNA وإعدادها لتسلسل عميق(الشكل 2). وهذا ينطوي على إصلاح ينتهي اللازمة بعد التحلل المائي القلوية (لرنا) والهضم الأنزيمي (لRF): dephosphorylation من 3 ‘ينتهي تليها الفوسفور من نهايات 5’. الخطوات التالية هي ربط المحولات والنسخ العكسي لإنشاء إدراجات cDNA مؤطرة بتسلسل مطلوب لتسلسل الجيل التالي (NGS) باستخدام منصة Illumina. المرحلة الأخيرة من إعداد المكتبة هي رد فعل PCR حيث يتم تضخيم البنى ووضع علامات عليها مع عينة من الباركود المحدد للسماح بتعدد وتسلسل عينات مختلفة على قناة واحدة. قبل التسلسل، يتم تقييم جودة وكمية المكتبات من خلال الحمض النووي عالي الحساسية على رقاقة الكهربائي. يمكن بعد ذلك تجميع مكتبات cDNA ذات المعلمات المناسبة وتسلسلها. يمكن إجراء التسلسل على منصات Illumina مختلفة، مثل MiSeq، NextSeq أو HighSeq، اعتمادا على عدد المكتبات، وطول القراءة المطلوبة وعمق التسلسل. بعد التسلسل، يتم إجراء التحليل المعلوماتي الحيوي.

الشكل 2. إعداد المكتبة. يتضمن إعداد المكتبة إصلاح النهايات وربط المحولات والنسخ العكسي والتضخيم باستخدام ترميز. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
التنميط ريبوسوم هو أسلوب عالمي التي يمكن تعديلها بسهولة وتعديلها وفقا للمسألة العلمية. في الأصل كان يستخدم في الخميرة1، ولكن بعد فترة وجيزة تم تطبيقه على الخلايا البكتيرية21 وكذلك الكائنات الحية نموذج eukaryotic بما في ذلك الماوس10، حمار وحشي22، ذبابة الفاكهة23 و Arabidopsis thaliana24. كما تم استخدامه لدراسة أنواع مختلفة من الريبوسوم: السيتوبلازمية، الميتوكوندريا25،26 وكلوروبلاست27،28. في eukaryotes RIBO-seq عادة ما يتم تكييفها وصقلها للتحقيق في جوانب محددة من الترجمة، بما في ذلك بدء10،11،29،30،31،32، استطالة1،10،11،31،33، ريبوسوم المماطلة33 وتغيير تشكيل33. معظم التعديلات تنطوي على استخدام مثبطات الترجمة المختلفة. في البكتيريا ومع ذلك، كانت الدراسات المماثلة من الصعب إجراء بسبب ندرة مثبطات مع الآلية المطلوبة للعمل34. مثبط الترجمة الأكثر استخداما في البكتيريا هو الكلورامفينيكول (CAM) الذي يرتبط بمركز نقل بيبتيديل (PTC) ويمنع تحديد المواقع الصحيحة لل AMINOACYL-tRNA في الموقع A. ونتيجة لذلك، يمنع CAM تشكيل رابطة الببتيد مما يؤدي إلى اعتقال ريبوسومات ممدود35. أمثلة أخرى من مثبطات الترجمة في البكتيريا هي التتراسيكلين (TET)36، retapamulin (RET)34 و Onc11237 التي استخدمت للتحقيق في مواقع بدء الترجمة. TET، الذي يمنع تسليم الحمض النووي الريبي إلى ريبوسوم عن طريق التداخل المباشر مع حلقة الجذعية anticodon من tRNA في الموقع A، تم تطبيقها أصلا للتحقق من النتائج التي تم الحصول عليها من العلاج CAM لأنها على حد سواء المضادات الحيوية تثبيط ترجمة استطالة38. تم العثور على TET للكشف عن الأولية، ولكن لم يتمكن من الكشف عن TIS36الداخلية. يربط RET في PTC من الريبوسوم البكتيري ، ويمنع تكوين أول رابطة الببتيد عن طريق التدخل في استطالة أميناسيل -tRNA في الموقع A. تطبيق نتائج RET في اعتقال الريبوسومات في كل من الابتدائية وكذلك الداخلية TISS34. Onc112، ببتيد مضاد للميكروبات غني بالبرولين، يربط في نفق الخروج ويمنع ربط أمينواسيل-ترنا في موقع ريبوسومال A. ونتيجة لذلك، يمنع Onc112 مجمعات البدء من دخول مرحلة الاستطالة37.
المعلومات الرئيسية التنميط ريبوسوم يوفر هو كثافة الريبوسومات وموقفها على مرنا. تم تطبيقه بنجاح للتحقيق في التعبير الجيني التفاضلي على مستوى الترجمة في ظروف النمو المختلفة1،6، قياس الكفاءة الترجمية1،38،39 والكشف عن أحداث تنظيم الترجمة مثل التوقف الريبوسومي10. RIBO-seq يسمح أيضا للكشف عن ترجمة ncRNA المشروح، pseudogenes وإطارات القراءة المفتوحة الصغيرة غير معطاة (ORF) مما يؤدي إلى تحديد الجينات الرواية و / أو قصيرة جدا البروتين الترميز10،12،22،30،37. في مثل هذه الحالات، يمكن RIBO-seq صقل وتحسين التعليقات التوضيحية الجينوم. مع حساسيته العالية لتحديد ORFs المترجمة وطبيعتها الكمية ، يمكن أن يكون التنميط الريبوسوم أيضا بمثابة وكيل لتحديد البروتيوم أو في مساعدة دراسات البروتيوميات31،34،39. عن طريق رسم خرائط TIS، التنميط ريبوسوم يكشف N-ميؤوس منها isoforms الموسعة والمقتطعة من البروتينات المعروفة10،32. كما تم تكييف RIBO-seq لدراسة الطي المترجم المشترك للبروتينات14و21و24. تمكن هذه الطريقة من قياس معدلات الاستطالة1،10،39 أو فك سرعة codons الفردية6 وتساعد في تطوير نماذج كمية للترجمة17. طريقة التنميط ريبوسوم هي أيضا قادرة على توفير رؤى ميكانيكية في التوقف ريبوسوم في البكتيريا7،15،17، frameshifting40، وقف كودون قراءة21، إنهاء / إعادة تدوير العيوب41،42 وتشكيل ريبوسومات التغييرات33 في eukaryotes. كما تم تكييف RIBO-seq لدراسة تأثير عوامل محددة عبر المفعول على الترجمة مثلميرناس 6 والبروتينات الرابطة للجيش الملكي النيبالي في eukaryotes16،43. ومع ذلك، من المهم أن نعترف بأن التصميم التجريبي والقرار الذي تم الحصول عليه من ريبو-seq تحديد كمية المعلومات التي يمكن استخلاصها من البيانات الناتجة التسلسل12.
Protocol
Representative Results
Discussion
التحدي التقني الرئيسي للتنميط ريبوسوم هو الحاجة إلى منع الترجمة بسرعة من أجل التقاط لقطة من الريبوسومات على الحمض النووي الريبي في حالة فسيولوجية معينة من الاهتمام. ولتحقيق ذلك، تستخدم مثبطات الترجمة والحصاد السريع وتجميد الفلاش في النيتروجين السائل بشكل شائع. تطبيق المضادات الحيوية اخ…
Disclosures
The authors have nothing to disclose.
Acknowledgements
تود ALS الاعتراف بالدعم المالي لمنح تثبيت EMBO IG 3914 وPOIR. 04.04.00-00-3E9C/17-00 نفذت في إطار برنامج الفريق الأول لمؤسسة العلوم البولندية التي يشارك في تمويلها الاتحاد الأوروبي في إطار الصندوق الأوروبي للتنمية الإقليمية.
Materials
| 10X TBE (powder) | Invitrogen | AM9864 | |
| 2-Mercaptoethanol, 99%, pure | Acros Organics | 125472500 | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| Aluminium oxide calcinated pure p.a. | Chempur | 114560600 | |
| Calcium chloride dihydrate | Sigma-Aldrich | C3881-500G | |
| Chloramphenicol | MP Biomedicals | 190321 | |
| DNA Clean & Concentrator -5 | Zymo Research | D4004 | |
| Dnase I recombinant, Rnase-free | Roche | 4716728001 | |
| EDTA disodium salt | Fisher Scientific | E/P140/48 | |
| Ethyl Alcohol Absolut 99,8% Pure-P.A.-Basic | POCH Avantor Performance Materials Poland S.A | BA6480111 | |
| Filtration apparatus | VWR Collection | 511-0265 | all-glass filtration apparatus, with funnel, fritted base, cap, 47 mm Ø spring clamp and ground joint flask |
| Gel 40 (19:1) | Rotiphorese | 3030.1 | |
| Gel Loading Dye, Blue, 6X | New England Biolabs | E6138G | |
| Guanosine 5′-[β,γ-imido]triphosphate trisodium salt hydrate | Sigma-Aldrich | G0635-25MG | |
| labZAP | A&A Biotechnology | 040-500 | |
| Magnesium acetate tetrahydrate | Sigma-Aldrich | M5661-250G | |
| MCE membrane fiter | Alfatec Technology | M47MCE45GWS | pore size: 0.45um |
| MICROBExpress Bacterial mRNA Purification | Invitrogen | AM1905 | |
| Multiplex Small RNA Library Prep Set for Illumina | New England Biolabs | E7300S | |
| Nuclease-Free Water | Ambion | AM9937 | |
| Potassium Acetate Anhydrous Pure P.A. | POCH Avantor Performance Materials Poland S.A | 744330113 | |
| Quick-Load pBR322 DNA-MspI Digest | New England Biolabs | E7323A | |
| RNA Clean & Concentrator -25 | Zymo Research | R1018 | |
| Sodium acetate | Sigma-Aldrich | S2889-250G | |
| Sodium carbonate | Sigma-Aldrich | 223530-500G | |
| Sodium hydrogen carbonate pure p.a. | POCH Avantor Performance Materials Poland S.A | 810530115 | |
| SYBR Gold nucleic acid gel stain | Life Technologies | S11494 | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| TBE-Urea Sample Buffer (2x) | Invitrogen | LC6876 | |
| Tris(hydroxymethyl)amino-methane, ultrapure, 99,9% | AlfaAesar | J65594 | |
| Triton X-100, 98% | Acros Organics | 327371000 | |
| Urea G.R. | lach:ner | 40096-AP0 |
References
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Takanami, M., Yan, Y., Jukes, T. H. Studies on the site of ribosomal binding of f2 bacteriophage RNA. Journal of Molecular Biology. 12 (3), 761-773 (1965).
- Steitz, J. A. Polypeptide Chain Initiation: Nucleotide Sequences of the Three Ribosomal Binding Sites in Bacteriophage R17 RNA. Nature. 224 (5223), 957-964 (1969).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. The EMBO journal. 7 (11), 3559-3569 (1988).
- Argüello, R. J., et al. SunRiSE – measuring translation elongation at single-cell resolution by means of flow cytometry. Journal of Cell Science. 131 (10), 214346 (2018).
- Michel, A. M., Baranov, P. V. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale. Wiley Interdisciplinary Reviews: RNA. 4 (5), 473-490 (2013).
- Woolstenhulme, C. J., Guydosh, N. R., Green, R., Buskirk, A. R. High-precision analysis of translational pausing by ribosome profiling in bacteria lacking EFP. Cell reports. 11 (1), 13-21 (2015).
- McGlincy, N. J., Ingolia, N. T. Transcriptome-wide measurement of translation by ribosome profiling. Methods (San Diego, Calif). 126, 112-129 (2017).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic acids research. 42 (17), 134 (2014).
- Ingolia, N. T., Lareau, L. F., Weissman, J. S. Ribosome Profiling of Mouse Embryonic Stem Cells Reveals the Complexity of Mammalian Proteomes. Cell. 147 (4), 789-802 (2011).
- Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature protocols. 7 (8), 1534-1550 (2012).
- Hsu, P. Y., et al. Super-resolution ribosome profiling reveals unannotated translation events in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 113 (45), 7126-7135 (2016).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proceedings of the National Academy of Sciences of the United States of America. 101 (35), 12893-12898 (2004).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature protocols. 8 (11), 2212-2239 (2013).
- Li, G. W., Oh, E., Weissman, J. S. The anti-Shine-Dalgarno sequence drives translational pausing and codon choice in bacteria. Nature. 484 (7395), 538-541 (2012).
- King, H. A., Gerber, A. P. Translatome profiling: methods for genome-scale analysis of mRNA translation. Briefings in functional genomics. 15 (1), 22-31 (2014).
- Mohammad, F., Woolstenhulme, C. J., Green, R., Buskirk, A. R. Clarifying the Translational Pausing Landscape in Bacteria by Ribosome Profiling. Cell reports. 14 (4), 686-694 (2016).
- Reid, D. W., Shenolikar, S., Nicchitta, C. V. Simple and inexpensive ribosome profiling analysis of mRNA translation. Methods (San Diego, Calif). 91, 69-74 (2015).
- Sanz, E., Yang, L., Su, T., Morris, D. R., McKnight, G. S., Amieux, P. S. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13939-13944 (2009).
- Petrova, O. E., Garcia-Alcalde, F., Zampaloni, C., Sauer, K. Comparative evaluation of rRNA depletion procedures for the improved analysis of bacterialbiofilm and mixed pathogen culture transcriptomes. Scientific Reports. 7, 41114 (2017).
- Oh, E. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Chew, G. L. Ribosome profiling reveals resemblance between long non-coding RNAs and 5′ leaders of coding RNAs. Development. 140 (13), 2828-2834 (2013).
- Dunn, J. G., Foo, C. K., Belletier, N. G., Gavis, E. R., Weissman, J. S. Ribosome profiling reveals pervasive and regulated stop codon readthrough in Drosophila melanogaster. eLife. 2, 01179 (2013).
- Liu, M. J., et al. Translational landscape of photomorphogenic Arabidopsis. The Plant cell. 25 (10), 3699-3710 (2013).
- Rooijers, K., Loayza-Puch, F., Nijtmans, L. G., Agami, R. Ribosome profiling reveals features of normal and disease-associated mitochondrial translation. Nature Communications. 4, 2886 (2013).
- Danielle, L., et al. Fidelity of translation initiation is required for coordinated respiratory complex assembly. Science Advances. 5 (12), 2118 (2019).
- Zoschke, R., Watkins, K. P., Barkan, A. A rapid ribosome profiling method elucidates chloroplast ribosome behavior in vivo. The Plant cell. 25 (6), 2265-2275 (2013).
- Chotewutmontri, P., Barkan, A. Dynamics of chloroplast translation during chloroplast differentiation in maize. PLoS genetics. 12 (7), 1006106 (2016).
- Lee, S., Liu, B., Huang, S. X., Shen, B., Qian, S. B. Global mapping of translation initiation sites in mammalian cells at single-nucleotide resolution. Proceedings of the National Academy of Sciences of the United States of America. 109 (37), 2424-2432 (2012).
- Martinez, T. F., Chu, Q., Donaldson, C., Tan, D., Shokhirev, M. N., Saghatelian, A. Accurate annotation of human protein-coding small open reading frames. Nature chemical biology. 16 (4), 458-468 (2020).
- Simsek, D., et al. The Mammalian Ribo-interactome Reveals Ribosome Functional Diversity and Heterogeneity. Cell. 169 (6), 1051-1065 (2017).
- Fritsch, C., et al. Genome-wide search for novel human uORFs and N-terminal protein extensions using ribosomal footprinting. Genome research. 22 (11), 2208-2218 (2012).
- Lareau, L. F., Hite, D. H., Hogan, G. J., Brown, P. O. Distinct stages of the translation elongation cycle revealed by sequencing ribosome-protected mRNA fragments. eLife. 3, 01257 (2014).
- Meydan, S., et al. Retapamulin-Assisted Ribosome Profiling Reveals the Alternative Bacterial Proteome. Molecular Cell. 74 (3), 481-493 (2019).
- Wilson, D. N. The A-Z of bacterial translation inhibitors. Critical Reviews in Biochemistry and Molecular Biology. 44, 393-433 (2009).
- Nakahigashi, K. Comprehensive identification of translation start sites by tetracycline-inhibited ribosome profiling. DNA research : an international journal for rapid publication of reports on genes and genomes. 23 (3), 193-201 (2016).
- Weaver, J., Mohammad, F., Buskirk, A. R., Storz, G. Identifying Small Proteins by Ribosome Profiling with Stalled Initiation Complexes. mBio. 10 (2), 02819 (2019).
- Nakahigashi, K. Effect of codon adaptation on codon-level and gene-level translation efficiency in vivo. BMC genomics. 15 (1), 1115 (2014).
- Li, G. W., Burkhardt, D., Gross, C., Weissman, J. S. Quantifying absolute protein synthesis rates reveals principles underlying allocation of cellular resources. Cell. 157 (3), 624-635 (2014).
- Michel, A. M., et al. Observation of dually decoded regions of the human genome using ribosome profiling data. Genome research. 22 (11), 2219-2229 (2012).
- Guydosh, N. R., Green, R. Dom34 rescues ribosomes in 3′ untranslated regions. Cell. 156 (5), 950-962 (2014).
- Young, D. J., Guydosh, N. R., Zhang, F., Hinnebusch, A. G., Green, R. Rli1/ABCE1 Recycles Terminating Ribosomes and Controls Translation Reinitiation in 3’UTRs In Vivo. Cell. 162 (4), 872-884 (2015).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Dobin, A., et al. ultrafast universal RNA seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- . . Babraham Bioinformatics. Fast QC. , (2021).
- Gerashchenko, M. V., Gladyshev, V. N. Ribonuclease selection for ribosome profiling. Nucleic acids research. 45 (2), 6 (2017).
- Verbruggen, S., Menschaert, G. mQC: A post-mapping data exploration tool for ribosome profiling. Computer Methods and Programs in Biomedicine. 181, 104806 (2019).
- Cui, H., Hu, H., Zeng, J., Chen, T. DeepShape: estimating isoform-level ribosome abundance and distribution with Ribo-seq data. BMC bioinformatics. 20, 678 (2019).
- Choi, J. . RiboToolkit: an integrated platform for analysis and annotation of ribosome profiling data to decode RNA translation at codon resolution. , (2021).
- Choi, J. Dynamics of the context-specific translation arrest by chloramphenicol and linezolid. Nature chemical biology. 16 (3), 310-317 (2020).
- Tompson, J., O’Connor, M., Mills, J. A., Dahlberg, A. E. The protein synthesis inhibitors, oxazolidinones and chloramphenicol, cause extensive translational inaccuracy in vivo. Journal of Molecular Biology. 322 (2), 273-279 (2002).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature reviews. Molecular cell biology. 16 (11), 651-664 (2015).
- Glaub, A., Huptas, C., Neuhaus, K., Ardern, Z. Recommendations for bacterial ribosome profiling experiments based on bioinformatic evaluation of published data. Journal of Biological Chemistry. 295, 8999-9011 (2020).
- Sorour, M. H., Hani, H. A., Shaalan, H. F., El-Sayed, M. M. H. Experimental screening of some chelating agents for calcium and magnesium removal from saline solutions. Desalination and Water Treatment. 57 (48-49), 22799-22808 (2015).
- Mohammad, F., Green, R., Buskirk, A. R. A systematically-revised ribosome profiling method for bacteria reveals pauses at single-codon resolution. Elife. 8, 42591 (2019).
- O’Connor, P. B., Li, G. W., Weissman, J. S., Atkins, J. F., Baranov, P. V. rRNA:mRNA pairing alters the length and the symmetry of mRNA-protected fragments in ribosome profiling experiments. Bioinformatics. 29 (12), 1488-1491 (2013).
- Protocol for RNA Clean & Concentrator -25. Zymo Research Available from: https://files.zymoresearch.com/protocols/_r1017_r1018_rna_clean_concentrator-25.pdf (2021)
- Protocol for use with NEBNext Small RNA Library Prep Set for Illumina (E7300, E7580, E7560, E7330). New England Biolabs Available from: https://www.international.neb.com/protocols/2018/03/27/protocol-for-use-with-nebnext-small-rna-library-prep-set-for-illumina-e7300-e7580-e7560-e7330 (2021)

