RIBO-seq in Bacteria: een Sample Collection and Library Preparation Protocol voor NGS Sequencing
Summary
Hier beschrijven we de stadia van monsterverzameling en voorbereiding voor RIBO-seq bij bacteriën. Sequencing van de bibliotheken die volgens deze richtlijnen zijn opgesteld, resulteert in voldoende gegevens voor een uitgebreide bio-informatische analyse. Het protocol dat we presenteren is eenvoudig, maakt gebruik van standaard laboratoriumapparatuur en duurt zeven dagen van lysis tot het verkrijgen van bibliotheken.
Abstract
De ribosoomprofileringstechniek (RIBO-seq) is momenteel het meest effectieve hulpmiddel voor het bestuderen van het proces van eiwitsynthese in vivo. Het voordeel van deze methode, in vergelijking met andere benaderingen, is het vermogen om de vertaling te controleren door de positie en het aantal ribosomen op een mRNA-transcript nauwkeurig in kaart te brengen.
In dit artikel beschrijven we de opeenvolgende stadia van monsterverzameling en voorbereiding voor de RIBO-seq-methode bij bacteriën, waarbij we de details benadrukken die relevant zijn voor de planning en uitvoering van het experiment.
Aangezien de RIBO-seq afhankelijk is van intacte ribosomen en gerelateerde mRNAs, is de belangrijkste stap een snelle remming van de vertaling en adequate desintegratie van cellen. Daarom raden we filtratie en flash-freezing in vloeibare stikstof voor celoogst aan met een optionele voorbehandeling met chlooramfenicol om vertaling in bacteriën te stoppen. Voor de desintegratie stellen we voor bevroren cellen te slijpen met mortel en stamper in aanwezigheid van aluminiumoxide om de celwand mechanisch te verstoren. In dit protocol is sucrosekussen of een sucrose gradiënt ultracentrifugatie voor monose zuivering niet vereist. In plaats daarvan wordt mRNA-scheiding met polyacrylamidegelelektroforese (PAGE) gevolgd door de ribosomale voetafdruk excisie (28-30 nt-band) toegepast en levert bevredigende resultaten op. Dit vereenvoudigt de methode grotendeels en vermindert de tijd- en uitrustingsvereisten voor de procedure. Voor de voorbereiding van de bibliotheek raden we aan om de in de handel verkrijgbaar kleine RNA-kit voor Illumina-sequencing van New England Biolabs te gebruiken, volgens de richtlijnen van de fabrikant met een zekere mate van optimalisatie.
De resulterende cDNA-bibliotheken bieden de juiste hoeveelheid en kwaliteit die nodig zijn voor de volgende generatie sequencing (NGS). Sequencing van de bibliotheken die volgens het beschreven protocol zijn voorbereid, resulteert in 2 tot 10 mln uniek in kaart gebrachte leespunten per monster die voldoende gegevens opleveren voor een uitgebreide bio-informatische analyse. Het protocol dat wij presenteren is snel en relatief eenvoudig en kan worden uitgevoerd met standaard laboratoriumapparatuur.
Introduction
De ribosoomprofileringstechniek (RIBO-seq) is ontwikkeld in het laboratorium van Jonathan Weissman aan de Universiteit van Californië, San Francisco1. In vergelijking met andere methoden die worden gebruikt om genexpressie op translationeel niveau te bestuderen, richt RIBO-seq zich op elke ribosoombinding aan mRNA en geeft informatie over de locatie en het relatieve aantal ribosomen op een transcript. Het maakt het mogelijk om het proces van eiwitsynthese in vivo te volgen en kan een enkele codonresolutie en nauwkeurigheid bieden, waardoor de ribosoomdichtheid op zowel het individuele mRNA als langs het hele transcriptoom in de cel kan worden meten. Aan de basis van de RIBO-seq techniek ligt het feit dat het ribosoom tijdens de vertaling het mRNA molecuul bindt en zo het begraven fragment van het transcript beschermt tegen een ribonuclease spijsvertering. Bij toevoeging van de ribonuclease wordt het onbeschermde mRNA verteerd en blijven de fragmenten omsloten door ribosomen – meestal van ~ 28-30 nt lang – intact. Deze fragmenten, ribosomale voetafdrukken (RF) genoemd, kunnen vervolgens worden geïsoleerd, gesequenced en toegewezen aan het transcript waaruit ze afkomstig zijn, wat resulteert in de detectie van de exacte positie van de ribosomen. In feite wordt het ribosoomvermogen om mRNA-fragmenten te beschermen sinds de jaren zestig gebruikt om ribosomale bindings – en vertaalinitiatieplaatsen (TIS)2,3,4te bestuderen . Met de vooruitgang in diepe sequencingtechnologie is RIBO-seq echter een gouden standaard geworden voor vertaalmonitoring5 die, door de ribosoombetrokkenheid, een genoombrede informatie over eiwitsynthese kan bieden6. Ribosoomprofilering vulde de technologische kloof die bestond tussen het kwantificeren van het transcriptoom en het proteoom6.
Om ribosoomprofilering uit te voeren, moeten we celllysaat verkrijgen van het organisme dat onder de onderzochte omstandigheden was gegroeid. Het verstoren van deze omstandigheden tijdens het verzamelen en lyse van cellen kan onbetrouwbare gegevens opleveren. Om dit te voorkomen, worden vaak vertaalremmers, snel oogsten en vlamvriezen in vloeibare stikstof gebruikt. Cellen kunnen worden gelyseerd door cryogene slijping in een mechanische homogenisator zoals een mengmolen7,8 of een kraalklopper9, en door trituratie door een pipet10 of met een naald11. De lysisbuffer kan vlak voor of kort na verpulvering van de cellen worden toegevoegd. In ons protocol gebruiken we vloeibare stikstof om mortel en stamper voor tekoelen, evenals aluminiumoxide als een zachtere benadering van verstoring van de bacteriële celwand, die voorkomt dat RNA-schaar vaak wordt aangetroffen wanneer methoden zoals sonificatie worden toegepast. Na verpulvering voegen we een ijskoude lysisbuffer toe aan de gekoelde inhoud van de mortel. Selectie van een geschikte lysisbuffer is belangrijk voor het verkrijgen van de beste resolutie van ribosomale voetafdrukken. Aangezien de ionische sterkte zowel de RF-grootte als de precisie van het leesframe beïnvloedt, wordt momenteel aanbevolen om lysisbuffers met een lage ionische sterkte en buffercapaciteit te gebruiken, zelfs als blijkt dat de buffersamenstelling geen invloed heeft op de ribosomale bezetting op mRNAs11,12. Belangrijke componenten van de lysisbuffer zijn magnesiumionen, waarvan de aanwezigheid dissociatie van de ribosomale subeenheden voorkomt en spontane conformatieveranderingen in de bacteriële ribosomen11,13remt . Calciumionen spelen ook een belangrijke rol en zijn essentieel voor de activiteit van microkokkennuclease (MNase) die wordt gebruikt in de bacteriële ribosoomprofileringsmethode14. Toevoeging van guanosine 5′-[β,γ-imido]trifosfaat (GMP-PNP), een niet-hydrolyzable analoog van GTP, samen met chlooramfenicol remt de vertaling tijdens lysis15.
Wanneer het lysaat wordt verkregen, wordt het verduidelijkt door centrifugeren en verdeeld in twee porties, elk voor een RIBO-seq en een totale mRNA-sequencing (RNA-seq) met hoge doorvoer, omdat ze tegelijkertijd worden uitgevoerd (figuur 1). RNA-seq biedt een referentiepunt waarmee gegevens van zowel RIBO-seq als RNA-seq tijdens gegevensanalyse kunnen worden vergeleken. Het onderzochte vertaaloom wordt gedefinieerd door normalisatie van ribosomale voetafdrukken naar mRNA-overvloed16. Gegevens van RNA-seq kunnen ook helpen bij het identificeren van kloon- of sequencingartefacten17.
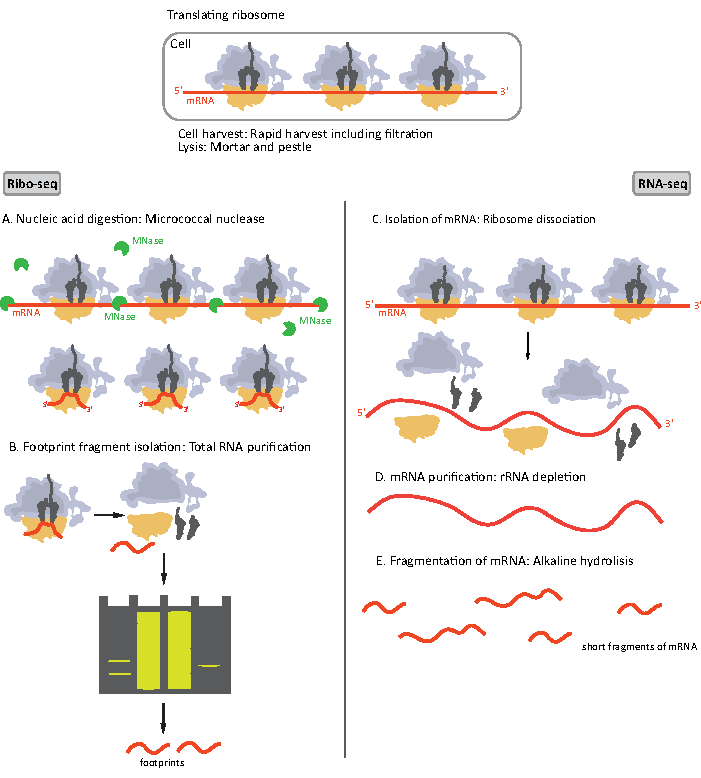
Figuur 1. Schema’s van mRNA monstervoorbereiding voor RIBO-seq en RNA-seq. Voor ribo-seq bibliotheekvoorbereiding, wordt RNA verteerd met MNase (A), gevolgd door de grootteselectie van RF van ~28-30 nt lengte (B); voor RNA-seq is RNA geïsoleerd (C), uitgeput van rRNA (D), en het resulterende mRNA wordt willekeurig gefragmenteerd in fragmenten van verschillende lengtes (E). Klik hier om een grotere versie van deze afbeelding te bekijken.
De eerste stappen van de monstervoorbereidingsprocedure voor RIBO-seq en RNA-seq verschillen enigszins (figuur 1). Voor de ribosomale profilering moet het lysaat worden verteerd door een specifiek endonuclease om de mRNA-moleculen af te voeren die niet door de ribosomen worden beschermd. In standaardprotocollen worden de verkregen monosomen teruggewonnen door een sucrosekussen ultracentrifugatie of een sucrose gradiënt ultracentrifugatie8,14. In dit artikel laten we zien dat deze stap niet nodig is om RF te isoleren die nodig is voor de RIBO-seq in bacteriën, ook voor eukaryotische cellen18, en dat de grootteselectie van de juiste lengte mRNA-fragmenten uit de polyacrylamidegel voldoende is.
Voor RNA-seq wordt mRNA verkregen door de uitputting van rRNA uit het totale RNA – rRNA moleculen hybridiseren naar de biotinylated oligonucleotide sondes die zich binden aan de streptavidin gecoate magnetische kralen. De rRNA-oligonucleotide-kraalcomplexen worden vervolgens met een magneet uit het monster verwijderd , wat resulteert in een rRNA-uitgeput monster19,20. De gezuiverde mRNA-moleculen worden vervolgens willekeurig gefragmenteerd door alkalische hydrolyse. De verkregen fragmenten van mRNA en de ribosomale voetafdrukken worden omgezet in cDNA-bibliotheken en voorbereid op diepe sequencing (figuur 2). Dit omvat eindreparatie die nodig is na alkalische hydrolyse (voor mRNA) en enzymatische vergisting (voor RF): defosforylatie van 3′ uiteinden gevolgd door fosforylering van 5′ uiteinden. De volgende stappen zijn adapters ligatie en de omgekeerde transcriptie om cDNA-inserts te maken die zijn omlijst door sequenties die nodig zijn voor de volgende generatie sequencing (NGS) met behulp van het Illumina-platform. De laatste fase van de voorbereiding van de bibliotheek is een PCR-reactie waarbij de constructies worden versterkt en geëtiketteerd met monsterspecifieke barcodes om multiplexing en sequencing van verschillende monsters op één kanaal mogelijk te maken. Voor de sequencing worden de kwaliteit en kwantiteit van de bibliotheken beoordeeld door de hooggevoelige DNA-on-chip elektroforese. cDNA-bibliotheken met de juiste parameters kunnen vervolgens worden samengevoegd en gesequenced. Sequencing kan worden uitgevoerd op verschillende Illumina-platforms, zoals MiSeq, NextSeq of HighSeq, afhankelijk van het aantal bibliotheken, de vereiste leeslengte en sequencingdiepte. Na sequencing wordt de bio-informatische analyse uitgevoerd.

Figuur 2. Bibliotheek voorbereiding. Bibliotheekvoorbereiding omvat de eindreparatie, adapters ligatie, omgekeerde transcriptie en versterking met barcodering. Klik hier om een grotere versie van deze afbeelding te bekijken.
De ribosoomprofilering is een universele methode die eenvoudig kan worden aangepast en aangepast aan de wetenschappelijke vraag. Oorspronkelijk werd het gebruikt in gist1, maar kort daarna werd het toegepast op bacteriële cellen21 evenals eukaryotische modelorganismen, waaronder muis10, zebravis22, fruitvlieg23 en Arabidopsis thaliana24. Het werd ook gebruikt voor het bestuderen van verschillende ribosoomtypen: cytoplasmatisch, mitochondriaal25,26 en chloroplast27,28. In eukaryoten wordt RIBO-seq gewoonlijk aangepast en verfijnd om specifieke aspecten van vertaling te onderzoeken, waaronder initiatie10,11,29,30,31,32, verlenging1,10,11,31,33, ribosoomstalling 33 en exterieurverandering33. De meeste wijzigingen betreffen het gebruik van verschillende vertaalremmers. Bij bacteriën zijn analoge studies echter moeilijk uit te voeren vanwege het gebrek aan remmers met het vereiste werkingsmechanisme34. De meest gebruikte vertaalremmer bij bacteriën is chlooramfenicol (CAM) dat zich bindt aan het peptidyltransferasecentrum (PTC) en de juiste positionering van het aminoacyl-tRNA in de A-site voorkomt. Als gevolg hiervan voorkomt CAM de vorming van een peptidebinding die leidt tot het arresteren van de langwerpige ribosomen35. Andere voorbeelden van vertaalremmers bij bacteriën zijn tetracycline (TET)36, retapamulin (RET)34 en Onc11237 die zijn gebruikt om vertaalinitiatieplaatsen te onderzoeken. TET, dat de toediening van tRNA aan het ribosoom voorkomt door direct te overlappen met de anticodonstamlus van tRNA op de A-site, werd oorspronkelijk toegepast om de resultaten van de CAM-behandeling te verifiëren, omdat het beide antibiotica zijn die de verlenging van de vertaling remmen38. TET bleek primaire TIS te detecteren, maar kon interne TIS36niet onthullen . RET bindt in de PTC van het bacteriële ribosoom en voorkomt de vorming van de eerste peptidebinding door te interfereren met een elongator aminoacyl-tRNA in de A-site. Het toepassen van RET resulteert in ribosomen arrestatie op zowel primaire als interne TISs34. Onc112, een proline-rijk antimicrobieel peptide, bindt in de uitgangstunnel en blokkeert aminoacyl-tRNA binding in de ribosomale A-site. Als gevolg hiervan voorkomt Onc112 dat initiatiecomplexen de verlengingsfase37ingaan .
De belangrijkste informatie die ribosoomprofilering biedt, is de ribosomdichtheid en hun positie op het mRNA. Het werd met succes toegepast om differentiële genexpressie op het niveau van vertaling te onderzoeken in verschillende groeiomstandigheden1,6, translationele efficiëntie1,38,39 te meten en vertaalregelgebeurtenissen zoals ribosomale pauze10te detecteren . RIBO-seq maakt het ook mogelijk om de vertaling van geannoteerde ncRNA, pseudogenen en ongeannoteerde kleine open leeskaders (ORF) te ontdekken die leiden tot de identificatie van nieuwe en/of zeer korte eiwitcoderingsgenen10,12,22,30,37. In dergelijke gevallen kan RIBO-seq de annotatie van het genoom verfijnen en verbeteren. Met zijn hoge gevoeligheid voor de identificatie van vertaalde ORF ‘s en zijn kwantitatieve aard kan ribosoomprofilering ook dienen als proxy voor de proteoombepaling of bij het helpen van proteomicsstudies31,34,39. Door TIS in kaart te brengen, onthult ribosoomprofilering N-terminaal verlengde en afgeknotte isovormen van bekende eiwitten10,32. RIBO-seq werd ook aangepast om co-translationele vouwen van eiwitten14,21,24 te bestuderen. Deze methode maakt het meten van reksnelheden1,10,39 of decoderingssnelheden van individuele codons6 mogelijk en helpt bij het ontwikkelen van kwantitatieve vertaalmodellen17. De ribosoomprofileringsmethode is ook in staat om mechanistische inzichten te geven in de ribosoomonderbreking bij bacteriën7,15,17, frameshifting40, stop-codon readthrough21, beëindiging / recyclingdefecten41,42 en ribosomale conformatieveranderingen33 bij eukaryoten. RIBO-seq werd ook aangepast om het effect van specifieke trans-acterende factoren op vertalingen zoals miRNAs6 en RNA-bindende eiwitten in eukaryoten16,43teonderzoeken . Het is echter belangrijk om te erkennen dat het experimentele ontwerp en de verkregen resolutie van RIBO-seq bepalend zijn voor de hoeveelheid informatie die kan worden geëxtraheerd uit de resulterende sequencinggegevens12.
Protocol
Representative Results
Discussion
De belangrijkste technische uitdaging van de ribosoomprofilering is de noodzaak om de vertaling snel te remmen om een momentopname van ribosomen op mRNAs in een bepaalde fysiologische staat van belang vast te leggen. Om dit te bereiken, worden vertaalremmers, snel oogsten en vlamvriezen in vloeibare stikstof vaak gebruikt. Het toepassen van antibiotica is optioneel omdat ze artefacten kunnen veroorzaken. Chlooramfenicol is een veelgebruikt medicijn om langwerpige ribosomen in bacteriële RIBO-seq te arresteren. Het belet…
Disclosures
The authors have nothing to disclose.
Acknowledgements
ALS wil graag de financiële steun van EMBO Installatiesubsidies IG 3914 en POIR erkennen. 04.04.00-00-3E9C/17-00 uitgevoerd in het kader van het Eerste TEAM-programma van de Stichting voor Poolse Wetenschap, medegefinancierd door de Europese Unie in het kader van het Europees Fonds voor Regionale Ontwikkeling.
Materials
| 10X TBE (powder) | Invitrogen | AM9864 | |
| 2-Mercaptoethanol, 99%, pure | Acros Organics | 125472500 | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| Aluminium oxide calcinated pure p.a. | Chempur | 114560600 | |
| Calcium chloride dihydrate | Sigma-Aldrich | C3881-500G | |
| Chloramphenicol | MP Biomedicals | 190321 | |
| DNA Clean & Concentrator -5 | Zymo Research | D4004 | |
| Dnase I recombinant, Rnase-free | Roche | 4716728001 | |
| EDTA disodium salt | Fisher Scientific | E/P140/48 | |
| Ethyl Alcohol Absolut 99,8% Pure-P.A.-Basic | POCH Avantor Performance Materials Poland S.A | BA6480111 | |
| Filtration apparatus | VWR Collection | 511-0265 | all-glass filtration apparatus, with funnel, fritted base, cap, 47 mm Ø spring clamp and ground joint flask |
| Gel 40 (19:1) | Rotiphorese | 3030.1 | |
| Gel Loading Dye, Blue, 6X | New England Biolabs | E6138G | |
| Guanosine 5′-[β,γ-imido]triphosphate trisodium salt hydrate | Sigma-Aldrich | G0635-25MG | |
| labZAP | A&A Biotechnology | 040-500 | |
| Magnesium acetate tetrahydrate | Sigma-Aldrich | M5661-250G | |
| MCE membrane fiter | Alfatec Technology | M47MCE45GWS | pore size: 0.45um |
| MICROBExpress Bacterial mRNA Purification | Invitrogen | AM1905 | |
| Multiplex Small RNA Library Prep Set for Illumina | New England Biolabs | E7300S | |
| Nuclease-Free Water | Ambion | AM9937 | |
| Potassium Acetate Anhydrous Pure P.A. | POCH Avantor Performance Materials Poland S.A | 744330113 | |
| Quick-Load pBR322 DNA-MspI Digest | New England Biolabs | E7323A | |
| RNA Clean & Concentrator -25 | Zymo Research | R1018 | |
| Sodium acetate | Sigma-Aldrich | S2889-250G | |
| Sodium carbonate | Sigma-Aldrich | 223530-500G | |
| Sodium hydrogen carbonate pure p.a. | POCH Avantor Performance Materials Poland S.A | 810530115 | |
| SYBR Gold nucleic acid gel stain | Life Technologies | S11494 | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| TBE-Urea Sample Buffer (2x) | Invitrogen | LC6876 | |
| Tris(hydroxymethyl)amino-methane, ultrapure, 99,9% | AlfaAesar | J65594 | |
| Triton X-100, 98% | Acros Organics | 327371000 | |
| Urea G.R. | lach:ner | 40096-AP0 |
References
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Takanami, M., Yan, Y., Jukes, T. H. Studies on the site of ribosomal binding of f2 bacteriophage RNA. Journal of Molecular Biology. 12 (3), 761-773 (1965).
- Steitz, J. A. Polypeptide Chain Initiation: Nucleotide Sequences of the Three Ribosomal Binding Sites in Bacteriophage R17 RNA. Nature. 224 (5223), 957-964 (1969).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. The EMBO journal. 7 (11), 3559-3569 (1988).
- Argüello, R. J., et al. SunRiSE – measuring translation elongation at single-cell resolution by means of flow cytometry. Journal of Cell Science. 131 (10), 214346 (2018).
- Michel, A. M., Baranov, P. V. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale. Wiley Interdisciplinary Reviews: RNA. 4 (5), 473-490 (2013).
- Woolstenhulme, C. J., Guydosh, N. R., Green, R., Buskirk, A. R. High-precision analysis of translational pausing by ribosome profiling in bacteria lacking EFP. Cell reports. 11 (1), 13-21 (2015).
- McGlincy, N. J., Ingolia, N. T. Transcriptome-wide measurement of translation by ribosome profiling. Methods (San Diego, Calif). 126, 112-129 (2017).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic acids research. 42 (17), 134 (2014).
- Ingolia, N. T., Lareau, L. F., Weissman, J. S. Ribosome Profiling of Mouse Embryonic Stem Cells Reveals the Complexity of Mammalian Proteomes. Cell. 147 (4), 789-802 (2011).
- Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature protocols. 7 (8), 1534-1550 (2012).
- Hsu, P. Y., et al. Super-resolution ribosome profiling reveals unannotated translation events in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 113 (45), 7126-7135 (2016).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proceedings of the National Academy of Sciences of the United States of America. 101 (35), 12893-12898 (2004).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature protocols. 8 (11), 2212-2239 (2013).
- Li, G. W., Oh, E., Weissman, J. S. The anti-Shine-Dalgarno sequence drives translational pausing and codon choice in bacteria. Nature. 484 (7395), 538-541 (2012).
- King, H. A., Gerber, A. P. Translatome profiling: methods for genome-scale analysis of mRNA translation. Briefings in functional genomics. 15 (1), 22-31 (2014).
- Mohammad, F., Woolstenhulme, C. J., Green, R., Buskirk, A. R. Clarifying the Translational Pausing Landscape in Bacteria by Ribosome Profiling. Cell reports. 14 (4), 686-694 (2016).
- Reid, D. W., Shenolikar, S., Nicchitta, C. V. Simple and inexpensive ribosome profiling analysis of mRNA translation. Methods (San Diego, Calif). 91, 69-74 (2015).
- Sanz, E., Yang, L., Su, T., Morris, D. R., McKnight, G. S., Amieux, P. S. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13939-13944 (2009).
- Petrova, O. E., Garcia-Alcalde, F., Zampaloni, C., Sauer, K. Comparative evaluation of rRNA depletion procedures for the improved analysis of bacterialbiofilm and mixed pathogen culture transcriptomes. Scientific Reports. 7, 41114 (2017).
- Oh, E. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Chew, G. L. Ribosome profiling reveals resemblance between long non-coding RNAs and 5′ leaders of coding RNAs. Development. 140 (13), 2828-2834 (2013).
- Dunn, J. G., Foo, C. K., Belletier, N. G., Gavis, E. R., Weissman, J. S. Ribosome profiling reveals pervasive and regulated stop codon readthrough in Drosophila melanogaster. eLife. 2, 01179 (2013).
- Liu, M. J., et al. Translational landscape of photomorphogenic Arabidopsis. The Plant cell. 25 (10), 3699-3710 (2013).
- Rooijers, K., Loayza-Puch, F., Nijtmans, L. G., Agami, R. Ribosome profiling reveals features of normal and disease-associated mitochondrial translation. Nature Communications. 4, 2886 (2013).
- Danielle, L., et al. Fidelity of translation initiation is required for coordinated respiratory complex assembly. Science Advances. 5 (12), 2118 (2019).
- Zoschke, R., Watkins, K. P., Barkan, A. A rapid ribosome profiling method elucidates chloroplast ribosome behavior in vivo. The Plant cell. 25 (6), 2265-2275 (2013).
- Chotewutmontri, P., Barkan, A. Dynamics of chloroplast translation during chloroplast differentiation in maize. PLoS genetics. 12 (7), 1006106 (2016).
- Lee, S., Liu, B., Huang, S. X., Shen, B., Qian, S. B. Global mapping of translation initiation sites in mammalian cells at single-nucleotide resolution. Proceedings of the National Academy of Sciences of the United States of America. 109 (37), 2424-2432 (2012).
- Martinez, T. F., Chu, Q., Donaldson, C., Tan, D., Shokhirev, M. N., Saghatelian, A. Accurate annotation of human protein-coding small open reading frames. Nature chemical biology. 16 (4), 458-468 (2020).
- Simsek, D., et al. The Mammalian Ribo-interactome Reveals Ribosome Functional Diversity and Heterogeneity. Cell. 169 (6), 1051-1065 (2017).
- Fritsch, C., et al. Genome-wide search for novel human uORFs and N-terminal protein extensions using ribosomal footprinting. Genome research. 22 (11), 2208-2218 (2012).
- Lareau, L. F., Hite, D. H., Hogan, G. J., Brown, P. O. Distinct stages of the translation elongation cycle revealed by sequencing ribosome-protected mRNA fragments. eLife. 3, 01257 (2014).
- Meydan, S., et al. Retapamulin-Assisted Ribosome Profiling Reveals the Alternative Bacterial Proteome. Molecular Cell. 74 (3), 481-493 (2019).
- Wilson, D. N. The A-Z of bacterial translation inhibitors. Critical Reviews in Biochemistry and Molecular Biology. 44, 393-433 (2009).
- Nakahigashi, K. Comprehensive identification of translation start sites by tetracycline-inhibited ribosome profiling. DNA research : an international journal for rapid publication of reports on genes and genomes. 23 (3), 193-201 (2016).
- Weaver, J., Mohammad, F., Buskirk, A. R., Storz, G. Identifying Small Proteins by Ribosome Profiling with Stalled Initiation Complexes. mBio. 10 (2), 02819 (2019).
- Nakahigashi, K. Effect of codon adaptation on codon-level and gene-level translation efficiency in vivo. BMC genomics. 15 (1), 1115 (2014).
- Li, G. W., Burkhardt, D., Gross, C., Weissman, J. S. Quantifying absolute protein synthesis rates reveals principles underlying allocation of cellular resources. Cell. 157 (3), 624-635 (2014).
- Michel, A. M., et al. Observation of dually decoded regions of the human genome using ribosome profiling data. Genome research. 22 (11), 2219-2229 (2012).
- Guydosh, N. R., Green, R. Dom34 rescues ribosomes in 3′ untranslated regions. Cell. 156 (5), 950-962 (2014).
- Young, D. J., Guydosh, N. R., Zhang, F., Hinnebusch, A. G., Green, R. Rli1/ABCE1 Recycles Terminating Ribosomes and Controls Translation Reinitiation in 3’UTRs In Vivo. Cell. 162 (4), 872-884 (2015).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Dobin, A., et al. ultrafast universal RNA seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- . . Babraham Bioinformatics. Fast QC. , (2021).
- Gerashchenko, M. V., Gladyshev, V. N. Ribonuclease selection for ribosome profiling. Nucleic acids research. 45 (2), 6 (2017).
- Verbruggen, S., Menschaert, G. mQC: A post-mapping data exploration tool for ribosome profiling. Computer Methods and Programs in Biomedicine. 181, 104806 (2019).
- Cui, H., Hu, H., Zeng, J., Chen, T. DeepShape: estimating isoform-level ribosome abundance and distribution with Ribo-seq data. BMC bioinformatics. 20, 678 (2019).
- Choi, J. . RiboToolkit: an integrated platform for analysis and annotation of ribosome profiling data to decode RNA translation at codon resolution. , (2021).
- Choi, J. Dynamics of the context-specific translation arrest by chloramphenicol and linezolid. Nature chemical biology. 16 (3), 310-317 (2020).
- Tompson, J., O’Connor, M., Mills, J. A., Dahlberg, A. E. The protein synthesis inhibitors, oxazolidinones and chloramphenicol, cause extensive translational inaccuracy in vivo. Journal of Molecular Biology. 322 (2), 273-279 (2002).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature reviews. Molecular cell biology. 16 (11), 651-664 (2015).
- Glaub, A., Huptas, C., Neuhaus, K., Ardern, Z. Recommendations for bacterial ribosome profiling experiments based on bioinformatic evaluation of published data. Journal of Biological Chemistry. 295, 8999-9011 (2020).
- Sorour, M. H., Hani, H. A., Shaalan, H. F., El-Sayed, M. M. H. Experimental screening of some chelating agents for calcium and magnesium removal from saline solutions. Desalination and Water Treatment. 57 (48-49), 22799-22808 (2015).
- Mohammad, F., Green, R., Buskirk, A. R. A systematically-revised ribosome profiling method for bacteria reveals pauses at single-codon resolution. Elife. 8, 42591 (2019).
- O’Connor, P. B., Li, G. W., Weissman, J. S., Atkins, J. F., Baranov, P. V. rRNA:mRNA pairing alters the length and the symmetry of mRNA-protected fragments in ribosome profiling experiments. Bioinformatics. 29 (12), 1488-1491 (2013).
- Protocol for RNA Clean & Concentrator -25. Zymo Research Available from: https://files.zymoresearch.com/protocols/_r1017_r1018_rna_clean_concentrator-25.pdf (2021)
- Protocol for use with NEBNext Small RNA Library Prep Set for Illumina (E7300, E7580, E7560, E7330). New England Biolabs Available from: https://www.international.neb.com/protocols/2018/03/27/protocol-for-use-with-nebnext-small-rna-library-prep-set-for-illumina-e7300-e7580-e7560-e7330 (2021)

