RIBO-seq i bakterier: ett protokoll för insamling av prover och biblioteksförberedelser för NGS-sekvensering
Summary
Här beskriver vi stadierna av provtagning och förberedelse för RIBO-seq i bakterier. Sekvensering av de bibliotek som utarbetats enligt dessa riktlinjer resulterar i tillräckliga data för omfattande bioinformatisk analys. Protokollet vi presenterar är enkelt, använder standard laboratorieutrustning och tar sju dagar från lysis till att få bibliotek.
Abstract
Ribosomprofileringstekniken (RIBO-seq) är för närvarande det mest effektiva verktyget för att studera processen med proteinsyntes in vivo. Fördelen med denna metod, i jämförelse med andra tillvägagångssätt, är dess förmåga att övervaka översättning genom att exakt kartlägga positionen och antalet ribosomer på en mRNA-transkription.
I den här artikeln beskriver vi de på varandra följande stadierna av provtagning och förberedelse för RIBO-seq-metoden i bakterier, vilket belyser de detaljer som är relevanta för planering och genomförande av experimentet.
Eftersom RIBO-seq förlitar sig på intakta ribosomer och relaterade mRNAs, är det viktigaste steget snabb hämning av översättning och adekvat sönderfall av celler. Således föreslår vi filtrering och blixtfrysning i flytande kväve för cellskörd med en valfri förbehandling med kloramfenikol för att stoppa översättning i bakterier. För upplösningen föreslår vi slipning av frusna celler med mortel och mortel i närvaro av aluminiumoxid för att mekaniskt störa cellväggen. I detta protokoll krävs inte sackaroskudde eller sackarosgradient ultracentrifugation för monosome rening. Istället appliceras mRNA-separation med polyakrylamidgelelektrofores (PAGE) följt av ribosomal fotavtryck excision (28-30 nt band) och ger tillfredsställande resultat. Detta förenklar till stor del metoden samt minskar tids- och utrustningskraven för förfarandet. För biblioteksförberedelse rekommenderar vi att du använder det kommersiellt tillgängliga lilla RNA-kitet för Illumina-sekvensering från New England Biolabs, enligt tillverkarens riktlinjer med viss grad av optimering.
De resulterande cDNA-biblioteken presenterar lämplig kvantitet och kvalitet som krävs för nästa generations sekvensering (NGS). Sekvensering av de bibliotek som utarbetats enligt det beskrivna protokollet resulterar i 2 till 10 mln unikt kartlagda läsningar per prov som ger tillräckliga data för omfattande bioinformatisk analys. Protokollet vi presenterar är snabbt och relativt enkelt och kan utföras med standard laboratorieutrustning.
Introduction
Ribosomprofileringstekniken (RIBO-seq) utvecklades i jonathan Weissmans laboratorium vid University of California, San Francisco1. I jämförelse med andra metoder som används för att studera genuttryck på translationell nivå fokuserar RIBO-seq på varje ribosombindning till mRNA och ger information om dess läge och det relativa antalet ribosomer på en transkription. Det möjliggör övervakning av processen för proteinsyntes in vivo och kan ge enkel kodonupplösning och noggrannhet som möjliggör mätning av ribosomtätheten på båda, det enskilda mRNA och längs hela transkriptomen i cellen. På fundamentet av RIBO-seq-tekniken ligger faktumet att under översättning binder ribosomen mRNA-molekylen och skyddar thus det begravda fragmentet av avskriften från en ribonuclease matsmältning. Vid tillsats av ribonukleasen smälts det oskyddade mRNA och fragmenten omslutna av ribosomer – vanligtvis på ~ 28-30 nt långa – förblir intakta. Dessa fragment, så kallade ribosomala fotspår (RF), kan sedan isoleras, sekvenseras och mappas på den transkription de härstammar från vilket resulterar i detektion av ribosomens exakta position. Faktum är att ribosomens förmåga att skydda mRNA-fragment har använts sedan 1960-talet för att studera ribosomala bindnings- och översättningsinitieringsplatser (TIS)2,3,4. Men med utvecklingen inom djupsekvenseringsteknik har RIBO-seq blivit en guldstandard föröversättningsövervakning 5 som genom ribosomengagemanget kan ge en genomomfattande information om proteinsyntes6. Ribosomprofilering fyllde det tekniska gapet som fanns mellan kvantifiering av transkriptomen och proteomen6.
För att genomföra ribosomprofilering måste vi få celllyat av organismen som hade vuxit under de undersökta förhållandena. Att störa dessa förhållanden under cellinsamling och lys kan ge otillförlitliga data. För att förhindra detta används ofta översättningshämmare, snabb skörd och blixtfrysning i flytande kväve. Celler kan lysas genom kryogen slipning i en mekanisk homogenisator som en mixerkvarn7,8 eller en pärlviss9, och genom trituration genom en pipett10 eller med ennål 11. Lysbufferten kan tillsättas strax före eller strax efter pulveriseringen av cellerna. I vårt protokoll använder vi flytande kväve för att förkyla mortel och mortel, samt aluminiumoxid som ett mildare tillvägagångssätt för störningar i bakteriecellväggen, vilket förhindrar RNA-shearing som ofta uppstår när metoder som sonifiering tillämpas. Efter pulverisering lägger vi till en iskall lysbuffert i det kylda innehållet i morteln. Val av lämplig lysbuffert är viktigt för att uppnå bästa upplösning av ribosomala fotavtryck. Eftersom jonisk styrka påverkar både RF-storleken och läsramens precision, rekommenderas det för närvarande att använda lysbuffertar med låg jonisk hållfasthet och buffertkapacitet, även om det verkar som om buffertsammansättningen inte påverkar ribosombeläggningen på mRNAs11,12. Viktiga komponenter i lysbufferten är magnesiumjoner, vars närvaro förhindrar dissociation av ribosomala underenheter och hämmar spontana konformationsförändringar i bakteriella ribosomer11,13. Kalciumjoner spelar också en viktig roll och är viktiga för aktiviteten hos mikrokoccal nukleas (MNase) som används i bakteriell ribosomprofileringsmetod14. Tillsats av guanosin 5′-[β,γ-imido]triphosphate (GMP-PNP), en icke-hydrolyserbar analog av GTP, tillsammans med kloramfenikol hämmar översättning under lysis15.
När lysatet erhålls förtydligas det genom centrifugation och delas in i två portioner, var och en för en RIBO-sekvensq och en hög genomströmning total mRNA-sekvensering (RNA-seq) eftersom de utförs samtidigt (Figur 1). RNA-seq ger en referenspunkt som möjliggör jämförelse av data från både RIBO-seq och RNA-seq under dataanalys. Den undersökta översättningen definieras av normalisering av ribosomal fotspår till mRNA överflöd16. Data från RNA-seq kan också hjälpa till att identifiera kloning eller sekvensering av artefakter17.
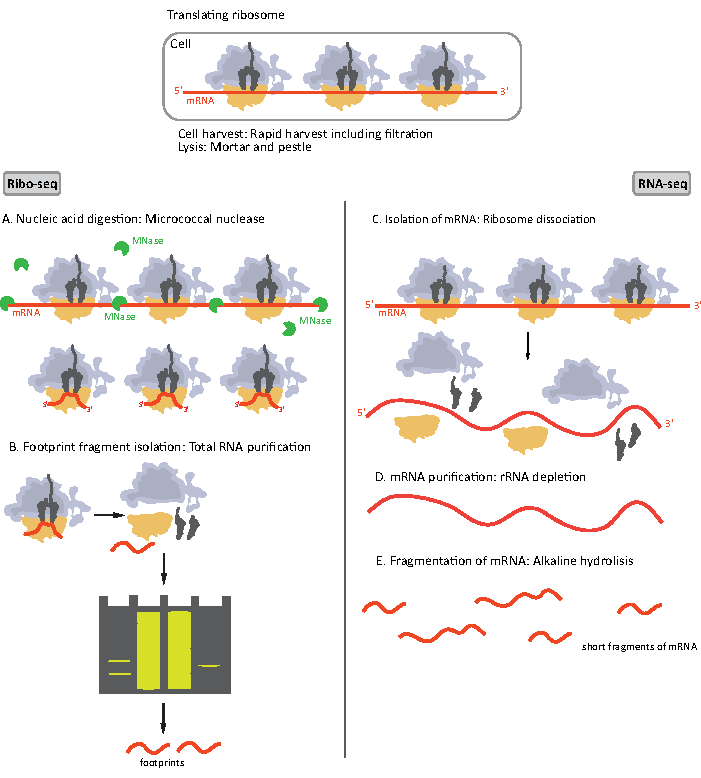
Figur 1. Ritningar av mRNA prov förberedelse för RIBO-seq och RNA-seq. För RIBO-seq biblioteksberedning smälts RNA med MNase (A), följt av storleksvalet av RF på ~ 28-30 nt längd (B); för RNA-seq RNA isoleras (C), utarmas av rRNA (D), och den resulterande mRNA är slumpmässigt fragmenterad i fragment av varierande längder (E). Klicka här om du vill visa en större version av den här figuren.
De första stegen i förfarandet för provberedning för RIBO-seq och RNA-seq skiljer sig något åt (figur 1). För ribosomprofilering måste lysatet smältas av en specifik endonukleas för att bryta ner mRNA-molekylerna som inte skyddas av ribosomerna. I standardprotokoll återvinns de erhållna monosomerna av en sackaroskudde ultracentrifugation eller en sackarosgradient ultracentrifugation8,14. I den här artikeln visar vi att detta steg inte är nödvändigt för att isolera RF som krävs för RIBO-seq i bakterier, likaså för eukaryotaceller 18, och att storleksval av lämpliga längd mRNA-fragment från polyakrylamidgelen är tillräckligt.
För RNA-seq erhålls mRNA genom utarmning av rRNA från den totala RNA – rRNA-molekyler hybridiseras till de biotinylerade oligonukleotidsonderna som binder till de streptavidinbelagda magnetiska pärlorna. RRNA-oligonukleotid-pärlkomplexen avlägsnas sedan från provet med en magnet som resulterar i ett rRNA-utarmat prov19,20. De renade mRNA-molekylerna fragmenteras sedan slumpmässigt av alkalisk hydrolys. De erhållna fragmenten av mRNA samt ribosomala fotavtryck omvandlas till cDNA-bibliotek och förbereds för djup sekvensering (figur 2). Detta innebär slutreparation som behövs efter alkalisk hydrolys (för mRNA) och enzymatisk matsmältning (för RF): defosforylering av 3 ändar följt av fosforylering av 5 ändar. Nästa steg är adapterligantion och omvänd transkription för att skapa cDNA-skär inramade av sekvenser som krävs för nästa generations sekvensering (NGS) med Illumina-plattformen. Den sista fasen av biblioteksberedningen är en PCR-reaktion där konstruktionerna förstärks och märks med exempelspecifika streckkoder för att möjliggöra multiplexering och sekvensering av olika prover på en kanal. Före sekvensering bedöms bibliotekens kvalitet och kvantitet med hög känslighet DNA-elektrofores. cDNA-bibliotek med lämpliga parametrar kan sedan slås samman och sekvenseras. Sekvensering kan utföras på olika Illumina-plattformar, till exempel MiSeq, NextSeq eller HighSeq, beroende på antalet bibliotek, önskad läslängd och sekvenseringsdjup. Efter sekvensering utförs den bioinformatiska analysen.

Figur 2. Förberedelse av bibliotek. Biblioteksförberedelserna omfattar reparation av ändar, adapterligion, omvänd transkription och förstärkning med streckkodning. Klicka här om du vill visa en större version av den här figuren.
Ribosomprofilering är en universell metod som lätt kan modifieras och justeras enligt den vetenskapliga frågan. Ursprungligen användes den i jäst1, men kort efter att den applicerades på bakterieceller21 samt eukaryota modellorganismer inklusive mus10, zebrafisk22, fruktfluga23 och Arabidopsis thaliana24. Det användes också för att studera olika ribosomtyper: cytoplasma, mitokondriell25,26 och kloroplast27,28. I eukaryoter är RIBO-seq ofta anpassad och raffinerad för att undersöka specifika aspekter av översättning, inklusiveinitiering 10,11,29,30,31,32, förlängning1,10,11,31,33, ribosom stalling33 och konformationsändring33. De flesta ändringarna innebär användning av olika översättningshämmare. I bakterier har dock liknande studier varit svåra att genomföra på grund av inhibitorens brist med den erforderliga verkningsmekanismen34. Den vanligaste översättningshämmaren i bakterier är kloramfenikol (CAM) som binder till peptidyltransferascentret (PTC) och förhindrar korrekt positionering av aminoacyl-tRNA på A-sitet. Som ett resultat förhindrar CAM bildandet av en peptidbindning som leder till att de långsträckta ribosomerna35. Andra exempel på översättningshämmare i bakterier är tetracyklin (TET)36, retapamulin (RET)34 och Onc11237 som har använts för att undersöka översättningsinitieringsplatser. TET, som förhindrar tRNA leverans till ribosomen genom att direkt överlappa med antikodon stam-loop av tRNA på A-platsen, tillämpades ursprungligen för att verifiera resultaten från CAM behandling eftersom de båda är antibiotika hämma översättning förlängning38. TET konstaterades för att upptäcka primära TIS, men kunde inte avslöja interna TIS36. RET binder i PTC av bakteriell ribosom, och förhindrar bildandet av den första peptidbindningen genom att störa en elongator aminoacyl-tRNA i A-platsen. Applicering av RET resulterar i ribosomer gripande vid både primära såväl som interna TISs34. Onc112, en proline-rik antimikrobiell peptid, binder i utgångstunneln och blockerar aminoacyl-tRNA-bindning i ribosomal A-platsen. Som ett resultat förhindrar Onc112 initieringskomplex från att komma in i förlängningsfasen37.
Den huvudsakliga informationen ribosom profilering ger är ribosom densitet och deras position på mRNA. Det tillämpades framgångsrikt för att undersöka differentiella genuttryck på översättningsnivå under olikatillväxtförhållanden 1,6, mäta translationelleffektivitet 1,38,39 och upptäcka översättningsregleringshändelser som ribosomalpausning 10. RIBO-seq gör det också möjligt att avslöja översättningen av kommenterade ncRNA, pseudogenes och unannotated små öppna läsramar (ORF) som leder till identifiering av nya och / eller mycket korta proteinkodningsgener10,12,22,30,37. I sådana fall kan RIBO-seq finjustera och förbättra genomanteckningen. Med sin höga känslighet för identifiering av översatta ORF och dess kvantitativa karaktär kan ribosomprofilering också fungera som en proxy för proteomebestämningen eller för att hjälpa proteomikstudier31,34,39. Genom att kartlägga TIS avslöjar ribosomprofilering N-terminalt utsträckta och trunkerade isoformer av kändaproteiner 10,32. RIBO-seq anpassades också för att studera samöversättningsvikning avproteiner 14,21,24. Denna metod möjliggör mätning av förlängningshastigheter1,10,39 eller avkodningshastigheter för enskilda kodoner6 och hjälper till att utveckla kvantitativa modeller av översättning17. Ribosomprofileringsmetoden kan också ge mekanistiska insikter om ribosomen som pausar ibakterierna 7,15,17, frameshifting40, stop-codon readthrough21, terminering / återvinningdefekter 41,42 och ribosomal konformation ändras33 i eukaryoter. RIBO-seq anpassades också för att undersöka effekten av specifika transverkande faktorer på översättning såsom miRNAs6 och RNA-bindande proteiner i eukaryoter16,43. Det är dock viktigt att erkänna att den experimentella utformningen och den erhållna upplösningen av RIBO-seq bestämmer mängden information som kan extraheras från de resulterande sekvenseringsdata12.
Protocol
Representative Results
Discussion
Den viktigaste tekniska utmaningen med ribosomprofilering är behovet av att snabbt hämma översättningen för att fånga en ögonblicksbild av ribosomer på mRNAs vid ett visst fysiologiskt tillstånd av intresse. För att åstadkomma detta används ofta översättningshämmare, snabb skörd och blixtfrysning i flytande kväve. Att applicera antibiotika är valfritt eftersom de kan orsaka artefakter. Kloramfenikol är ett vanligt läkemedel för att stoppa långsträckta ribosomer i bakteriell RIBO-seq. Det förhindra…
Disclosures
The authors have nothing to disclose.
Acknowledgements
ALS vill bekräfta det ekonomiska stödet från EMBO Installationsbidrag IG 3914 och POIR. 04.04.00-00-3E9C/17-00 inom ramen för fonden för polsk vetenskaps första gruppprogram som medfinansieras av Europeiska unionen inom ramen för Europeiska regionala utvecklingsfonden.
Materials
| 10X TBE (powder) | Invitrogen | AM9864 | |
| 2-Mercaptoethanol, 99%, pure | Acros Organics | 125472500 | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| Aluminium oxide calcinated pure p.a. | Chempur | 114560600 | |
| Calcium chloride dihydrate | Sigma-Aldrich | C3881-500G | |
| Chloramphenicol | MP Biomedicals | 190321 | |
| DNA Clean & Concentrator -5 | Zymo Research | D4004 | |
| Dnase I recombinant, Rnase-free | Roche | 4716728001 | |
| EDTA disodium salt | Fisher Scientific | E/P140/48 | |
| Ethyl Alcohol Absolut 99,8% Pure-P.A.-Basic | POCH Avantor Performance Materials Poland S.A | BA6480111 | |
| Filtration apparatus | VWR Collection | 511-0265 | all-glass filtration apparatus, with funnel, fritted base, cap, 47 mm Ø spring clamp and ground joint flask |
| Gel 40 (19:1) | Rotiphorese | 3030.1 | |
| Gel Loading Dye, Blue, 6X | New England Biolabs | E6138G | |
| Guanosine 5′-[β,γ-imido]triphosphate trisodium salt hydrate | Sigma-Aldrich | G0635-25MG | |
| labZAP | A&A Biotechnology | 040-500 | |
| Magnesium acetate tetrahydrate | Sigma-Aldrich | M5661-250G | |
| MCE membrane fiter | Alfatec Technology | M47MCE45GWS | pore size: 0.45um |
| MICROBExpress Bacterial mRNA Purification | Invitrogen | AM1905 | |
| Multiplex Small RNA Library Prep Set for Illumina | New England Biolabs | E7300S | |
| Nuclease-Free Water | Ambion | AM9937 | |
| Potassium Acetate Anhydrous Pure P.A. | POCH Avantor Performance Materials Poland S.A | 744330113 | |
| Quick-Load pBR322 DNA-MspI Digest | New England Biolabs | E7323A | |
| RNA Clean & Concentrator -25 | Zymo Research | R1018 | |
| Sodium acetate | Sigma-Aldrich | S2889-250G | |
| Sodium carbonate | Sigma-Aldrich | 223530-500G | |
| Sodium hydrogen carbonate pure p.a. | POCH Avantor Performance Materials Poland S.A | 810530115 | |
| SYBR Gold nucleic acid gel stain | Life Technologies | S11494 | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| TBE-Urea Sample Buffer (2x) | Invitrogen | LC6876 | |
| Tris(hydroxymethyl)amino-methane, ultrapure, 99,9% | AlfaAesar | J65594 | |
| Triton X-100, 98% | Acros Organics | 327371000 | |
| Urea G.R. | lach:ner | 40096-AP0 |
References
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Takanami, M., Yan, Y., Jukes, T. H. Studies on the site of ribosomal binding of f2 bacteriophage RNA. Journal of Molecular Biology. 12 (3), 761-773 (1965).
- Steitz, J. A. Polypeptide Chain Initiation: Nucleotide Sequences of the Three Ribosomal Binding Sites in Bacteriophage R17 RNA. Nature. 224 (5223), 957-964 (1969).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. The EMBO journal. 7 (11), 3559-3569 (1988).
- Argüello, R. J., et al. SunRiSE – measuring translation elongation at single-cell resolution by means of flow cytometry. Journal of Cell Science. 131 (10), 214346 (2018).
- Michel, A. M., Baranov, P. V. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale. Wiley Interdisciplinary Reviews: RNA. 4 (5), 473-490 (2013).
- Woolstenhulme, C. J., Guydosh, N. R., Green, R., Buskirk, A. R. High-precision analysis of translational pausing by ribosome profiling in bacteria lacking EFP. Cell reports. 11 (1), 13-21 (2015).
- McGlincy, N. J., Ingolia, N. T. Transcriptome-wide measurement of translation by ribosome profiling. Methods (San Diego, Calif). 126, 112-129 (2017).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic acids research. 42 (17), 134 (2014).
- Ingolia, N. T., Lareau, L. F., Weissman, J. S. Ribosome Profiling of Mouse Embryonic Stem Cells Reveals the Complexity of Mammalian Proteomes. Cell. 147 (4), 789-802 (2011).
- Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature protocols. 7 (8), 1534-1550 (2012).
- Hsu, P. Y., et al. Super-resolution ribosome profiling reveals unannotated translation events in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 113 (45), 7126-7135 (2016).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proceedings of the National Academy of Sciences of the United States of America. 101 (35), 12893-12898 (2004).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature protocols. 8 (11), 2212-2239 (2013).
- Li, G. W., Oh, E., Weissman, J. S. The anti-Shine-Dalgarno sequence drives translational pausing and codon choice in bacteria. Nature. 484 (7395), 538-541 (2012).
- King, H. A., Gerber, A. P. Translatome profiling: methods for genome-scale analysis of mRNA translation. Briefings in functional genomics. 15 (1), 22-31 (2014).
- Mohammad, F., Woolstenhulme, C. J., Green, R., Buskirk, A. R. Clarifying the Translational Pausing Landscape in Bacteria by Ribosome Profiling. Cell reports. 14 (4), 686-694 (2016).
- Reid, D. W., Shenolikar, S., Nicchitta, C. V. Simple and inexpensive ribosome profiling analysis of mRNA translation. Methods (San Diego, Calif). 91, 69-74 (2015).
- Sanz, E., Yang, L., Su, T., Morris, D. R., McKnight, G. S., Amieux, P. S. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13939-13944 (2009).
- Petrova, O. E., Garcia-Alcalde, F., Zampaloni, C., Sauer, K. Comparative evaluation of rRNA depletion procedures for the improved analysis of bacterialbiofilm and mixed pathogen culture transcriptomes. Scientific Reports. 7, 41114 (2017).
- Oh, E. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Chew, G. L. Ribosome profiling reveals resemblance between long non-coding RNAs and 5′ leaders of coding RNAs. Development. 140 (13), 2828-2834 (2013).
- Dunn, J. G., Foo, C. K., Belletier, N. G., Gavis, E. R., Weissman, J. S. Ribosome profiling reveals pervasive and regulated stop codon readthrough in Drosophila melanogaster. eLife. 2, 01179 (2013).
- Liu, M. J., et al. Translational landscape of photomorphogenic Arabidopsis. The Plant cell. 25 (10), 3699-3710 (2013).
- Rooijers, K., Loayza-Puch, F., Nijtmans, L. G., Agami, R. Ribosome profiling reveals features of normal and disease-associated mitochondrial translation. Nature Communications. 4, 2886 (2013).
- Danielle, L., et al. Fidelity of translation initiation is required for coordinated respiratory complex assembly. Science Advances. 5 (12), 2118 (2019).
- Zoschke, R., Watkins, K. P., Barkan, A. A rapid ribosome profiling method elucidates chloroplast ribosome behavior in vivo. The Plant cell. 25 (6), 2265-2275 (2013).
- Chotewutmontri, P., Barkan, A. Dynamics of chloroplast translation during chloroplast differentiation in maize. PLoS genetics. 12 (7), 1006106 (2016).
- Lee, S., Liu, B., Huang, S. X., Shen, B., Qian, S. B. Global mapping of translation initiation sites in mammalian cells at single-nucleotide resolution. Proceedings of the National Academy of Sciences of the United States of America. 109 (37), 2424-2432 (2012).
- Martinez, T. F., Chu, Q., Donaldson, C., Tan, D., Shokhirev, M. N., Saghatelian, A. Accurate annotation of human protein-coding small open reading frames. Nature chemical biology. 16 (4), 458-468 (2020).
- Simsek, D., et al. The Mammalian Ribo-interactome Reveals Ribosome Functional Diversity and Heterogeneity. Cell. 169 (6), 1051-1065 (2017).
- Fritsch, C., et al. Genome-wide search for novel human uORFs and N-terminal protein extensions using ribosomal footprinting. Genome research. 22 (11), 2208-2218 (2012).
- Lareau, L. F., Hite, D. H., Hogan, G. J., Brown, P. O. Distinct stages of the translation elongation cycle revealed by sequencing ribosome-protected mRNA fragments. eLife. 3, 01257 (2014).
- Meydan, S., et al. Retapamulin-Assisted Ribosome Profiling Reveals the Alternative Bacterial Proteome. Molecular Cell. 74 (3), 481-493 (2019).
- Wilson, D. N. The A-Z of bacterial translation inhibitors. Critical Reviews in Biochemistry and Molecular Biology. 44, 393-433 (2009).
- Nakahigashi, K. Comprehensive identification of translation start sites by tetracycline-inhibited ribosome profiling. DNA research : an international journal for rapid publication of reports on genes and genomes. 23 (3), 193-201 (2016).
- Weaver, J., Mohammad, F., Buskirk, A. R., Storz, G. Identifying Small Proteins by Ribosome Profiling with Stalled Initiation Complexes. mBio. 10 (2), 02819 (2019).
- Nakahigashi, K. Effect of codon adaptation on codon-level and gene-level translation efficiency in vivo. BMC genomics. 15 (1), 1115 (2014).
- Li, G. W., Burkhardt, D., Gross, C., Weissman, J. S. Quantifying absolute protein synthesis rates reveals principles underlying allocation of cellular resources. Cell. 157 (3), 624-635 (2014).
- Michel, A. M., et al. Observation of dually decoded regions of the human genome using ribosome profiling data. Genome research. 22 (11), 2219-2229 (2012).
- Guydosh, N. R., Green, R. Dom34 rescues ribosomes in 3′ untranslated regions. Cell. 156 (5), 950-962 (2014).
- Young, D. J., Guydosh, N. R., Zhang, F., Hinnebusch, A. G., Green, R. Rli1/ABCE1 Recycles Terminating Ribosomes and Controls Translation Reinitiation in 3’UTRs In Vivo. Cell. 162 (4), 872-884 (2015).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Dobin, A., et al. ultrafast universal RNA seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- . . Babraham Bioinformatics. Fast QC. , (2021).
- Gerashchenko, M. V., Gladyshev, V. N. Ribonuclease selection for ribosome profiling. Nucleic acids research. 45 (2), 6 (2017).
- Verbruggen, S., Menschaert, G. mQC: A post-mapping data exploration tool for ribosome profiling. Computer Methods and Programs in Biomedicine. 181, 104806 (2019).
- Cui, H., Hu, H., Zeng, J., Chen, T. DeepShape: estimating isoform-level ribosome abundance and distribution with Ribo-seq data. BMC bioinformatics. 20, 678 (2019).
- Choi, J. . RiboToolkit: an integrated platform for analysis and annotation of ribosome profiling data to decode RNA translation at codon resolution. , (2021).
- Choi, J. Dynamics of the context-specific translation arrest by chloramphenicol and linezolid. Nature chemical biology. 16 (3), 310-317 (2020).
- Tompson, J., O’Connor, M., Mills, J. A., Dahlberg, A. E. The protein synthesis inhibitors, oxazolidinones and chloramphenicol, cause extensive translational inaccuracy in vivo. Journal of Molecular Biology. 322 (2), 273-279 (2002).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature reviews. Molecular cell biology. 16 (11), 651-664 (2015).
- Glaub, A., Huptas, C., Neuhaus, K., Ardern, Z. Recommendations for bacterial ribosome profiling experiments based on bioinformatic evaluation of published data. Journal of Biological Chemistry. 295, 8999-9011 (2020).
- Sorour, M. H., Hani, H. A., Shaalan, H. F., El-Sayed, M. M. H. Experimental screening of some chelating agents for calcium and magnesium removal from saline solutions. Desalination and Water Treatment. 57 (48-49), 22799-22808 (2015).
- Mohammad, F., Green, R., Buskirk, A. R. A systematically-revised ribosome profiling method for bacteria reveals pauses at single-codon resolution. Elife. 8, 42591 (2019).
- O’Connor, P. B., Li, G. W., Weissman, J. S., Atkins, J. F., Baranov, P. V. rRNA:mRNA pairing alters the length and the symmetry of mRNA-protected fragments in ribosome profiling experiments. Bioinformatics. 29 (12), 1488-1491 (2013).
- Protocol for RNA Clean & Concentrator -25. Zymo Research Available from: https://files.zymoresearch.com/protocols/_r1017_r1018_rna_clean_concentrator-25.pdf (2021)
- Protocol for use with NEBNext Small RNA Library Prep Set for Illumina (E7300, E7580, E7560, E7330). New England Biolabs Available from: https://www.international.neb.com/protocols/2018/03/27/protocol-for-use-with-nebnext-small-rna-library-prep-set-for-illumina-e7300-e7580-e7560-e7330 (2021)

