RIBO-seq in Bacteria: un protocollo di raccolta dei campioni e preparazione della biblioteca per il sequenziamento NGS
Summary
Qui descriviamo le fasi di raccolta e preparazione dei campioni per RIBO-seq nei batteri. Il sequenziamento delle biblioteche elaborate secondo queste linee guida si traduce in dati sufficienti per un’analisi bioinformatica completa. Il protocollo che presentiamo è semplice, utilizza attrezzature di laboratorio standard e richiede sette giorni dalla lisi all’ottenimento delle biblioteche.
Abstract
La tecnica di profilazione ribosoma (RIBO-seq) è attualmente lo strumento più efficace per studiare il processo di sintesi proteica in vivo. Il vantaggio di questo metodo, rispetto ad altri approcci, è la sua capacità di monitorare la traduzione mappando con precisione la posizione e il numero di ribosomi su una trascrizione di mRNA.
In questo articolo, descriviamo le fasi consecutive della raccolta e della preparazione dei campioni per il metodo RIBO-seq nei batteri, evidenziando i dettagli rilevanti per la pianificazione e l’esecuzione dell’esperimento.
Poiché il RIBO-seq si basa su ribosomi intatti e mRNA correlati, il passo chiave è la rapida inibizione della traduzione e un’adeguata disintegrazione delle cellule. Pertanto, suggeriamo la filtrazione e il congelamento flash nell’azoto liquido per la raccolta cellulare con un pretrattamento opzionale con cloramfenicolo per arrestare la traduzione nei batteri. Per la disintegrazione, proponiamo la macinazione di cellule congelate con malta e pestello in presenza di ossido di alluminio per interrompere meccanicamente la parete cellulare. In questo protocollo non è richiesto un cuscino di saccarosio o un ultracentrifugazione gradiente di saccarosio per la purificazione monosomatica. Invece, viene applicata la separazione dell’mRNA utilizzando l’elettroforesi del gel di poliacrilammide (PAGE) seguita dall’escissione dell’impronta ribosomiale (banda 28-30 nt) e fornisce risultati soddisfacenti. Ciò semplifica in gran parte il metodo e riduce i tempi e i requisiti delle apparecchiature per la procedura. Per la preparazione della biblioteca, si consiglia di utilizzare il piccolo kit di RNA disponibile in commercio per il sequenziamento Illumina da New England Biolabs, seguendo le linee guida del produttore con un certo grado di ottimizzazione.
Le librerie cDNA risultanti presentano la quantità e la qualità appropriate necessarie per il sequenziamento di nuova generazione (NGS). Il sequenziamento delle librerie preparate secondo il protocollo descritto comporta da 2 a 10 mln letture mappate in modo univoco per campione fornendo dati sufficienti per un’analisi bioinformatica completa. Il protocollo che presentiamo è rapido e relativamente semplice e può essere eseguito con apparecchiature di laboratorio standard.
Introduction
La tecnica di profilazione ribosoma (RIBO-seq) è stata sviluppata nel laboratorio di Jonathan Weissman presso l’Università della California, San Francisco1. Rispetto ad altri metodi usati per studiare l’espressione genica a livello traslazionale, RIBO-seq si concentra su ogni legame ribosoma all’mRNA e fornisce informazioni sulla sua posizione e sul numero relativo di ribosomi su una trascrizione. Consente di monitorare il processo di sintesi proteica in vivo e può fornire una risoluzione e una precisione del codone singolo consentendo la misurazione della densità ribosoma su entrambi, il singolo mRNA e lungo l’intero trascrittame nella cellula. Alla base della tecnica RIBO-seq si trova il fatto che durante la traduzione il ribosoma lega la molecola di mRNA e quindi protegge il frammento sepolto della trascrizione da una digestione della ribonucleasi. Dopo l’aggiunta della ribonucleasi, l’mRNA non protetto viene digerito e i frammenti racchiusi da ribosomi – tipicamente lunghi ~28-30 nt – rimangono intatti. Questi frammenti, chiamati impronte ribosomiali (RF), possono quindi essere isolati, sequenziati e mappati sulla trascrizione da cui hanno avuto origine con conseguente rilevamento della posizione esatta dei ribosomi. Infatti, la capacità ribosoma di proteggere i frammenti di mRNA è stata utilizzata dagli anni ’60 per studiare i siti di rilegatura ribosomiale e iniziazione alla traduzione (TIS)2,3,4. Tuttavia, con l’avanzamento della tecnologia di sequenziamento profondo, RIBO-seq è diventato un gold standard per il monitoraggio dellatraduzione 5 che, attraverso l’impegno ribosoma, può fornire informazioni a livello genomico sulla sintesi proteica6. La profilazione ribosoma ha colmato il divario tecnologico esistente tra la quantificazione del trascrittame e il proteoma6.
Per condurre la profilazione ribosoma è necessario ottenere il lisato cellulare dell’organismo che era cresciuto nelle condizioni studiate. Interrompere queste condizioni durante la raccolta e lalisi cellulare può fornire dati inaffidabili. Per evitare ciò, vengono comunemente utilizzati inibitori della traduzione, raccolta rapida e congelamento lampo nell’azoto liquido. Le cellule possono essere lisciviate mediante macinazione criogenica in un omogeneizzatore meccanico come un miscelatore7,8 o un battitore diperline 9e per triturazione attraverso una pipetta10 o con un ago11. Il tampone di lysis può essere aggiunto poco prima o poco dopo la polverizzazione delle cellule. Nel nostro protocollo utilizziamo azoto liquido per preraffreddare malta e pestello, così come l’ossido di alluminio come approccio più delicato all’interruzione della parete cellulare batterica, che impedisce la tosatura dell’RNA spesso riscontrata quando vengono applicati metodi come la sonificazione. Dopo la polverizzazione, aggiungiamo un tampone di lisi ghiacciata nel contenuto raffreddato della malta. La selezione di un buffer di lisi appropriato è importante per ottenere la migliore risoluzione delle impronte ribosomiali. Poiché la forza ionica influisce sia sulla dimensione RF che sulla precisione del fotogramma di lettura, si consiglia attualmente di utilizzare buffer di lisi con bassa forza ionica e capacità tampone, anche se sembra che la composizione tampone non influenzi l’occupazione ribosomiale su mRNA11,12. Componenti importanti del tampone di lisi sono gli ioni di magnesio, la cui presenza previene la dissociazione delle subunità ribosomiali e inibisce i cambiamenti conformazionali spontanei nei ribosomibatterici 11,13. Anche gli ioni di calcio svolgono un ruolo significativo e sono essenziali per l’attività della nucleasi micrococcica (MNasi) utilizzata nel metodo di profilazione ribosomabatterica 14. L’aggiunta di guanosina 5′-[β,γ-imido]trifosfato (GMP-PNP), un analogo non idrolabile di GTP, insieme al cloramfenicolo inibisce la traduzione durante la lisi15.
Quando il lisato viene ottenuto, viene chiarito per centrifugazione e diviso in due porzioni, ognuna per un RIBO-seq e un sequenziamento totale dell’mRNA ad alta produttività (RNA-seq) poiché vengono eseguite simultaneamente(Figura 1). RNA-seq fornisce un punto di riferimento che consente il confronto dei dati sia ribo-seq che RNA-seq durante l’analisi dei dati. Il translatoma studiato è definito dalla normalizzazione delle impronte ribosomiche all’abbondanza di mRNA16. I dati di RNA-seq possono anche aiutare a identificare la clonazione o sequenziare gliartefatti 17.
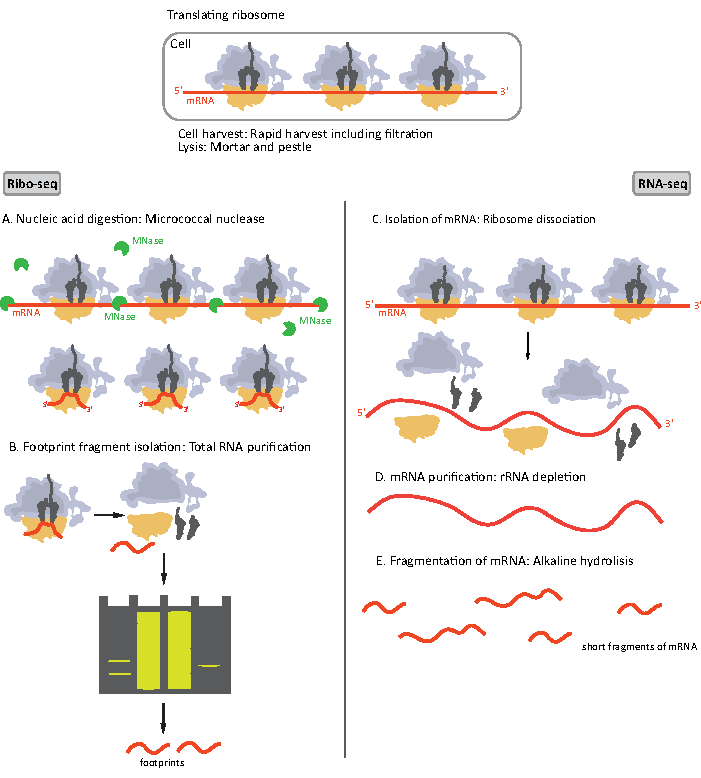
Figura 1. Schemi di preparazione del campione di mRNA per RIBO-seq e RNA-seq. Per la preparazione della libreria RIBO-seq, l’RNA viene digerito con MNasi (A), seguito dalla selezione delle dimensioni di RF di ~28-30 nt di lunghezza (B); per l’RNA RNA-seq è isolato (C), impoverito di rRNA (D), e l’mRNA risultante è frammentato casualmente in frammenti di varie lunghezze (E). Clicca qui per visualizzare una versione più grande di questa figura.
I primi passi della procedura di preparazione del campione per RIBO-seq e RNA-seq differiscono leggermente (Figura 1). Per la profilazione ribosomiale, il lisato deve essere digerito da una specifica endonucleasi per degradare le molecole di mRNA non protette dai ribosomi. Nei protocolli standard, i monosomi ottenuti vengono recuperati da un cuscino di saccarosio ultracentrifugato o da un gradiente di saccarosio ultracentrifugazione8,14. In questo articolo, mostriamo che questo passaggio non è necessario per isolare RF richiesto per il RIBO-seq nei batteri, allo stesso modo per le cellule eucariotiche18e che la selezione delle dimensioni dei frammenti di mRNA di lunghezza appropriata dal gel di poliacrilammide è sufficiente.
Per l’RNA-seq, l’mRNA è ottenuto dall’esaurimento dell’rRNA dalle molecole totali di RNA – rRNA che si ibridano alle sonde oligonucleotidi biotinylate che si legano alle perline magnetiche rivestite di streptavidina. I complessi rRNA-oligonucleotide-perline vengono quindi rimossi dal campione con un magnete con conseguente campionamento impoverito di rRNA19,20. Le molecole di mRNA purificate vengono quindi frammentate casualmente per idrolisi alcalina. I frammenti ottenuti di mRNA e le impronte ribosomiali vengono convertiti in librerie cDNA e preparati per il sequenziamento profondo (Figura 2). Ciò comporta la riparazione delle estremità necessaria dopo l’idrolisi alcalina (per mRNA) e la digestione enzimatica (per RF): defosforilazione di 3′ estremità seguita da fosforilazione di 5′ estremità. I passaggi successivi sono la legatura degli adattatori e la trascrizione inversa per creare inserti cDNA incorniciati da sequenze necessarie per il sequenziamento di nuova generazione (NGS) utilizzando la piattaforma Illumina. L’ultima fase della preparazione della libreria è una reazione PCR in cui i costrutti vengono amplificati ed etichettati con codici a barre specifici del campione per consentire il multiplexing e il sequenziamento di vari campioni su un canale. Prima del sequenziamento, la qualità e la quantità delle librerie sono valutate dall’elettroforesi su chip del DNA ad alta sensibilità. Le librerie cDNA con parametri appropriati possono quindi essere raggruppate e sequenziate. Il sequenziamento può essere eseguito su diverse piattaforme Illumina, come MiSeq, NextSeq o HighSeq, a seconda del numero di librerie, della lunghezza di lettura e della profondità di sequenziamento richieste. Dopo il sequenziamento, viene eseguita l’analisi bioinformatica.

Figura 2. Preparazione della biblioteca. La preparazione della libreria include la riparazione delle estremità, la legatura degli adattatori, la trascrizione inversa e l’amplificazione con codifica a barre. Clicca qui per visualizzare una versione più grande di questa figura.
La profilazione ribosoma è un metodo universale che può essere facilmente modificato e regolato in base alla questione scientifica. Originariamente è stato utilizzato nellievito 1,ma poco dopo è stato applicato alle cellulebatteriche 21 così come agli organismi modello eucariotico tra cui topo10,zebrafish22,mosca dellafrutta 23 e Arabidopsis thaliana24. È stato anche usato per studiare diversi tipi di ribosomi: citoplasmatico, mitocondriale25,26 e cloroplasto27,28. Negli eucarioti RIBO-seq è comunemente adattato e raffinato per indagare aspetti specifici della traduzione, tra cuiiniziazione 10,11,29,30,31,32,allungamento 1,10,11,31,33,ribosoma stallo33 e modifica conformazione33. La maggior parte delle modifiche comporta l’uso di diversi inibitori della traduzione. Nei batteri, tuttavia, studi analoghi sono stati difficili da condurre a causa della scarsità di inibitori con il meccanismo d’azionerichiesto 34. L’inibitore della traduzione più comunemente usato nei batteri è il cloramfenicolo (CAM) che si lega al centro peptidil transferasi (PTC) e impedisce il corretto posizionamento dell’amminoacil-tRNA nel sito A. Di conseguenza, CAM impedisce la formazione di un legame peptidico che porta ad arrestare i ribosomi allunganti35. Altri esempi di inibitori della traduzione nei batteri sono la tetraciclina (TET)36, la retapamulina (RET)34 e l’Onc11237 che sono stati utilizzati per studiare i siti di iniziazione alla traduzione. TET, che impedisce la consegna di tRNA al ribosoma sovrapponendosi direttamente con l’anello staminali anticodonte del tRNA nel sito A, è stato originariamente applicato per verificare i risultati ottenuti dal trattamento CAM poiché sono entrambi antibiotici che inibiscono l’allungamento dellatraslazione 38. Tet è stato trovato per rilevare tis primario, tuttavia non è stato in grado di rivelare tis36 interno. Il RET si lega nel PTC del ribosoma batterico e previene la formazione del primo legame peptidico interferendo con un aminoacil-tRNA allungatore nel sito A. L’applicazione del RET comporta l’arresto dei ribosomi sia al TISs34primario che a quello interno. Onc112, un peptide antimicrobico ricco di prolina, si lega nel tunnel di uscita e blocca il legame amminoacil-tRNA nel sito ribosomiale A. Di conseguenza, Onc112 impedisce ai complessi di iniziazione di entrare nella fase diallungamento 37.
La principale profilazione ribosoma delle informazioni fornisce la densità dei ribosomi e la loro posizione sull’mRNA. È stato applicato con successo per studiare l’espressione genica differenziale a livello di traduzione in varie condizioni dicrescita 1,6,misurare l’efficienza traslazionale1,38,39 e rilevare eventi di regolazione della traduzione come la pausa ribosomiale10. RIBO-seq consente inoltre di scoprire la traduzione di ncRNA annotato, pseudogeni e piccoli telai di lettura aperti senza preavviso (ORF) che portano all’identificazione di geni di codifica proteica nuovi e/o molto brevi10,12,22,30,37. In questi casi, RIBO-seq può perfezionare e migliorare l’annotazione del genoma. Con la sua elevata sensibilità per l’identificazione degli ORF tradotti e la sua natura quantitativa, la profilazione ribosoma può anche servire da proxy per la determinazione del proteoma o per aiutare gli studi di proteomica31,34,39. Mappando TIS, la profilazione ribosoma rivela isoforme estese e troncate N-terminale di proteinenote 10,32. RIBO-seq è stato anche adattato per studiare il ripiegamento co-traslazionaledelle proteine 14,21,24. Questo metodo consente di misurare i tassi diallungamento 1,10,39 o velocità di decodifica dei singolicodoni 6 e aiuta a sviluppare modelli quantitativi ditraduzione 17. Il metodo di profilazione ribosoma è anche in grado di fornire approfondimenti meccanicistici sulla pausa ribosoma neibatteri 7,15,17,frameshifting40,stop-codon readthrough21,difetti di terminazione /riciclaggio41,42 e modifiche alla conformazione ribosomiale33 negli eucarioti. RIBO-seq è stato anche adattato per esaminare l’impatto di specifici fattori trans-acting sulla traduzione come le miRNA6 e le proteine leganti l’RNA negli eucarioti16,43. Tuttavia, è importante riconoscere che il progetto sperimentale e la risoluzione ottenuta di RIBO-seq determinano la quantità di informazioni che possono essere estratte dai dati di sequenziamentorisultanti 12.
Protocol
Representative Results
Discussion
La sfida tecnica chiave della profilazione ribosoma è la necessità di inibire rapidamente la traduzione al fine di catturare un’istantanea dei ribosomi sugli mRNA in un particolare stato fisiologico di interesse. A tal fine, vengono comunemente utilizzati inibitori della traduzione, raccolta rapida e congelamento flash nell’azoto liquido. L’applicazione di antibiotici è facoltativa poiché possono causare artefatti. Il cloramfenicolo è un farmaco comunemente usato per arrestare i ribosomi allunganti nel ribo-seq batt…
Disclosures
The authors have nothing to disclose.
Acknowledgements
ALS desidera riconoscere il sostegno finanziario delle sovvenzioni per l’installazione EMBO IG 3914 e POIR. 04.04.00-00-3E9C/17-00 realizzato nell’ambito del programma First TEAM della Fondazione per la scienza polacca cofinanziato dall’Unione europea nell’ambito del Fondo europeo di sviluppo regionale.
Materials
| 10X TBE (powder) | Invitrogen | AM9864 | |
| 2-Mercaptoethanol, 99%, pure | Acros Organics | 125472500 | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| Aluminium oxide calcinated pure p.a. | Chempur | 114560600 | |
| Calcium chloride dihydrate | Sigma-Aldrich | C3881-500G | |
| Chloramphenicol | MP Biomedicals | 190321 | |
| DNA Clean & Concentrator -5 | Zymo Research | D4004 | |
| Dnase I recombinant, Rnase-free | Roche | 4716728001 | |
| EDTA disodium salt | Fisher Scientific | E/P140/48 | |
| Ethyl Alcohol Absolut 99,8% Pure-P.A.-Basic | POCH Avantor Performance Materials Poland S.A | BA6480111 | |
| Filtration apparatus | VWR Collection | 511-0265 | all-glass filtration apparatus, with funnel, fritted base, cap, 47 mm Ø spring clamp and ground joint flask |
| Gel 40 (19:1) | Rotiphorese | 3030.1 | |
| Gel Loading Dye, Blue, 6X | New England Biolabs | E6138G | |
| Guanosine 5′-[β,γ-imido]triphosphate trisodium salt hydrate | Sigma-Aldrich | G0635-25MG | |
| labZAP | A&A Biotechnology | 040-500 | |
| Magnesium acetate tetrahydrate | Sigma-Aldrich | M5661-250G | |
| MCE membrane fiter | Alfatec Technology | M47MCE45GWS | pore size: 0.45um |
| MICROBExpress Bacterial mRNA Purification | Invitrogen | AM1905 | |
| Multiplex Small RNA Library Prep Set for Illumina | New England Biolabs | E7300S | |
| Nuclease-Free Water | Ambion | AM9937 | |
| Potassium Acetate Anhydrous Pure P.A. | POCH Avantor Performance Materials Poland S.A | 744330113 | |
| Quick-Load pBR322 DNA-MspI Digest | New England Biolabs | E7323A | |
| RNA Clean & Concentrator -25 | Zymo Research | R1018 | |
| Sodium acetate | Sigma-Aldrich | S2889-250G | |
| Sodium carbonate | Sigma-Aldrich | 223530-500G | |
| Sodium hydrogen carbonate pure p.a. | POCH Avantor Performance Materials Poland S.A | 810530115 | |
| SYBR Gold nucleic acid gel stain | Life Technologies | S11494 | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| TBE-Urea Sample Buffer (2x) | Invitrogen | LC6876 | |
| Tris(hydroxymethyl)amino-methane, ultrapure, 99,9% | AlfaAesar | J65594 | |
| Triton X-100, 98% | Acros Organics | 327371000 | |
| Urea G.R. | lach:ner | 40096-AP0 |
References
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Takanami, M., Yan, Y., Jukes, T. H. Studies on the site of ribosomal binding of f2 bacteriophage RNA. Journal of Molecular Biology. 12 (3), 761-773 (1965).
- Steitz, J. A. Polypeptide Chain Initiation: Nucleotide Sequences of the Three Ribosomal Binding Sites in Bacteriophage R17 RNA. Nature. 224 (5223), 957-964 (1969).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. The EMBO journal. 7 (11), 3559-3569 (1988).
- Argüello, R. J., et al. SunRiSE – measuring translation elongation at single-cell resolution by means of flow cytometry. Journal of Cell Science. 131 (10), 214346 (2018).
- Michel, A. M., Baranov, P. V. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale. Wiley Interdisciplinary Reviews: RNA. 4 (5), 473-490 (2013).
- Woolstenhulme, C. J., Guydosh, N. R., Green, R., Buskirk, A. R. High-precision analysis of translational pausing by ribosome profiling in bacteria lacking EFP. Cell reports. 11 (1), 13-21 (2015).
- McGlincy, N. J., Ingolia, N. T. Transcriptome-wide measurement of translation by ribosome profiling. Methods (San Diego, Calif). 126, 112-129 (2017).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic acids research. 42 (17), 134 (2014).
- Ingolia, N. T., Lareau, L. F., Weissman, J. S. Ribosome Profiling of Mouse Embryonic Stem Cells Reveals the Complexity of Mammalian Proteomes. Cell. 147 (4), 789-802 (2011).
- Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature protocols. 7 (8), 1534-1550 (2012).
- Hsu, P. Y., et al. Super-resolution ribosome profiling reveals unannotated translation events in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 113 (45), 7126-7135 (2016).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proceedings of the National Academy of Sciences of the United States of America. 101 (35), 12893-12898 (2004).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature protocols. 8 (11), 2212-2239 (2013).
- Li, G. W., Oh, E., Weissman, J. S. The anti-Shine-Dalgarno sequence drives translational pausing and codon choice in bacteria. Nature. 484 (7395), 538-541 (2012).
- King, H. A., Gerber, A. P. Translatome profiling: methods for genome-scale analysis of mRNA translation. Briefings in functional genomics. 15 (1), 22-31 (2014).
- Mohammad, F., Woolstenhulme, C. J., Green, R., Buskirk, A. R. Clarifying the Translational Pausing Landscape in Bacteria by Ribosome Profiling. Cell reports. 14 (4), 686-694 (2016).
- Reid, D. W., Shenolikar, S., Nicchitta, C. V. Simple and inexpensive ribosome profiling analysis of mRNA translation. Methods (San Diego, Calif). 91, 69-74 (2015).
- Sanz, E., Yang, L., Su, T., Morris, D. R., McKnight, G. S., Amieux, P. S. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13939-13944 (2009).
- Petrova, O. E., Garcia-Alcalde, F., Zampaloni, C., Sauer, K. Comparative evaluation of rRNA depletion procedures for the improved analysis of bacterialbiofilm and mixed pathogen culture transcriptomes. Scientific Reports. 7, 41114 (2017).
- Oh, E. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Chew, G. L. Ribosome profiling reveals resemblance between long non-coding RNAs and 5′ leaders of coding RNAs. Development. 140 (13), 2828-2834 (2013).
- Dunn, J. G., Foo, C. K., Belletier, N. G., Gavis, E. R., Weissman, J. S. Ribosome profiling reveals pervasive and regulated stop codon readthrough in Drosophila melanogaster. eLife. 2, 01179 (2013).
- Liu, M. J., et al. Translational landscape of photomorphogenic Arabidopsis. The Plant cell. 25 (10), 3699-3710 (2013).
- Rooijers, K., Loayza-Puch, F., Nijtmans, L. G., Agami, R. Ribosome profiling reveals features of normal and disease-associated mitochondrial translation. Nature Communications. 4, 2886 (2013).
- Danielle, L., et al. Fidelity of translation initiation is required for coordinated respiratory complex assembly. Science Advances. 5 (12), 2118 (2019).
- Zoschke, R., Watkins, K. P., Barkan, A. A rapid ribosome profiling method elucidates chloroplast ribosome behavior in vivo. The Plant cell. 25 (6), 2265-2275 (2013).
- Chotewutmontri, P., Barkan, A. Dynamics of chloroplast translation during chloroplast differentiation in maize. PLoS genetics. 12 (7), 1006106 (2016).
- Lee, S., Liu, B., Huang, S. X., Shen, B., Qian, S. B. Global mapping of translation initiation sites in mammalian cells at single-nucleotide resolution. Proceedings of the National Academy of Sciences of the United States of America. 109 (37), 2424-2432 (2012).
- Martinez, T. F., Chu, Q., Donaldson, C., Tan, D., Shokhirev, M. N., Saghatelian, A. Accurate annotation of human protein-coding small open reading frames. Nature chemical biology. 16 (4), 458-468 (2020).
- Simsek, D., et al. The Mammalian Ribo-interactome Reveals Ribosome Functional Diversity and Heterogeneity. Cell. 169 (6), 1051-1065 (2017).
- Fritsch, C., et al. Genome-wide search for novel human uORFs and N-terminal protein extensions using ribosomal footprinting. Genome research. 22 (11), 2208-2218 (2012).
- Lareau, L. F., Hite, D. H., Hogan, G. J., Brown, P. O. Distinct stages of the translation elongation cycle revealed by sequencing ribosome-protected mRNA fragments. eLife. 3, 01257 (2014).
- Meydan, S., et al. Retapamulin-Assisted Ribosome Profiling Reveals the Alternative Bacterial Proteome. Molecular Cell. 74 (3), 481-493 (2019).
- Wilson, D. N. The A-Z of bacterial translation inhibitors. Critical Reviews in Biochemistry and Molecular Biology. 44, 393-433 (2009).
- Nakahigashi, K. Comprehensive identification of translation start sites by tetracycline-inhibited ribosome profiling. DNA research : an international journal for rapid publication of reports on genes and genomes. 23 (3), 193-201 (2016).
- Weaver, J., Mohammad, F., Buskirk, A. R., Storz, G. Identifying Small Proteins by Ribosome Profiling with Stalled Initiation Complexes. mBio. 10 (2), 02819 (2019).
- Nakahigashi, K. Effect of codon adaptation on codon-level and gene-level translation efficiency in vivo. BMC genomics. 15 (1), 1115 (2014).
- Li, G. W., Burkhardt, D., Gross, C., Weissman, J. S. Quantifying absolute protein synthesis rates reveals principles underlying allocation of cellular resources. Cell. 157 (3), 624-635 (2014).
- Michel, A. M., et al. Observation of dually decoded regions of the human genome using ribosome profiling data. Genome research. 22 (11), 2219-2229 (2012).
- Guydosh, N. R., Green, R. Dom34 rescues ribosomes in 3′ untranslated regions. Cell. 156 (5), 950-962 (2014).
- Young, D. J., Guydosh, N. R., Zhang, F., Hinnebusch, A. G., Green, R. Rli1/ABCE1 Recycles Terminating Ribosomes and Controls Translation Reinitiation in 3’UTRs In Vivo. Cell. 162 (4), 872-884 (2015).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Dobin, A., et al. ultrafast universal RNA seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- . . Babraham Bioinformatics. Fast QC. , (2021).
- Gerashchenko, M. V., Gladyshev, V. N. Ribonuclease selection for ribosome profiling. Nucleic acids research. 45 (2), 6 (2017).
- Verbruggen, S., Menschaert, G. mQC: A post-mapping data exploration tool for ribosome profiling. Computer Methods and Programs in Biomedicine. 181, 104806 (2019).
- Cui, H., Hu, H., Zeng, J., Chen, T. DeepShape: estimating isoform-level ribosome abundance and distribution with Ribo-seq data. BMC bioinformatics. 20, 678 (2019).
- Choi, J. . RiboToolkit: an integrated platform for analysis and annotation of ribosome profiling data to decode RNA translation at codon resolution. , (2021).
- Choi, J. Dynamics of the context-specific translation arrest by chloramphenicol and linezolid. Nature chemical biology. 16 (3), 310-317 (2020).
- Tompson, J., O’Connor, M., Mills, J. A., Dahlberg, A. E. The protein synthesis inhibitors, oxazolidinones and chloramphenicol, cause extensive translational inaccuracy in vivo. Journal of Molecular Biology. 322 (2), 273-279 (2002).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature reviews. Molecular cell biology. 16 (11), 651-664 (2015).
- Glaub, A., Huptas, C., Neuhaus, K., Ardern, Z. Recommendations for bacterial ribosome profiling experiments based on bioinformatic evaluation of published data. Journal of Biological Chemistry. 295, 8999-9011 (2020).
- Sorour, M. H., Hani, H. A., Shaalan, H. F., El-Sayed, M. M. H. Experimental screening of some chelating agents for calcium and magnesium removal from saline solutions. Desalination and Water Treatment. 57 (48-49), 22799-22808 (2015).
- Mohammad, F., Green, R., Buskirk, A. R. A systematically-revised ribosome profiling method for bacteria reveals pauses at single-codon resolution. Elife. 8, 42591 (2019).
- O’Connor, P. B., Li, G. W., Weissman, J. S., Atkins, J. F., Baranov, P. V. rRNA:mRNA pairing alters the length and the symmetry of mRNA-protected fragments in ribosome profiling experiments. Bioinformatics. 29 (12), 1488-1491 (2013).
- Protocol for RNA Clean & Concentrator -25. Zymo Research Available from: https://files.zymoresearch.com/protocols/_r1017_r1018_rna_clean_concentrator-25.pdf (2021)
- Protocol for use with NEBNext Small RNA Library Prep Set for Illumina (E7300, E7580, E7560, E7330). New England Biolabs Available from: https://www.international.neb.com/protocols/2018/03/27/protocol-for-use-with-nebnext-small-rna-library-prep-set-for-illumina-e7300-e7580-e7560-e7330 (2021)

